Arginin-Deiminase
Arginin-Deiminase (ADI) ist ein Enzym, das hauptsächlich in Bakterien und Eukaryoten vorkommt.[1] Die Arginin-Deiminase katalysiert den ersten Schritt des Arginin-Deaminase-Stoffwechselweges (auch als Arginindihydrolase-Stoffwechselweg bekannt), der für die Produktion von Adenosintriphosphat (ATP) und des Säure-Basen-Haushalts von Bakterien notwendig ist und sich als Überlebensstrategie für bestimmte Bakterien entwickelt hat.[2]
| Arginin-Deiminase | ||
|---|---|---|
| ||
| Bändermodell der Arginin-Deiminase von P. aeruginosa, nach PDB 1RXX | ||
| Andere Namen |
| |
|
Vorhandene Strukturdaten: 4BOF, 2CMU, 2A9G, 2AAF, 2ABR, 2ACI, 1S9R, 1LXY | ||
| Bezeichner | ||
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 3.5.3.6, Hydrolase | |
| Reaktionsart | Hydrolyse | |
| Substrat | L-Arginin + H2O | |
| Produkte | L-(+)-Citrullin + NH3 | |
| Vorkommen | ||
| Übergeordnetes Taxon | Bakterien, Eukaryoten | |
Arginin-Deiminase-Stoffwechselweg
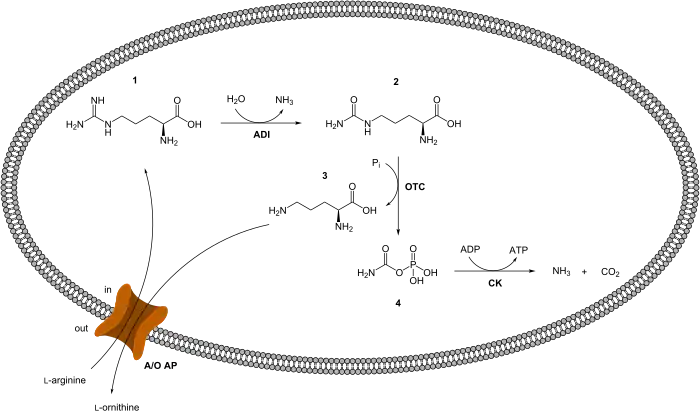
Der Arginin-Deiminase-Stoffwechselweg ist ein katabolischer Stoffwechselweg, der in verschiedenen Bakterien untersucht wurde, beispielsweise in Milchsäurebakterien, Mykoplasmen, Halobakterien, Pseudomonas spp. und Bacillus spp. Dabei wird L-Arginin (1) über den Arginin/Ornithin-Antiporter (A/O AP) in den Stoffwechselweg eingeführt. L-Arginin wird zunächst durch das Enzym Arginin-Deiminase zu L-(+)-Citrullin (2) und Ammoniak hydrolysiert. Das L-(+)-Citrullin wird danach durch Abspaltung des Aminocarbonylrestes mittels des Enzyms Ornithin-Transcarbamylase (OTC) zu L-(+)-Ornithin (3) und anschließender Phosphorylierung des Aminocarbonylrestes zu Carbamoylphosphat (4) umgesetzt. Anschließend wird Carbamoylphosphat durch das Enzym Carbamatkinase (CK) unter ATP-Bildung zu Ammoniak und Kohlenstoffdioxid umgesetzt. Das gebildete L-(+)-Ornithin verlässt den Stoffwechselweg über den gleichen Antiporter.
Dieser Stoffwechselweg wird insbesondere bei Sauerstoffmangel verwendet oder, in geringerem Maße, wenn sämtliche Kohlenstoff- oder andere Energiequellen aufgebraucht sind. Kontrolliert wird der Stoffwechselweg durch Katabolitrepression. Dabei werden, wenn energetisch günstigere Produkte in signifikanter Menge vorhanden sind, die Gene katabolischer Enzyme so reguliert, dass die Produktion dieser Enzyme gehemmt wird. Des Weiteren beeinflusst der Stoffwechselweg den Säure-Basen-Haushalt verschiedener Bakterien. Bei Säurestress wird die Glykolyse und das Wachstum der Bakterien gehemmt. Bakterien können sich vom Säurestress durch den im Stoffwechselweg produzierten Ammoniak und den daraus resultierenden pH-Anstieg erholen. Dies ermöglicht ein Überleben der Bakterien und der Anreicherung von weiterer Säure, ohne dass es zum Säurestress kommt.[2]
Nachweis des bakteriellen Enzyms
Da der Arginin-Deiminase-Stoffwechselweg in zahlreichen Bakterienarten vorkommt, wird er zu ihrer Differenzierung und Bestimmung verwendet. In der mikrobiologischen Fachliteratur wird der Nachweis der beiden zuerst genannten Enzyme als Arginindihydrolase (ADH) zusammengefasst[3][4] und beinhaltet die Umwandlung von L-Arginin (1) über L-Citrullin (2) zu L-Ornithin (3), unter Abspaltung von Ammoniak (vergleiche Stoffwechselschema). Der Nachweis der Arginindihydrolase in Vertretern der gramnegativen Enterobakterien dient zur Charakterisierung und ist Bestandteil einer Bunten Reihe zur Bestimmung der Gattung oder Art.[5] Das Testverfahren wurde 1955 eingeführt[6] und ist seit den 1970er Jahren Bestandteil von miniaturisierten Testsystemen (z. B. im API 20 E-System).[7]
Für den Nachweis der bakteriellen Arginindihydrolase wird das standardisierte, argininhaltige Nährmedium mit Bakterienmaterial beimpft und inkubiert.[3] Die Inkubation soll unter anoxischen Bedingungen erfolgen, um den Zutritt von Sauerstoff in das Teströhrchen zu verhindern, wird der inokulierte Ansatz mit Paraffinöl überschichtet.[8] Durch die Bildung von Ornithin und Ammoniak steigt der pH-Wert im Testmedium, die Auswertung erfolgt anhand des Farbumschlags des im Nährmediums integrierten pH-Indikators. Meist wird Phenolrot eingesetzt, es soll eine Inkubationsdauer von 18 bis 24 Stunden eingehalten werden, bevor man die ADH-Reaktion beurteilt.[3][7] Als schnellere Variante ist eine Methode unter Verwendung der Dünnschichtchromatographie beschrieben: Das flüssige, argininhaltige Nährmedium (pH-Wert 5,5) wird nach Inokulation eine Stunde bebrütet, zentrifugiert und der Überstand mit Dansylchlorid versetzt und eine weitere Stunde inkubiert. Nach erneuter Zentrifugation kann der Überstand mit Hilfe der zweidimensionalen Dünnschichtchromatographie untersucht werden, die mit Dansylchlorid umgesetzten Reaktionsprodukte der Arginindihydrolase werden durch ihre Fluoreszenz detektiert.[4]
Der Nachweis der bakteriellen Arginindihydrolase ist für die Unterscheidung der Enterobakterien von Bedeutung. Vertreter der Gattungen Enterobacter, Cronobacter und Cedecea verfügen über dieses Enzym, ebenso Serovare von Salmonella enterica subsp. arizonae und die Spezies Plesiomonas shigelloides. Hingegen sind Vertreter der Gattungen Escherichia, Klebsiella (inkl. Klebsiella aerogenes), Morganella, Pantoea, Proteus, Providencia, Raoultella, Serratia, Shigella und Yersinia ADH-negativ. Innerhalb der Gattungen Salmonella wie auch Citrobacter gibt es ADH-positive und -negative Vertreter, zu deren Unterscheidung der Nachweis der Arginindihydrolase-Reaktion beiträgt.[5]
Beispiele für weitere Gammaproteobacteria, die von medizinischer Bedeutung sind und über die Arginindihydrolase verfügen, sind einige Arten aus der Familie Vibrionaceae, beispielsweise Vibrio fluvialis und Photobacterium damselae. Auch die Gattungen Aeromonas und Pseudomonas (Gammaproteobacteria) sind ADH-positiv, während die ADH-positive Spezies Chromobacterium violaceum zu den Betaproteobacteria gehört. Die genannten Bakterien sind für die medizinische Mikrobiologie relevant, werden zum Teil in der Gruppe „nicht-fermentierende gramnegative Stäbchen“ geführt und können dann beispielsweise mit dem API 20 NE-System identifiziert werden.[4][9]
Einzelnachweise
- UniProtKB results. In: UniProtKB. Abgerufen am 31. Dezember 2019.
- Jon J. Kabara: Preservative-Free and Self-Preserving Cosmetics and Drugs: Principles and Practices. CRC Press, 1997, ISBN 0-8247-9366-8, S. 27–28 (englisch, eingeschränkte Vorschau in der Google-Buchsuche).
- Roland Süßmuth, Jürgen Eberspächer, Rainer Haag, Wolfgang Springer: Biochemisch-mikrobiologisches Praktikum. 1. Auflage. Thieme Verlag, Stuttgart/New York 1987, ISBN 3-13-685901-4, S. 78–85.
- K. C. Chen, N. J. Culbertson, J. S. Knapp, G. E. Kenny, K. K. Holmes: Rapid method for simultaneous detection of the arginine dihydrolase system and amino acid decarboxylases in microorganisms. In: Journal of Clinical Microbiology. Band 16, Nr. 5, November 1982, S. 909–919, PMID 7153341, PMC 272502 (freier Volltext).
- J. J. Farmer III, B. R. Davis u. a.: Biochemical identification of new species and biogroups of Enterobacteriaceae isolated from clinical specimens. In: Journal of Clinical Microbiology. Band 21, Nr. 1, Januar 1985, S. 46–76, PMID 3881471, PMC 271578 (freier Volltext).
- Vagn Møller: Simplified tests for some amino acid decarboxylases and for the arginine dihydrolase system. In: Acta pathologica et microbiologica Scandinavica. Band 36, Nr. 2, 1955, S. 158–172, doi:10.1111/j.1699-0463.1955.tb04583.x, PMID 14375937.
- P. B. Smith, K. M. Tomfohrde, D. L. Rhoden, A. Balows: API system: a multitube micromethod for identification of Enterobacteriaceae. In: Applied Microbiology. Band 24, Nr. 3, September 1972, S. 449–452, PMID 4562482, PMC 376540 (freier Volltext).
- System zur Identifizierung von Enterobacteriaceae und anderen gramnegativen, nicht anspruchsvollen Stäbchen. In: bioMérieux sa (Hrsg.): Bedienungsanleitung api® 20 E API, REF 20 100 / 20 160. Mai 2004, S. 1–5.
- Species identifiable by the various identification systems. In: bioMérieux sa (Hrsg.): API & ID 32 Identification Databases. April 2015 (biomerieux.de).