Pyruvatdehydrogenase-Komplex
Der Pyruvatdehydrogenase-Komplex (PDC) ist ein sehr großer Multienzymkomplex, der die irreversible oxidative Decarboxylierung von Pyruvat katalysiert. Er ist für die Energiegewinnung aus Kohlenhydraten, beispielsweise D-Glucose, essenziell nötig, da er die Glykolyse mit dem Citratzyklus verbindet.
| Übergeordnet |
| Zytosol |
| Gene Ontology |
|---|
| QuickGO |
Ein funktionierender Pyruvatdehydrogenase-Komplex wurde in jedem aeroben Eukaryot sowie aeroben Prokaryot gefunden.[1] Bei fakultativ anaeroben Bakterien ist der Komplex unter anaeroben Bedingungen inaktiv. Obligat anaerobe Bakterien wie Clostridien oder aerobe Archaeen verwenden dagegen eine Pyruvat-Ferredoxin-Oxidoreduktase.
Aufbau
Der Komplex findet sich bei Eukaryoten in der mitochondrialen Matrix, bei Prokaryoten im Cytoplasma[2] und bei Pflanzen zusätzlich in Plastiden.[3] Er besteht aus multiplen Kopien dreier Enzym-Untereinheiten, die jede für sich eine Teilreaktion katalysieren. Die aktiven Zentren der jeweiligen Untereinheiten sind dabei eng benachbart:[4]
Der gesamte Komplex zählt in Eukaryoten zu einem der größten, bekannten Multienzymkomplexe. Er hat einen Durchmesser von ca. 500 Å und eine molare Masse von 9,5 Megadalton.[4] Die Kernstruktur, ein Dodekaeder, wird von 60 E2-Untereinheiten gebildet, die an den 20 Ecken des Dodekaeders Trimere bilden. An diese Kernstruktur sind 30 E1 Heterotetramere und 12 E3-Homodimere lokalisiert. Darüber hinaus findet sich noch ein E3-Bindeprotein, (E3BP), welches die E3-Untereinheiten an den Gesamtkomplex bindet.[5]
Bakterien haben eine etwas andere Zusammensetzung des Komplexes. Am besten ist der aus Escherichia coli untersucht, was auf die Arbeiten von Lester Reed zurückgeht.[6] Dort besteht der PDC aus 24 E1-Untereinheiten, 24 E2-Untereinheiten und 12 E3-Untereinheiten. Er hat eine Masse von ca. 4,6 MDa und einen ca. 300 Å großen Durchmesser. Dessen E2-Untereinheiten bilden hierbei einen Würfel, an dessen Ecken jeweils Trimere lokalisiert sind.[7] Es gibt aber auch ein paar Gram-negative Bakterien, deren Kernstruktur wie die bei Eukaryoten aufgebaut ist.
An den enzymatischen Reaktionen sind zahlreiche Cofaktoren beteiligt: TPP, proteingebundenes Liponamid und FAD sind katalytische Cofaktoren, während Coenzym A sowie NAD+ als stöchiometrische Cofaktoren fungieren.[8]
Die Reaktion
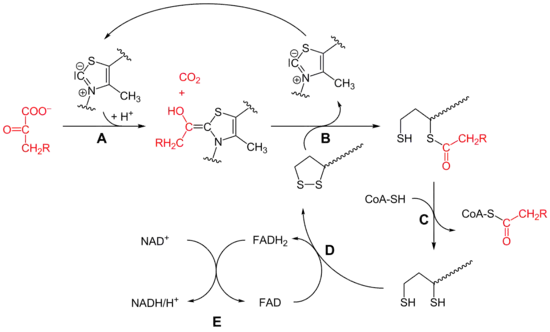
Bei der oxidativen Decarboxylierung wird vom Pyruvat (C3) Kohlenstoffdioxid (CO2) abgespalten und ein NADH gewonnen. Dabei wird eine energiereiche Thioesterbindung zwischen Coenzym A und dem Acetatrest gebildet, so dass Acetyl-CoA entsteht. Die Energie hierfür stammt aus der Decarboxylierung. Die Umwandlung von Pyruvat zu Acetyl-CoA ist unter physiologischen Bedingungen irreversibel.
Teilschritte
- Die Decarboxylierung von Pyruvat erfolgt mit Hilfe der Pyruvatdehydrogenase (E1) des Pyruvatdehydrogenase-Komplex (A). Bei dieser katalysierten Reaktion ist Thiaminpyrophosphat (TPP) die prosthetische Gruppe und bildet eine Atombindung mit Pyruvat. Das Reaktionsprodukt ist Hydroxyethyl-TPP und CO2. Diese Hydroxyethylgruppe wird zu einer Acetylgruppe oxidiert und von Liponamid übernommen, so dass eine energiereiche Thioesterbindung, S-Acetylliponamid (B), entsteht. Liponamid ist an der Transacetylase-Untereinheit kovalent gebunden. Die Disulfidgruppe des Liponamids wird bei dieser Reaktion zur Disulfhydrylform reduziert.
- Der Acetylrest von Acetylliponamid wird auf Coenzym A übertragen, somit entstehen Acetyl-CoA und Dihydroliponamid (C). Dies wird von der Dihydrolipoyl-Transacetylase (E2) katalysiert. Formal erfolgt bei dieser Reaktion eine Umesterung, wodurch die energiereiche Thioesterbindung erhalten bleibt.[9]
- Dihydroliponamid wird durch die Dihydrolipoyl-Dehydrogenase (E3)-Untereinheit zu Liponamid regeneriert. Dabei wird ein kovalent gebundenes FAD zu FADH2 reduziert (D), welches durch die Reduktion von NAD+ wieder regeneriert wird (E). Die Übertragung von Elektronen findet normalerweise in umgekehrter Richtung von NADH zu FAD statt. Das Elektronenübertragungspotential FADs ist durch seine kovalente Bindung mit dem Protein aber ausreichend erhöht, so dass die Reaktion ablaufen kann.[9]
Somit ergibt sich folgende Gesamtreaktion:
Durch die Generierung von Acetyl-CoA aus Pyruvat wird eine Verbindung zwischen der Glykolyse und Citratzyklus hergestellt. Das entstandene Acetyl-CoA kann dann mit Oxalacetat durch die Citratsynthase weiter zu Citrat umgesetzt werden. Das NADH/H+ kann durch die Atmungskette wieder reoxidiert werden.
Essentialität von Vitamin B1 und Mangel
Der Pyruvatdehydrogenase-Komplex ist, gemäß der beschriebenen Reaktion, für alle (netto-) Energiegewinnung aus Kohlenhydraten (im Gegensatz zu Fetten) notwendig. Mit dem Anteil von Vitamin B1 (Thiamin) ist hierzu auch ein Vitamin nötig, also ein Stoff der von außen zugeführt werden muss. Es gibt einen erhöhten Bedarf für Thiamin bei stark erhöhter Kohlenhydratzufuhr. Bei normaler gesunder Ernährung ohne Alkoholkonsum ist eine zusätzliche Thiaminzufuhr nicht notwendig.[10][11][12][13][14]
Regulation
Die Endprodukte Acetyl-CoA und auch NADH können zu einer Hemmung des Pyruvatdehydrogenase-Komplexes führen (Produkthemmung). Darüber hinaus wird der Komplex auch durch zwei Modifikationen reguliert. Hierbei katalysieren eine Pyruvatdehydrogenase-Kinase (PDK) und eine Phosphopyruvatdehydrogenase-Phosphatase (PDP) die reversible Phosphorylierung des cytosolischen PDC.[15] In Säugern werden drei, in Pflanzen zwei hochkonservierte Serinreste der E1-Untereinheit durch die PDK unter ATP-Verbrauch phosphoryliert. Dies bewirkt eine komplette Inaktivierung der PDC. Die Phosphatase macht die Phosphorylierung wieder rückgängig und aktiviert damit den Gesamtkomplex.
Beim Menschen wird die PDP durch Calcium- sowie Magnesiumionen stimuliert.[16] Eine Steigerung des Calciumspiegels kann auch von α-Sympathomimetika und Vasopressin hervorgerufen werden. Die PDK wird dagegen von Acetyl-CoA und NADH stimuliert, während Pyruvat, ADP und Calciumionen einen hemmenden Effekt haben. In Pflanzen ist die Aktivität der Kinase höher als die der Phosphatase, so dass sie dort noch zusätzlich reguliert werden muss. Hierbei aktiviert Ammonium (NH4+) die PDK, während Pyruvat und ADP diese hemmen.
Hemmstoffe
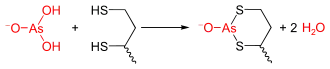
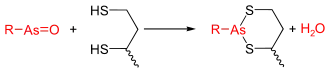
Arsen(III)-verbindungen wie Arsenit (AsO33−) oder organische Arsenverbindungen gehen kovalente Verbindungen mit Sulfhydrylgruppen ein. Daher vermögen sie das Liponamid aus der PDC zu inaktivieren und wirken damit toxisch.
PDC in Plastiden
In Pflanzen kommt der Pyruvatdehydrogenase-Komplex nicht nur in Mitochondrien vor, sondern auch in Plastiden. Dort ist er in der Bereitstellung von Acetyl-CoA für die Fettsäuresynthese involviert.[17] Jedoch ist die Aktivität des Komplexes – je nach Entwicklungsstadium der Zelle – eher gering. Der größte Teil des Acetyl-CoA wird nämlich aus Acetat bezogen, was ATP-abhängig von einer Acetyl-CoA-Synthetase katalysiert wird.
Siehe auch
Literatur
- Zhou, ZH. et al. (2001): The remarkable structural and functional organization of the eukaryotic pyruvate dehydrogenase complexes. In: Proc Natl Acad Sci USA 98(26); 14802–14807; PMID 11752427; PDF (freier Volltextzugriff, engl.)
Weblinks
- Citratzyklus mit Stellung von Pyruvat-Dehydrogenase und Moleküldarstellungen in Farbe (engl.). Archiviert vom Original am 21. Februar 2014; abgerufen am 17. Januar 2016.
- Oxidative decarboxylation of pyruvate to acetyl CoA by pyruvate dehydrogenase
- OrphaNet: Pyruvat-Dehydrogenase-Mangel
Einzelnachweise
- Garabed Antranikian: Angewandte Mikrobiologie. Springer, Berlin 2006; ISBN 978-3-540-24083-9; S. 52f.
- Katharina Munk (Hrsg.): Taschenlehrbuch Biologie: Mikrobiologie. Thieme Verlag Stuttgart 2008, ISBN 978-3-13-144861-3, S. 355.
- Caroline Bowsher, Martin W. Steer, Alyson K. Tobin: Plant Biochemistry. Garland Pub, New York, NY 2008, ISBN 978-0-8153-4121-5. S. 157
- Reginald Garrett und Charles M. Grisham: Biochemistry. (International Student Edition). Cengage Learning Services; 4. Auflage 2009; ISBN 978-0-495-11464-2; S. 566–571
- Caroline Bowsher, Martin W. Steer, Alyson K. Tobin: Plant Biochemistry. Garland Pub, New York, NY 2008, ISBN 978-0-8153-4121-5. S. 160
- Donald Voet, Judith G. Voet, Alfred Maelicke (Hrsg.), Werner Müller-Esterl (Hrsg.): Biochemie. Wiley-VCH 1992. ISBN 3-527-28242-4; S. 185
- Zhou, ZH. et al. (2001): The remarkable structural and functional organization of the eukaryotic pyruvate dehydrogenase complexes. In: Proc Natl Acad Sci USA 98(26); 14802–14807; PMID 11752427; PDF (freier Volltextzugriff, engl.)
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie. 6 Auflage. Spektrum Akademischer Verlag, Heidelberg 2007; ISBN 978-3-8274-1800-5; S. 534
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer: Biochemie. 6 Auflage. Spektrum Akademischer Verlag, Heidelberg 2007; ISBN 978-3-8274-1800-5; S. 536
- Sauberlich et al.: Thiamin requirement of the adult human. Am J Clin Nutr. 1979 Nov;32(11):2237-48. PMID 495541
- Buchseite zu Thiamin-Verbrauch aus „Ernährungsmedizin“, Thieme Verlag
- Tasevska N, Runswick SA, McTaggart A, Bingham SA: Twenty-four-hour urinary thiamine as a biomarker for the assessment of thiamine intake. In: Eur J Clin Nutr. 62, Nr. 9, September 2008, S. 1139–47. doi:10.1038/sj.ejcn.1602829. PMID 17565356.. PMID 17565356.
- Iber FL, Blass JP, Brin M, Leevy CM: Thiamin in the elderly--relation to alcoholism and to neurological degenerative disease. In: Am. J. Clin. Nutr.. 36, Nr. 5 Suppl, November 1982, S. 1067–82. PMID 6765072.
- Webster MJ, Scheett TP, Doyle MR, Branz M: The effect of a thiamin derivative on exercise performance. In: Eur J Appl Physiol Occup Physiol. 75, Nr. 6, 1997, S. 520–4. PMID 9202948.
- Caroline Bowsher, Martin W. Steer, Alyson K. Tobin: Plant Biochemistry. Garland Pub, New York, NY 2008, ISBN 978-0-8153-4121-5. S. 162ff.
- Melanie Königshoff und Timo Brandenburger: Kurzlehrbuch Biochemie. Nach dem neuen GK 1. Thieme, Stuttgart 2004; ISBN 3-13-136411-4; S. 124
- Hans W. Heldt, Birgit Piechulla: Pflanzenbiochemie. 4. Auflage. Spektrum Akademischer Verlag, Heidelberg 2008; ISBN 978-3-8274-1961-3; S. 354