Diphtherietoxin
Das Diphtherietoxin (DT) ist ein Exotoxin aus Corynebacterium diphtheriae, dem Erreger der Diphtherie und hemmt die Proteinbiosynthese bei Eukaryoten und Archaeen durch Blockierung der Translation während der Elongationsphase. Das hochmolekulare und hitzelabile Toxin[1] zählt zu den Lektinen. Die genetische Information ist im Prophagen β („Corynebacterium-Phage beta“) enthalten. Nur wenn das Bakterium von ihm infiziert wird, kann es selbst das Toxin bilden. Demnach können via Phagenkonversion (Infektion mit tox+-Phagen) nichttoxigene C. diphtheriae-Stämme die Fähigkeit, DT zu erzeugen, erwerben.[2][3][4]
| Diphtherietoxin (Corynephage β) | ||
|---|---|---|
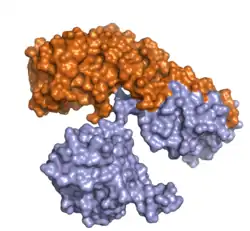
| ||
| Oberflächenmodell nach PDB 1DDT. Kette A (Enzym) orange, Kette B (Transporter) hellblau. | ||
| Masse/Länge Primärstruktur | 535 = 193+342 Aminosäuren | |
| Sekundär- bis Quartärstruktur | A+B | |
| Bezeichner | ||
| Gen-Name(n) | DT | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 2.4.2.36, Glycosyltransferase | |
| Reaktionsart | Übertragung eines ADP-Riboserests | |
| Substrat | NAD+ + EF-2 | |
| Produkte | NAD + EF-2(defekt) | |
Wirkungsweise
Das Toxin besteht aus zwei Proteinen Toxin A und Toxin B, die über eine Disulfidbrücke (Cys186-Cys201)[5] miteinander verbunden sind und bindet selektiv an die 80 S-Ribosomen von eukaryotischen Zellen. Seine Masse beträgt etwa 61 kDa. Es sind drei Funktionsbereiche (Domänen) auf dem Toxin bekannt:[6]
- R-Domäne: es handelt sich um eine Rezeptorbindungsstelle und ermöglicht das Binden an ein Rezeptorprotein der Zielzelle
- T-Domäne: diese Domäne vermittelt die Translokation des Enzymanteils des Toxins in die Zielzelle
- C-Domäne: enzymatischer Teil, der die ADP-Ribosylierung (s. u.) katalysiert
Auf der N-terminalen A-Kette befindet sich die C-Domäne, auf der B-Kette die R- und T-Domäne.[5]
Das Eindringen in die Zelle unterliegt dem gleichen Mechanismus wie bei Ricin: Die B-Kette heftet sich an einen Rezeptor auf der Zelloberfläche, dadurch spaltet sich das Toxin in ein 21 kDa A-Fragment und ein 40 kDa B-Fragment, wobei das A-Fragment in die Zelle eindringt. Diesen Mechanismus hat das Diphtherietoxin mit etlichen anderen Bakterientoxinen gemeinsam, es gehört zur Gruppe der AB-Toxine.
Das Ziel der enzymatischen Aktivität des A-Fragmentes ist der Elongationsfaktor eEF-2, der die Translation bei der Proteinsynthese von Eukaryoten katalysiert. eEF-2 enthält Diphthamid, einen ungewöhnlichen Aminosäurerest an Position 715 mit unbekannter Funktion, der posttranslational aus Histidin gebildet wird.[5] Das A-Fragment des Toxins katalysiert die kovalente Modifikation dieses Diphthamids. Dabei wird ein ADP-Ribosylrest aus NAD+ unter Abspaltung von Nicotinamid auf ein Stickstoffatom im Diphthamidring übertragen.[7] Man spricht von einer ADP-Ribosylierung.[6]
Das Diphthamid kommt nur im eEF-2 vor, was die hohe Spezifität erklärt. Dadurch hemmt das Diphtherietoxin die Fähigkeit des eEF-2, die Translation der wachsenden Polypeptidkette auszuführen. Die Proteinsynthese stoppt, und dies ist die Ursache für die bemerkenswerte Toxizität des Diphtherietoxins. Bereits ein A-Kettenmolekül reicht aus, um eine Zelle abzutöten.[6] Aufgrund der hohen Oberflächenrezeptorendichte bei Herz- und Nervenzellen sind diese Zellen am empfindlichsten.
Das Diphtherietoxin ist der wirksame Bestandteil in Diphtherieimpfstoffen.
Die Kristallstruktur des Toxins wurde 1992 in einer Auflösung von 2,5 Å bestimmt.[8]
CRM197
Für Konjugatimpfstoffe nutzt man die ungiftige Variante des Toxins mit der Bezeichnung CRM197 (cross-reacting material 197).[5] Hierbei ist die Aminosäure Glycin an Position 52 durch Glutaminsäure ersetzt, wodurch die ADP-Ribosyltransferaseaktivität verloren geht.[9] Die Mutante CRM197 wurde erstmals 1973 isoliert, bis Mitte der 1980er Jahre weiter optimiert und in einen Bakterienstamm zur Produktion kloniert.
Ähnliche Toxine
Auch andere Gifte, die von Krankheitserregern stammen, bestehen aus zwei miteinander verbundenen Peptidketten, deren eine an einen Rezeptor auf der Zelloberfläche bindet und so der anderen Kette Zutritt in das Zellinnere verschafft wie zum Beispiel das Choleratoxin, das Pertussistoxin (Keuchhustentoxin) und das Milzbrandtoxin. Allerdings sind die toxischen Mechanismen dieser Toxine unterschiedlich. Den identischen Mechanismus der NAD-abhängigen ADP-Ribosylierung nutzt auch das Exotoxin A aus Pseudomonas aeruginosa.
Literatur
- Stryer Lubert: Biochemie. Verlag Spektrum der Wissenschaft, Heidelberg 1990, ISBN 3-89330-690-0, S. 791–796.
Einzelnachweise
- Marlies Höck und Helmut Hahn: Korynebakterien. In: Sebastian Suerbaum, Gerd-Dieter Burchard, Stefan H. E. Kaufmann, Thomas F. Schulz (Hrsg.): Medizinische Mikrobiologie und Infektiologie. Springer-Verlag, 2016, ISBN 978-3-662-48678-8, S. 309.
- Diphtherie - RKI-Ratgeber. In: RKI. 10. Januar 2018, abgerufen am 2. Februar 2021.
- SIB: Viral exotoxin. Expasy: ViralZone
- SIB: Modulation of host virulence by virus. Expasy: ViralZone
- Michael Bröker et al.: Biochemical and biological characteristics of cross-reacting material 197 CRM197, a non-toxic mutant of diphtheria toxin: use as a conjugation protein in vaccines and other potential clinical applications. In: Biologicals: Journal of the International Association of Biological Standardization. Band 39, Nr. 4, Juli 2011, S. 195–204, doi:10.1016/j.biologicals.2011.05.004, PMID 21715186.
- Marlies Höck und Helmut Hahn: Korynebakterien. In: Sebastian Suerbaum, Gerd-Dieter Burchard, Stefan H. E. Kaufmann, Thomas F. Schulz (Hrsg.): Medizinische Mikrobiologie und Infektiologie. Springer-Verlag, 2016, ISBN 978-3-662-48678-8, S. 311 f.
- Jeremy M. Berg et al.: Stryer Biochemie. 8. Auflage. Springer-Verlag, 2018, ISBN 978-3-662-54620-8, S. 1085.
- Seunghyon Choe et al.: The crystal structure of diphtheria toxin. In: Nature. Band 357, Nr. 6375, Mai 1992, S. 216–222, doi:10.1038/357216a0.
- Enrico Malito et al.: Structural basis for lack of toxicity of the diphtheria toxin mutant CRM197. In: Proceedings of the National Academy of Sciences of the United States of America. Band 109, Nr. 14, 3. April 2012, S. 5229–5234, doi:10.1073/pnas.1201964109, PMID 22431623, PMC 3325714 (freier Volltext).