Elongation (Translation)
Die Elongation während der Translation behandelt den Prozess der Verlängerung der Aminosäurenkette während der Biosynthese der Proteine; sie findet am Erkennungs- und am Bindungsort des Ribosoms statt. Ein einzelner Elongationsschritt enthält drei Schritte: Bindung der beladenen tRNA, Ausbildung der Peptidbindung und Vorbereitung auf den nächsten Elongationsschritt. Dies wiederholt sich so lange, bis ein terminierendes Codon erreicht ist.
| Übergeordnet |
| Translation |
| Gene Ontology |
|---|
| QuickGO |
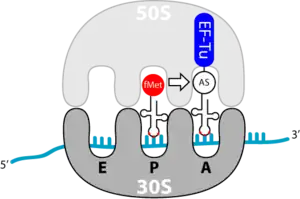
Bindung der Aminoacyl-tRNA in der A-Position
Die Elongation beginnt mit der Bindung einer beladenen tRNA in der A-Stelle. Dieser Vorgang beruht auf dem Transport durch den Elongationsfaktor Tu (EF-Tu). Bei diesem handelt es sich wieder um ein G-Protein. EF-Tu hat zwei wichtige Aufgaben:
- Schutz der Esterbindung zwischen 3'-OH der tRNA und der transportierten Aminosäure
- sich selbst freizusetzen, wenn die korrekte tRNA an die Codon-Sequenz der A-Stelle des Ribosoms gebunden hat.
Die Entlassung beruht auf der Hydrolyse des gebundenen GTP. Diese kann nur stattfinden bei einer korrekten Paarung zwischen Ribosom und tRNA, also wenn sich die richtige tRNA im Ribosom befindet. Eine falsche tRNA kann nicht vom EF-Tu dissoziieren, wodurch der Einbau der richtigen Aminosäure garantiert wird.
Interessant ist weiterhin zu bemerken, dass EF-Tu jede tRNA erkennen kann, außer der Initiator-tRNA, fmet-tRNA. Somit kann gewährleistet werden, dass bei internen AUG-Codons, nicht fMet, sondern Methionin eingebaut wird.
Die Ausbildung der Peptidbindung
Dies ist ein spontaner Schritt, aufgrund der räumlichen Gegebenheiten der Aminosäuren zueinander und der größeren Bindungsstärke der Peptidbindung gegenüber der Esterbindung (zwischen tRNA und AS). Hierbei wird die Peptidbindung zwischen der Carboxygruppe (C-Termius) der bestehenden Peptidkette und der Aminogruppe (N-Termius) der neuen Aminosäure gebildet. Das Wachstum der Peptidkette erfolgt folglich vom N- zum C-Termius. Die katalytische Aktivität hat bei dieser Reaktion die 28S rRNA; das Ribosom ist also ein Ribozym. Durch die Reaktion wird die Bindung des Formyl-Methionins an die Initiator-tRNA gebrochen. Die Initiator-tRNA ist also entladen und es entsteht ein Dipeptid an der zweiten tRNA. Das Peptid bleibt in der P-Stelle der 50S UE (Untereinheit) des Ribosoms, die 30S UE mit mRNA und die beiden tRNAs befinden sich aber noch in ihrer Ausgangsposition.
Der Grund für die Formylierung des Methionins an der Aminogruppe (N-Termius) wird nun ersichtlich. Das Dipeptid, welches nach der ersten Elongationsrunde gebildet wird, besitzt die Möglichkeit zum Ringschluss. Der Stickstoff des Methionins hätte die Möglichkeit eines nukleophilen Angriffs auf das Carboxyl-Kohlenstoffatom der zweiten Aminosäure. Es käme zum Ablösen des Dipeptids von der tRNA und somit zur Termination der Elongation.
Wiederherstellung der Ausgangssituation
Die Translokation (Bewegung) der entladenen Initiator- sowie der zweiten tRNA erfolgt durch den Elongationsfaktor-G (EF-G). Dieser besitzt eine molekulare Ähnlichkeit zu dem EF-Tu-tRNA-Komplex. Es wird vermutet, dass der EF-G, ähnlich dem EF-Tu, an die 50S UE bindet. Der untere Part des EF-G ähnelt einer tRNA, eine Wechselwirkung mit der 30S UE liegt also nahe. Da es sich bei EF-G wiederum um ein G-Protein handelt, unterliegt dieser bei GTP-Hydrolyse einer Konformationsänderung, er verändert also seine Struktur. Dadurch wandert der tRNA-ähnliche Teil Richtung Ribosomeninnenraum und "stößt" die beiden tRNAs eine Position weiter. Die Initiator-tRNA befindet sich nun an der E-Stelle, die zweite tRNA in der P-Stelle. Mit der Dissoziation von EF-G ist der Ausgangszustand wieder erreicht. Eine neue tRNA kann an die A-Stelle binden und das Wachstum der Peptidkette vorantreiben.
Literatur
- Kapp LD, Lorsch JR: The molecular mechanics of eukaryotic translation. In: Annu. Rev. Biochem.. 73, 2004, S. 657–704. doi:10.1146/annurev.biochem.73.030403.080419. PMID 15189156.
Weblinks
- M. Anand, B. A. Balar, S. Gross, P. A. Ortiz, S. Ozturk, Y. R. Pittman, R. Ulloque, T. G. Kinzy: Eukaryotic Translation Elongation. In: Reactome - a curated knowledgebase of biological pathways. 13, 2005, doi:10.3180/REACT_1477.1.