Vibrio-Virus CTXphi
Vibrio-Virus CTXphi (offiziell Vibrio virus CTXphi) ist eine Spezies (Art) filamentöser Bakteriophagen mit einer unsegmentierten Einzelstrang-DNA (ssDNA) positiver Polarität und zirkulär geschlossener Topologie. Es ist die einzige Spezies der Gattung Affertcholeramvirus innerhalb der Familie Inoviridae (Ordnung Tubulavirales).[1]
| Vibrio-Virus CTXphi | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Vibrio virus CTXphi | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| CTXφ, CTX-nct | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Der Referenzstamm der Spezies Vibrio-Virus CTXphi ist Vibrio-Phage CTXphi (auch Choleratoxin-Phage, englisch Vibrio phage CTXphi, CTXφ oder CTXΦ).[Anm. 1] In der Spezies Vibrio-Virus CTXphi gibt es neben diesem Referenzstamm CTXφ nach NCBI einen weiteren Stamm Vibrio-Phage CTX-nct (en. Vibrio phage CTX-nct, CTX-nct)[2] bzw. Vorläufer (pre-CTXΦ), wie CTXETΦ, CTXClassΦ, CTXVarΦ, CTXCalcΦ and CTXEnvΦ.[3]
CTXφ infiziert einige Stämme von Vibrio cholerae (Cholera-Bakterium), sowie von V, mimicus [en], das auch eine Durchfallerkrankung hervorrufen kann.[4]
Genom
Das Genom von CTXφ ist 6,9 kb (Kilobasen) lang und besteht aus zwei Regionen.
- Die Gene der Kernregion kodieren für das Choleratoxin CTX selbst und einige Proteine, von denen angenommen wird, dass sie als Kapsidproteine das Kapsid des Virus bilden:[5][6]
- Die Gene ctxA und ctxB (zusammen als ctxAB bezeichnet) kodieren für das Choleratoxin CTX, einem Hexamer mit einer Untereinheit A und fünf Untereinheiten B.
- das größere: MCP, hier genannt Cep (kodiert von Gen cep)
- einige kleinere: mCPs, hier genannt Psh (Gen psh), OrfU alias pIIICTX (Gen orfU), Ace (Gen ace)
- und ein Nicht-CT-Toxin (Zot – Gen zot)
- Die sog. RS2-Region des Phagengenoms enthält Gene, die Replikation, Regulation und Integration von CTXφ in das Wirtsgenom steuern.[4]
CTXφ ist in der Lage, die Choleratoxin-Gene ctxAB von einem V. cholerae-Stamm auf einen anderen zu übertragen.[6][4]
Insertion, Replikation und Freisetzung aus der Wirtszelle
Dae tcpA-Gen der pathogenen Bakterienstämme kodiert für den sog. TCP-Faktor (Toxin-regulierter Pilus, en. toxin-coregulated pilus), wodurch das Bakterium in der Lage ist, spezielle Pili (Typ IV Pili) auszubilden.[7][4] Diese filamentösen Zelloberflächenstrukturen wirken als Adhäsine und ermöglichen somit das Anheften der Bakterienzellen an die Darmzellwand, d. h. an die Oberfläche der Mikrovilli der Darmzellen.[8][7]
Das Eindringen der freien CTXφ-Viruspartikel in die Wirtsbakterien wird durch zwei Rezeptoren vermittelt:[9]
- Der erste ist der TCP-Faktor. Es wird angenommen, dass der TCP an das kleinere CTXφ-Kapsidprotein (en. minor capsid protein, mCP) bindet, das hier als OrfU bezeichnet wird.
- Der zweite ist die TolQRA-Membranproteinstruktur (kodiert durch das Gen tolQRA)
Diese Anforderungen für die Bindung der Phagenmembran sind denen der Ff-Phagen (Gattung Inovirus, ebenfalls Familie Inoviridae) recht ähnlich: Diese infizieren Kolibakterien (Escherichia coli) und benötigen dazu als Rezeptoren den F-Pilus zusammen mit der TolQRA-Struktur; siehe Inovirus §Infektion. In E. coli wirkt TolQRA, um den Ff-Phagen in den periplasmatischen Raum zu verbringen (translozieren), wo eine mögliche Membranfusion zur Insertion des Ff-Genoms in das E. coli-Zytoplasma führt. Ein ähnlicher Mechanismus wird bei der Injektion des CTXφ-Genmaterials in die V. cholerae-Zelle vermutet, obwohl weitere Untersuchungen erforderlich sind, um dies zu bestätigen.[10]
Nach der Insertion der ssDNA des Phagen in das Zytoplasma von V. cholerae wird ein komplementärer DNA-Strang gebildet, um die Plasmidform des viralen Genoms, pCTX, zu erzeugen. pCTX kann durch DNA-Replikation neue ssDNA-Genome erzeugen und/oder als Prophage in das bakterielle Genom eingebaut werden. Da dieser Prophage in einigen Biotypen von V. cholerae im Tandem vorhanden sein kann (s. o.), schließen sich horizontaler Gentransfer (HGT) und vertikale Übertragung (Vererbung) der hinzugefügten CTXφ-Gene nicht gegenseitig aus.[9]
Nach der Produktion der Proteine und des Genom-Materials, die notwendig sind, um neue exogene Viruspartikel des Bakteriophagen (als Nachkommen) zu erzeugen, assemblieren die Proteine an einem Proteinkomplex, dem EpsD-Sekretin. Sobald sich das neue ssDNA-Genom innerhalb der assemblierten Proteine befindet, löst sich das CTXφ-Virus vom EpsD und ist frei, um andere Bakterien zu infizieren. Phagenpartikel werden ohne Lyse aus Bakterienzellen ausgeschieden (sezerniert).[9]
Prophagenstadium
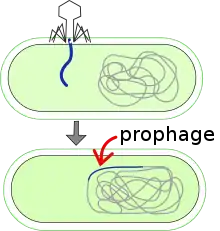
CTXφ ist ein temperenter Phage, d. h. im Allgemeinen in das Genom des V. cholerae-Bakteriums (als Prophage) integriert, seltener als Virion (Viruspartikel) außerhalb des Bakteriums anzutreffen.
Das Genom von V. cholerae verteilt sich auf zwei zirkuläre (ringförmige) Segmente (bipartit), oft auch „Bakterienchromosome“ genannt. Dies ist für Bakterien ungewöhnlich, da die meisten Bakterien nur ein einziges kovalent geschlossenes, ringförmiges Doppelstrang-DNA-Molekül besitzen. Man nimmt an, dass das kleinere ursprünglich ein Plasmid („Megaplasmid“) war, denn es umfasst Gene, die typischerweise auf einem Plasmid lokalisiert sind, nicht aber im Genom der Gammaproteobacteria, zu denen die Gattung Vibrio gehört.[11]
Die Bildung des Choleratoxins ist im Genom der pathogenen V. cholerae-Stämme kodiert, so bei den Serogruppen V. cholerae O1 und O139.[8]
Abhängig vom Stamm von V. cholerae gibt es zwei Möglichkeiten:[4]
- bei den klassischen Biotypen der Serogruppe O1 von befinden sich die Prophagen von CTXφ auf jedem der beiden bakteriellen Genom-Segmente
- beim El Tor-Biotyp [en] jedoch sind sie in einer Tandem-Anordnung (en. tandemly arranged genes, TAGs [en]) auf dem größeren der beiden Genomsegmente
Ähnlich ist es bei V. mimicus:[4]
- Stamm PT5 enthält zwei CTX-Prophagen, die an verschiedenen Stellen im V. mimicus-Genom integriert sind
- die Stämme PT48, 523-80 und 9583 enthalten jeweils tandemartig angeordnete Kopien von CTXφ
Es wurde zudem festgestellt, dass der CTX-Prophage im Stamm PT5 infektiöse CTXΦ-Partikel (Virionen) produziert. Die Sequenzen der CTXφ-Gene orfU und zot aus V. mimicus Stamm PT5 und V. cholerae Stamm N16961 waren identisch, was horizontalen Gentransfer vermuten lässt.[4]
Beim Biotyp El Tor von V cholarae ist der CTXφ Prophage Genom oft von einer Region (bezeichnet als RS1) mit 2,7 kb Länge flankiert, diese ist ähnlich wie RS2, hat aber ein zusätzliches Gen.[4]
Nicht-CT-Toxine
Neuere Forschungen legen nahe, dass noch mindestens zwei andere Toxine (en. Non-CT toxins) neben CTX von Genen des CTXφ-Genoms produziert werden.
Das erste von ihnen ist das akzessorische Cholera-Enterotoxin (en. accessory cholera enterotoxin, Ace). Es wird angenommen, dass Ace ein untergeordnetes Hüllprotein des Virionstadiums von CTXφ ist. Der Prozess, durch den das Toxin aus der Proteinhülle freigesetzt werden könnte, ist aber noch nicht identifiziert.
Das zweite Nicht-CT-Toxin, das im CTXφ-Genom kodiert wird, ist das Zonula-occludens-Toxin (en. zonula occludens toxin, Zot).[12] Zot ist zwar für die Produktion des CTXφ-Virions absolut notwendig, ist aber im Phagenpartikel selbst nicht vorhanden. Die Rolle, die Ace und Zot bei der Virulenz der Cholera spielen, ist noch nicht sehr klar.[9]
VPIΦ
Im Bakteriengenom bezeichnet man die Abschnitte, die Virulenzfaktoren kodieren, als Pathogenitätsinseln (en. pathogenicity island, PI), speziell bei den Vibrionen wie V. cholerae wird diese Pathogenitätsinsel als VPI (Vibrio PI) bezeichnet. Auf der VPI finden sich das tcpA-Gen für den TCP-Faktor (Toxin-regulierter Pilus, en. toxin-coregulated pilus), ein spezieller Typ IV Piluss.[7][4] Diese Pili ermöglichen das Anheften der Bakterienzellen an die Darmzellwand und sind daher ein weiterer Virulenzfaktor.[8][7] Die VPI befindet sich in der Nähe des ssrA-Gens des Wirtsbakteriums.[4]
Es wird vermutet, dass die Gene der Pathogenitätsinsel von einem weiteren Bakteriophagen stammen. In einer Untersuchung aus dem Jahr 1999 wurde gezeigt, dass die tcp-Gene der VPI identisch sind mit denen eines Phagen, der folglich als VPIΦ („flamentous bacteriophage VPIphi“, „Vibrio Pathogenicity Island Phage“) bezeichnet wurde.[7][4] VPIΦ ist demzufolge verantwortlich für den horizontalen Gentransfer zwischen V. cholerae Stämmen. Weiterhin wurde die TcpA-Untereinheit des Typ IV Pilus als Kapsidprotein des Bakteriophagen VPIΦ erkannt.[7][13]
Die Sequenzen der VPIΦ-Gene aldA und toxT aus V. mimicus Stamm PT5 und aus V. cholerae Stamm N16961 waren identisch, was auf einen horizontalen Transfer dieses Phagen zwischen V. mimicus und V. cholerae vor nicht allzu langer Zeit hindeutet. In V. mimicus wurde der VPI-Prophage an der gleichen chromosomalen Anheftungsstelle wie in V. cholerae integriert. Diese Ergebnisse legen nahe, dass V. mimicus ein bedeutendes Reservoir sowohl für CTXΦ als auch für VPIΦ sein kann.[4]
VCYΦ
Die Spezies Vibrio coralliilyticus [en] steht im Verdacht, für Korallenbleiche (mit)verantwortlich zu sein. Anhand von fünf vollständig sequenzierten Stämmen von V. coralliilyticus wurde geprüft, ob in den Genomen Prophagen gefunden werden können, die den Stämmen Virulenzfaktoren vermitteln und so ihre Toxizität herbeiführen.[14]
Stämme von V. corallilyticus, bei denen im Genom intakte Prophagen gefunden wurden (mit Prophagen-Genomlänge in kb):[14]
- BAA450 — Vibrio-Phage VCYΦ — 7,8[15]
- P1 — Pseudomonas-Phage φCTX — 20[16][Anm. 2]
- OCN008 — Vibrio-Phage K139 — 39,3[17]
- OCN014 — Vibrio-Phage VP882 — 40,8[18]
- RE98 — Vibrio-Phage vB_VpaM_MAR — 13,3[19]
Vibrio-Phage VCY-phi (VCYΦ oder VCYφ) ist ein Stamm der Spezies Vibrio-Virus VCY (en. Vibrio virus VCY, Gattung Vicialiavirus) in der Familie Inoviridae — die anderen Phagen sind alle Myoviren aus der Klasse Caudoviricetes.[15] Das VCYφ-Prophagengenom weist Homologie (Sequenzähnlichkeit) mit dem CTXφ-Prophagen auf und ist offenbar wie CTXφ ein evolutionär junges Virus, das sich durch HGT-Ereignisse und Rekombination entwickelt hat. Ein Prophagengenom, das für eine Reihe von Genen kodiert, die homolog zu den von VCYφ kodierten sind, wurde auch im P1-Stamm nachgewiesen.[14]
Auch in anderen Spezies der Gattung Vibrio wurden Prophagen gefunden, die Virulenzfaktoren darstellen.[20]
Anmerkungen
- Der Vibrio-Phage CTXphi ist wohl zu unterscheiden von Pseudomonas-Phage PhiCTX, dem Referenzstamm der Spezies Pseudomonas virus phiCTX [en], Gattung Citexvirus (Myoviridae), dessen Wirte Bakterien der Gattung Pseudomonas sind. Der Phage trägt ein anderes (ebenfalls) als ctx bezeichnetes Gen für ein (nicht näher) als Cytotoxin bezeichnetes Exotoxin.
Referenzen:
- NCBI: Pseudomonas virus phiCTX (species)
- SIB: Modulation of host virulence by virus und Viral exotoxin, auf: ViralZone
- Augenscheinlich ist beim Stamm P1 der Pseudomonas-Phage φCTX irrtümlich als CTXφ bezeichnet
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- NCBI: Vibrio phage CTX-nct (no rank)
- Talena Ledón, Javier Campos, Edith Suzarte, Boris Rodríguez, Karen Marrero, Rafael Fando: El Tor and Calcutta CTXΦ precursors coexisting with intact CTXΦ copies in Vibrio cholerae O139 isolates, in: Research in Microbiology, Band 159, Nr. 2, März 2008, S. 81–87, doi:10.1016/j.resmic.2007.11.015
- E. Fidelma Boyd, Kathryn E. Moyer, Lei Shi, Matthew K. Waldor: Infectious CTXΦ and the Vibrio Pathogenicity Island Prophage in Vibrio mimicus: Evidence for Recent Horizontal Transfer between V. mimicus and V. cholerae. In: Infection and Immunity. 68, Nr. 3, 2000, S. 1507–1513. doi:10.1128/IAI.68.3.1507-1513.2000. PMID 10678967. PMC 97308 (freier Volltext).
- Andrew J. Heilpern, Matthew K. Waldor: pIIICTX, a predicted CTXphi minor coat protein, can expand the host range of coliphage fd to include Vibrio cholerae, in: J Bacteriol 185(3), Februar 2003, S. 1037–1044, doi:10.1128/jb.185.3.1037-1044.2003, PMID 12533480, PMC 142820 (freier Volltext)
- E. Fidelma Boyd: Efficiency and specificity of CTXϕ chromosomal integration: dif makes all the difference. In: Proceedings of the National Academy of Sciences of the United States of America. 107, Nr. 9, 2010, S. 3951–3952. bibcode:2010PNAS..107.3951B. doi:10.1073/pnas.1000310107. PMID 20197438. PMC 2840105 (freier Volltext).
- David K. R. Karaolis, Sita Somara, David R. Maneval, Judith A. Johnson, James B. Kaper: A bacteriophage encoding a pathogenicity island, a type-IV pilus and a phage receptor in cholera bacteria. In: Nature. Band 399, Nummer 6734, Mai 1999, S. 375–379, doi:10.1038/20715. PMID 10360577.
- M. Li, M. Kotetishvili, Y. Chen, S. Sozhamannan: Comparative genomic analyses of the vibrio pathogenicity island and cholera toxin prophage regions in nonepidemic serogroup strains of Vibrio cholerae. In: Applied and environmental microbiology. Band 69, Nummer 3, März 2003, S. 1728–1738, PMID 12620865. PMC 150053 (freier Volltext).
- Brigid M. Davis, Matthew K. Waldor: Filamentous phages linked to virulence of Vibrio cholerae. In: Current Opinion in Microbiology. 6, Nr. 1, 2003, S. 35–42. doi:10.1016/S1369-5274(02)00005-X. PMID 12615217.
- Andrew J. Heilpern, Matthew K. Waldor: CTXφ Infection of Vibrio cholerae Requires the tolQRA Gene Products. In: Journal of Bacteriology. 182, Nr. 6, 2000, S. 1739–1747. doi:10.1128/JB.182.6.1739-1747.2000. PMID 10692381. PMC 94473 (freier Volltext).
- John F. Heidelberg, Jonathan A. Eisen u. a.: DNA sequence of both chromosomes of the cholera pathogen Vibrio cholerae. In: Nature. Band 406, Nummer 6795, August 2000, S. 477–483, doi:10.1038/35020000. PMID 10952301.
- Eugene V. Koonin: The second cholera toxin, Zot, and its plasmid-encoded and phage-encoded homologues constitute a group of putative ATPases with an altered purine NTP-binding motif, in: FEBS Lett 312(1), 2. November 1992, S. 3–6, doi:10.1016/0014-5793(92)81398-6
- SIB: Modulation of host virulence by virus, auf: ViralZone
- Karen D. Weynberg, Christian R. Voolstra, Matthew J. Neave, Patrick Buerger, Madeleine J. H. van Oppen: From cholera to corals: Viruses as drivers of virulence in a major coral bacterial pathogen, in: Scientific Reports Band 5, Nr. 17889 (2016), Epub 8. Dezember 2015, doi:10.1038/srep17889
- NCBI: NC_016162 Tubulavirales; Inoviridae; Vicialiavirus; Vibrio virus VCY
- NCBI: NC_003278.1 Myoviridae; Peduovirinae; Citexvirus; Pseudomonas virus phiCTX
- NCBI: NC_003313 Myoviridae; Peduovirinae; Longwoodvirus; Vibrio virus K139
- NCBI: NC_009016 Myoviridae; Hapunavirus; Vibrio virus VP882
- NCBI: NC_019722.1 Myoviridae; Vhmlvirus; Vibrio virus MAR
- Daniel Castillo, Kathryn Kauffman, Fatima Hussain, Panos Kalatzis, Nanna Rørbo, Martin F. Polz, Mathias Middelboe: Widespread distribution of prophage-encoded virulence factors in marine Vibrio communities/s41598-018-28326-9, in: Scientific Reports Band 8,Nr. 9973, 2. Juli 2018, doi:10.1038/s41598-018-28326-9
Weblinks
- Bhabatosh Das, G. Balakrish Nair, Rupak K. Bhadra: Acquisition and dissemination mechanisms of CTXΦ in Vibrio cholerae: New paradigm for dif residents, in: World J Med Genet 4(2), 27. Mai 2014, S. 27–33 doi:10.5496/wjmg.v4.i2.27
- Justin T. Cruite, Gabriela Kovacikova, Kenzie A. Clark, Anne K. Woodbrey, Karen Skorupski, F. Jon Kull: Structural basis for virulence regulation in Vibrio cholerae by unsaturated fatty acid components of bile, in: Commun Biol 2, 440, 28. November 2019, doi:10.1038/s42003-019-0686-x

- Simon Roux, Mart Krupovic, Rebecca A. Daly, Adair L. Borges, Stephen Nayfach, Frederik Schulz et al.: Cryptic inoviruses revealed as pervasive in bacteria and archaea across Earth’s biomes, in: Nat Microbiol 4, 22. Juli 2019, S. 1895–1906, doi:10.1038/s41564-019-0510-x

- Andrew J. Heilpern, Matthew K. Waldor: pIIICTX, a predicted CTXphi minor coat protein, can expand the host range of coliphage fd to include Vibrio cholerae, in: J Bacteriol 185(3), Februar 2003, S. 1037–1044, doi:10.1128/jb.185.3.1037-1044.2003, PMID 12533480, PMC 142820 (freier Volltext)
- E Fidelma Boyd: Bacteriophage-Encoded Bacterial Virulence Factors and Phage-Pathogenicity Island Interactions, in: Advances in Virus Research 82, Dezember 2012, S. 91–118, doi:10.1016/B978-0-12-394621-8.00014-5, PMID 22420852