Ribonukleotidreduktase
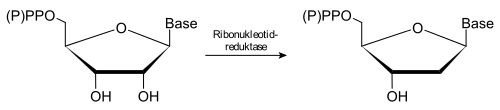
Die Ribonukleotidreduktase (oft auch als RNR abgekürzt) ist ein Enzym, welches das letzte Glied in der Kette der Synthese der DNA-Bausteine (Desoxynukleotide) bildet. Es reduziert, d. h. entfernt die Hydroxygruppe am Kohlenstoffatom C-2' des Ribose-Teils der Nukleotide. Substrate des Enzyms sind Ribonukleotid-phosphate, z. B. Adenosindiphosphat und Cytidindiphosphat, in Verbindung mit dem Kofaktor Thioredoxin.
| Ribonukleotiddiphosphat-Reduktase, große Untereinheit | ||
|---|---|---|
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 792 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Heterodimer | |
| Bezeichner | ||
| Gen-Namen | RRM1 R1; RIR1; RR1 | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.17.4.1, Oxidoreduktase | |
| Reaktionsart | Reduktion einer Hydroxygruppe | |
| Substrat | Ribonukleotid-Diphosphat + Thioredoxin | |
| Produkte | Deoxyribonukleotid-Diphosphat + Thioredoxindisulfid + H2O | |
| Vorkommen | ||
| Übergeordnetes Taxon | Lebewesen[1] | |
| Ausnahmen | Archaea | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 6240 | 20133 |
| Ensembl | ENSG00000167325 | ENSMUSG00000030978 |
| UniProt | P23921 | P07742 |
| Refseq (mRNA) | NM_001033 | NM_009103 |
| Refseq (Protein) | NP_001024 | NP_033129 |
| Genlocus | Chr 11: 4.09 – 4.14 Mb | Chr 7: 102.44 – 102.47 Mb |
| PubMed-Suche | 6240 | 20133 |
| Ribonukleotiddiphosphat-Reduktase, kleine Untereinheit | ||
|---|---|---|
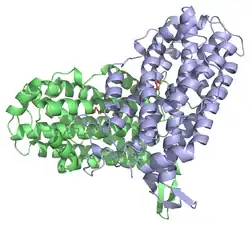 | ||
| Bändermodell des Dimer der R2-Untereinheit der Class I-RNR von Escherichia coli, nach PDB 1AV8 | ||
| Eigenschaften des menschlichen Proteins | ||
| Masse/Länge Primärstruktur | 389 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Heterodimer | |
| Kofaktor | 2 Fe2+ | |
| Bezeichner | ||
| Gen-Namen | RRM2 R2; RR2; RR2M | |
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.17.4.1, Oxidoreduktase | |
| Reaktionsart | Reduktion einer Hydroxygruppe | |
| Substrat | Ribonukleotid-Diphosphat + Thioredoxin | |
| Produkte | Deoxyribonukleotid-Diphosphat + Thioredoxindisulfid + H2O | |
| Vorkommen | ||
| Homologie-Familie | RNR subunit M2 | |
| Übergeordnetes Taxon | Euteleostomi | |
| Orthologe | ||
| Mensch | Hausmaus | |
| Entrez | 6241 | 20135 |
| Ensembl | ENSG00000171848 | ENSMUSG00000020649 |
| UniProt | P31350 | P11157 |
| Refseq (mRNA) | NM_001034 | NM_009104 |
| Refseq (Protein) | NP_001025 | NP_033130 |
| Genlocus | Chr 2: 10.12 – 10.13 Mb | Chr 12: 24.71 – 24.71 Mb |
| PubMed-Suche | 6241 | 20135 |
Bedeutung und Vorkommen
Die Ribonukleotidreduktase ist ein unentbehrliches Enzym im Organismus, um DNA-Bausteine herzustellen. Es wurde in fast allen eukariontischen Organismen gefunden, von Hefe und Algen bis zu Pflanzen, Säugetieren und Menschen. In einigen Prokarionten und Viren sind ebenfalls RNR-Enzyme enthalten.
Da das Enzym die 2'-Hydroxy-Gruppe der Nukleotide reduziert, kann es auch als Bindeglied zwischen der DNA- und RNA-Welt gesehen werden. In diesem Zusammenhang wird auch spekuliert, ob RNR das Enzym war, welches den Weg aus einer primitiven RNA- zur heutigen DNA-Welt ebnete. (Siehe dazu auch: Chemische Evolution und Ribozym)
Wie viele Enzyme katalysiert die Ribonukleotidreduktase auch die entsprechende Rückreaktion (Oxidation von Desoxynukleotid zum Nukleotid). Diese Reaktion spielt für biologische Prozesse allerdings keine Rolle.
Biologische Funktion
Die Ribonukleotidreduktase transformiert die Nukleotide in ihre jeweiligen Desoxynukleotide - ohne zu unterscheiden um welches Nukleotid es sich handelt (Adenosin-, Guanosin-, Cytidin- oder Thymidin-Phosphate). Sowohl Di- als auch die jeweiligen Triphosphate werden reduziert, nicht aber Monophosphate und (phosphatfreie) Nukleoside.
Die Reduktion erfolgt vermutlich durch einen Elektronentransfer-Mechanismus, bei dem – wie unten erläutert – freie Radikale entstehen.
Es gibt mehrere Klassen der Ribonukleotidreduktase (Class I bis Class III). Obwohl alle Klassen die gleiche Reaktion katalysieren, sind die Enzyme strukturell sehr verschieden. Die Klassen sind nach der Art eingeteilt, wie das System das Radikal erzeugt. Jede Klasse teilt sich in weitere Unterklassen ein, dies wird hier aber nicht ausgeführt.
Class I
Class I (EC 1.17.4.1) ist der Enzymtyp, welcher im Menschen vorkommt und am besten untersucht ist. Die Enzyme sind aerob.
Class I-RNR sind aus zwei verschiedenen Untereinheiten aufgebaut: R1 und R2.[2] R1 und R2 haben eine sehr geringe Affinität, so dass das Enzym selten als Gesamtkomplex vorliegt. Dies vermutet man als Grund, dass man bisher keine Ribonukleotidreduktase als Ganzes kristallisieren konnte.
R1 beinhaltet mehrere Bindungsstellen für Effektoren, sowie die Bindungstasche (active site), in der das Nukleotid reduziert wird. In R2 wird ein Radikal auf einer Tyrosinseitenkette übertragen, dort sozusagen ‚gespeichert‘. Dieses Radikal hat eine erstaunlich hohe Halbwertszeit von etwa vier Tagen.
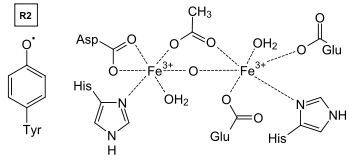
Für die Katalyse muss das Elektron vom Tyrosylradikal in R2 auf ein Cystein in R1 transportiert werden.[3] Der Abstand wird dabei auf 35 Ångström geschätzt. Vermutlich überspringt das Elektron dabei mehrere Aminosäureeinheiten, um die (für molekulare Verhältnisse) enorm weite Strecke zurückzulegen.
Class II
Class II-RNR (EC 1.17.4.2) kommen unter anderem im Organismus Lactobacillus leichmannii vor. Hier wird das Radikal in situ generiert, indem im Coenzym B12 die Cobalt-Kohlenstoff-Bindung gebrochen wird. Enzyme dieser Klasse sind fakultativ aerob. Sie können in An- als auch in Abwesenheit von Sauerstoff arbeiten.
Class III
Hier wird das Radikal auf eine Glycyl-Seitenkette übertragen. Bakteriophage T4 zum Beispiel arbeitet mit dieser Klasse des Enzyms. Class III-RNR sind anaerob, das heißt, sie arbeiten nur in einer sauerstofffreien Umgebung.
Die RNR-Enzyme könnten einst aus einem gemeinsamen Vorfahren entstanden sein. Von den anaerob wirkenden Enzymen der Klasse III steht vermutlich die Reduktase von Escherichia coli dem hypothetischen „Urenzym“ am nächsten.[4][5]
Mechanismus der Reduktion
Der genaue Ablauf der Nukleotid-Reduktion ist bisher nicht vollständig aufgeklärt. Es bestehen deutliche Hinweise auf einen Radikalmechanismus, wobei der zentrale Schritt in der Bildung eines Thiylradikals, eines schwefelzentrierten Radikals, bestehen kann.[6][7] Dazu speichert das Enzym ein stabiles Radikal, welches bei jedem Turn-Over in die Bindungstasche transportiert werden muss. Nach der chemischen Reaktion wird das Radikal zurückgewonnen und wieder zur ‚Speicherstelle‘ zurücktransportiert. Dabei wird angenommen, dass das Elektron über mehrere Aminosäure-Einheiten „hüpft“, die alle über Wasserstoffbrücken verbunden sind.
Im folgenden Schema ist der Ablauf mit den vermuteten Zwischenschritten aufgeführt, wie er – Stand 1998 – in der Wissenschaft diskutiert wird.[8]

- Zunächst wird ein Radikal auf die 3'-Position der Ribose übertragen (Schritt vom 1. zum 2. Bild).
- Nach Abspaltung von Wasser an der 2'-Position (Bild 2 → 3) wird das Radikal auf zwei Cysteinketten des Enzyms übertragen (Bild 3 → 4).
- Danach wird das Radikal über Ribose zurück auf das ursprüngliche Cystein übertragen (Bild 4 → 6).
Insgesamt wird unter Oxidation von zwei Cystein-Seitenketten die Ribose zur Desoxyribose reduziert. Aus einem RNA- wurde ein DNA-Baustein hergestellt. Dieser Baustein ist nun bereit, um von der DNA-Polymerase in die DNA-Doppelhelix eingebaut zu werden.
Regulation
Da die RNR nur in speziellen Phasen der Zellteilung aktiv sein muss, gibt es wie bei den meisten Enzymen Mechanismen, um die RNR an- bzw. abzuschalten. Zudem wird in einem komplizierten System genau geregelt, welches der vier Nukleotide reduziert werden soll. Hier sollen nur kurz die Aktivatoren und Effektoren aufgezählt werden, ohne genauer auf die Details einzugehen:
Aktivatoren:
- ATP aktiviert das Enzym
- dATP deaktiviert es
Effektoren:
- ATP und dATP → CDP/CTP und UDP/UTP werden reduziert
- dGTP → ADP/ATP
- dTTP → GDP/GTP
Entdeckung der Ribonukleotidreduktase
Seit der Strukturaufklärung der DNA durch James Watson und Francis Crick im Jahr 1953 stellte sich die Frage, wie die Zelle die einzelnen Bausteine für das DNA-Polymer herstellt. Acht Jahre später wurde zum ersten Mal aus verschiedenen Zellen ein Enzymgemisch isoliert mit hoher Aktivität, Nukleotide in ihre entsprechenden Desoxynukleotide zu reduzieren.[9]
In den 1990er Jahren wurden die dreidimensionalen Strukturen der beiden Untereinheiten separat mittels Kristallstrukturuntersuchungen ermittelt.[2][10] Fragen nach dem Mechanismus der Andockung der beiden Untereinheiten R1 und R2 aneinander, dem Radikaltransport durch das Enzym über eine verhältnismäßig große Distanz hinweg bzw. nach der Generierung des Radikals bei der Herstellung des Enzyms werden untersucht.
Die RNR in der Krebsforschung und Krebstherapie
Die Ribonukleotidreduktase steht auch im Fokus der Krebsforschung. Weil das Enzym immer dann benötigt wird, wenn die Zelle sich teilt oder DNA-Schäden reparieren muss, ist die Zelle beim Wachstum auf die RNR angewiesen. Das Enzym ist mit einer Umsatzrate von ca. 10 s−1 vergleichsweise langsam. Das ist aufgrund der langsamen Teilungsrate der normalen Zelle nicht problematisch, Krebszellen werden aber so an raschem Wachstum gehemmt. Es gibt jedoch Krebszellen, die die Umsatzrate der RNR durch Modifizierungen erhöhen. Ein Wirkstoff, der genau diese modifizierten Enzyme blockiert, würde das Krebswachstum verlangsamen oder sogar stoppen.[11]
Inzwischen wurden zahlreiche Hemmstoffe der RNR entwickelt.[12] So wird zur Behandlung von myeloischen Leukämien (vor allem bei Anzeichen einer Leukostase) und anderen myeloproliferativen Erkrankungen wie essentielle Thrombozythämie und Polycythaemia vera (rubra) Hydroxycarbamid (z. B. Syrea®, Litalir®) als Chemotherapeutikum eingesetzt.
Literatur
- B. M. Sjöberg: Ribonucleotide reductases — a group of enzymes with different metallosites and a similar reaction mechanism, In: P. J. Sadler: Metal Sites in Proteins and Models - Iron Centres, In: Structure and Bonding Vol. 88, S. 139–173, Springer Verlag, ISBN 3-540-62870-3.
- JoAnne Stubbe, Wilfred A. van der Donk: Protein Radicals in Enzyme Catalysis, In: Chemical Reviews Jg. 1998, Bd. 98, S. 705–762.
Weblinks
Einzelnachweise
- Homologe bei OMA
- Ulla Uhlin, Hans Eklund: Structure of ribonucleotide reductase protein R1. In: Nature, Band 370, 1994, S. 533–539.
- Britt-Marie Sjöberg et al.: Two conserved tyrosine residues in R1 participate in an intermolecular electron transfer in ribonucleotide reductase. In: J. Biol. Chem., Band 271, Nr. 34, 1996, S. 20655–20659.
- Peter Reichard: From RNA to DNA, why so many ribonucleotide reductases?, In: Science, Bd. 260, Heft 5115, 18 Jun 1993, S. 1773–1777. DOI: 10.1126/science.8511586
- E. Torrents, P. Aloy, I. Gibert et al.: Ribonucleotide Reductases: Divergent Evolution of an Ancient Enzyme, In: Journal of Molecular Evolution, Jg. 2002, Bd. 55, S. 138–152. https://doi.org/10.1007/s00239-002-2311-7
- JoAnne Stubbe, Wilfred A. van der Donk: Protein Radicals in Enzyme Catalysis. In: Chemical Reviews. Band 98, Nr. 2, 1. April 1998, S. 705–762, doi:10.1021/cr9400875.
- JoAnne Stubbe, Daniel G. Nocera, Cyril S. Yee, Michelle C. Y. Chang: Radical Initiation in the Class I Ribonucleotide Reductase: Long-Range Proton-Coupled Electron Transfer? In: Chemical Reviews. Band 103, Nr. 6, 1. Juni 2003, S. 2167–2202, doi:10.1021/cr020421u.
- JoAnne Stubbe, Wilfred A. van der Donk: Protein Radicals in Enzyme Catalysis, In: Chemical Reviews Jg. 1998, Bd. 98, S. 705–762.
- Peter Reichard, Astor Baldesten, Lars Rutberg: Formation of deoxycytidine phosphates from cytidine phosphates in extracts from Escherichia coli. In: J Biol Chem., Band 236, Nr. 4, 1961, S. 1150–1157.
- P. Nordlund, B.-M. Sjöberg, H. Eklund: Three-dimensional structure of the free radical protein of ribonucleotide reductase. In: Nature, Band 345, 1990, S. 593–598.
- Yun Yen: Ribonucleotide reductase subunit one as gene therapy target. In: Clinical Cancer Research, Band 9, 2003, S. 4304–4308.
- J. Shao, B. Zhou, Bernard Chu, Y. Yen: Ribonucleotide Reductase Inhibitors and Future Drug Design, In: Current Cancer Drug Targets, Jg. 2006, Bd. 6, S. 409–431.