Streptococcus-pyogenes-Phage T12
„Streptococcus-pyogenes-Phage T12“ (englisch „Streptococcus pyogenes phage T12“, „Streptococcus phage T12“) ist eine vorgeschlagene Spezies von Viren, die die Bakterienart Streptococcus pyogenes infizieren (Bakteriophagen). Der natürlich vorkommende Wildtyp (Typusstamm) wird mit Bacteriophage T12 oder kurz Phage T12 bezeichnet. Die Spezies gehört zur Familie Siphoviridae in der Ordnung Caudovirales (Viren mit Kopf-Schwanz-Struktur).[2]
| „Streptococcus-pyogenes-Phage T12“ | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| „Streptococcus pyogenes phage T12“ | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| T12 | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Der Phage T12 wandelt einen an sich harmlosen Bakterienstamm in einen krankmachenden, virulenten Stamm um. Es trägt das speA-Gen, das für das Erythrogene Toxin A (en. erythrogenic toxin A [en], speA oder SPE-A abgekürzt) kodiert.[3] speA wird auch als Streptococcus-pyogenes-Exotoxin A, Scharlach-Toxin A oder einfach Scharlach-Toxin (en. scarlatina toxin) bezeichnet.[4][5][Anmerkung 1] Im Unterschied zu anderen Bakteriophagen mit lytischem Reproduktionszyklus, an dessen Ende das Virus durch Platzen der Wirtszelle freigesetzt und diese dabei abgetötet wird, liegt bei T12 ein so genannter lysogener Reproduktionszyklus vor. Dieser zeichnet sich durch die Fähigkeit des Virus-Genoms aus, Teil des Genoms der Wirtszelle zu werden und dort über Generationen stabil erhalten zu bleiben.[6] Phagen mit einem lysogenen Zyklus werden auch als temperente Phagen bezeichnet.[3] Durch diese Lysogenie, d. h. Integration des T12-Genoms (inklusive des speA-Gens) in das Bakteriengenom, kann das Bakterium jetzt das Erythrogene Toxin A produzieren. Damit ist ein harmloser, nicht-virulenter Stamm von Streptococcus pyogenes in einen virulenten Stamm umgewandelt.
Bakteriophage T12 ist zum Prototyp erklärt worden für alle verwandten Bakteriophagen mit dem speA-Gen, die S. pyogenes befallen. D. h., dass auch sein Genom prototypisch für die Genome aller solcher Phagen ist.[7] Es gilt damit als Hauptverdächtiger, die Kinderkrankheit Scharlach zu verursachen.[6]
Forschungsgeschichte
Die Möglichkeit, dass Bakteriophagen an der speA-Produktion beteiligt sind, wurde erstmals 1926 vorgeschlagen. Damals berichteten J. Cantacuzène und O. Boncieu, dass nicht-virulente Stämme von S. pyogenes durch ein übertragbares Element (en. some transferable element) in virulente Stämme transformiert wurden,[8] und M. Frobisher und J. H. Brown berichteten im darauf folgenden Jahr (1927) über ähnliche Ergebnisse. Diese Forschungsergebnisse wurden 1949 wurden durch K. F. Bingel bestätigt.[9][10][11] Im Jahr 1964 berichtete John B. Zabriskie, dass der Phage T12 die speA-Produktion durch Lysogenese in Stämmen verursachen konnte, d. h. wenn er in deren Genom eingegliedert wurde.[12][13] Johnson, Schlievert und Watson konnten 1980 dies bestätigen und zeigen, dass das Gen für die speA-Produktion von toxigenen (das Toxin produzierenden) Bakterienstämmen durch Lysogenese auf nicht-toxigene Stämme übertragen wurde. Sie zeigten, dass jede das Toxin produzierende Bakterienkolonie lysogen war, also das entsprechende Gen (speA) von T12 enthielt. Umgekehrt war keine der Kolonien, die das T12-Gen enthielten, negativ für das speA Toxin.[14]
Larry McKane und Joseph J. Ferretti bestätigten dies nochmals 1981, indem sie zeigten, dass der nicht-toxigene (kein Toxin produzierende, ungefährliche) S. pyogenes-Stamm T253 nach Infektion mit dem Bakteriophagen T12 den lysogenen Stamm T253(T12) bildet, der das Toxin (speA) produziert. Sie zeigten weiter, dass die Eigenschaft der Lysogenität alleine nicht ausreicht, das Gift zu produzieren:
- Die Variante (spontane Mutante) T12cp1 des Phagen T12 mit lytischem Reproduktionszyklus war in der Lage, T253 in einen toxigenen Stamm zu verwandeln.
- Der nicht nahe verwandte virulente (lytische)[Anmerkung 2] Bakteriophage A25 der Spezies „Streptococcus-Phage A25“[15] (vorgeschlagen, ebenfalls in der Familie Siphoviridae) hatte dagegen keinen Einfluss auf die Fähigkeit, das Toxin zu erzeugen (Toxigenität).
- Der temperate (lysogene) Bakteriophage H4489A der Spezies „Streptococcus-pyogenes-Phage H4489A“[16] (vorgeschlagen, ohne Familienzuordnung), der Lysogenität in T253 erzeugt, bewirkt in den Bakterien keine Toxigenität.
Nur T12 und nahe verwandte Stämme wie T12cp1 bewirkten also, dass die infizierten Bakterien das Toxin bilden konnten.[17]
Im Jahr 1983 veröffentlichten Lane P. Johnson und Patrick M. Schlievert eine Karte des T12-Genoms.[13] Im Jahr darauf fanden Johnson und Schlievert sowie Weeks und Ferretti unabhängig voneinander, dass der Bakteriophage T12 das Gen (genannt speA) für das Toxin (Protein) speA trägt.[10][18] 1986 fanden Johnson, Tomai und Schlievert heraus, dass im Genom von T12 gleich neben dem Toxin-Gen speA ein Abschnitt (attP genannt) liegt, der für die Anheftung der Phage-DNA an die Bakterien-DNA verantwortlich ist. Sie stellten zudem fest, dass alle Bakterienstämme, die das Toxin produzieren, entweder den Phagen T12 selbst oder einen eng verwandten Bakteriophagen tragen.[6] Schließlich publizierten McShan und Ferretti 1997, dass sie eine zweite Anheftungsstelle (attR) für T12 gefunden hatten. In einer weiteren Publikation zusammen mit Y. F. Tang enthüllten sie, dass der Bakteriophage T12 sich im Wirtsbakterium in ein Gen einfügt, das für eine Serin-tRNA kodiert.[3][7]
Genom
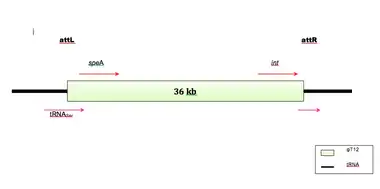
Das T12-Genom erwies sich als kreisförmig mit einer Gesamtlänge von 36,0 kb (Kilobasenpaare).[13] Das Phagengenom trägt das speA-Gen[18] mit einer Länge von 1,7 kbp, flankiert vom Gen für die Restriktionsenzyme SalI und HindIII.[10]
Das Phagen-Integrase-Gen (int) und das Gen für die Phagen-Anheftung (attP) befinden sich im Phagengenom direkt in Leserichtung aufwärts von speA. Der Bakteriophage T12 integriert sich in das Genom von S. pyogenes durch Rekombination in die Anticodon-Schleife eines Gens, das für die Serin-tRNA kodiert: Die bakterielle Anheftungsstelle (attB) hat eine 96 Basenpaare lange Sequenz, die homolog zur Phagen-Anheftungsstelle attP ist und sich am 3'-Ende des tRNA-Gens befindet, so dass die kodierende Sequenz des tRNA-Gens nach der Integration des Phagen zum Prophagen intakt bleibt. Der Phage T12 ist das erste Beispiel für einen Phagen aus einem gram-positiven bakteriellen Wirt mit niedrigem GC-Gehalt, der diese Art von Integration (englisch site specific recombination[19]) verwendet.[3][7]
Systematik
Für die vorgeschlagene Spezies „Streptococcus-pyogenes-Phage T12“ gibt es (wie auch für die oben erwähnte „Streptococcus-Phage A25“) derzeit (Stand Januar 2021) keine Gattungs-, nur die Familienzuweisung zu den Siphoviren (Siphoviridae). Die Spezies beinhaltet die folgenden Virustypen:[17]
- Bakteriophage T12 (kurz Phage T12) – Typusstamm (Wildtyp), lysogen
- Bakteriophage T12cp1 (kurz Phage T12cp1, mit Ziffer 1 am Ende) – eine spontane Mutante (Variante), lytisch[Anmerkung 4]
Pathogenese
Krankheiten wie Scharlach und das Streptokokken-induzierte Toxische Schocksyndrom (StrepTSS oder STSS) werden durch lysogenisierte Streptokokkenstämme verursacht, die das Toxin speA produzieren. Die Krankheiten sind systemische Reaktionen auf das im Körper zirkulierende Toxin.[20]
Scharlach
Scharlach, auch Scarlatina genannt, wird so wegen des charakteristischen hellroten Ausschlags genannt. Es tritt am häufigsten bei Kindern zwischen vier und acht Jahren auf.[21][22][23][24]
Streptokokken-induziertes TSS
Beim Streptokokken-induzierten Toxischen Schocksyndrom (StrepTSS oder STSS) wirkt das von infizierten Streptokokkenstämmen produzierte speA als Superantigen und interagiert mit menschlichen Monozyten und T-Lymphozyten, was die T-Zell-Proliferation und die Produktion von Monokinen bzw. Zytokinen (wie z. B. Tumornekrosefaktor α – TNFα, Interleukin-1, Interleukin-6) und Lymphokinen (wie z. B. Tumornekrosefaktor β – TNFβ, Interleukin 2 und Gamma-Interferon) induziert. Diese Zytokine (TNFα, TNFβ) scheinen das für die Krankheit charakteristische Fieber, den Schock und das Organversagen zu vermitteln.[20][25][26]
Diagnose und Therapie
Das Vorhandensein des lysogenen Bakteriophagen T12 kann durch Plaque-Assays getestet werden, wenn der verwendete Indikatorstamm für den zu testenden Phagen empfindlich ist. Plaque-Assays bestehen darin, eine Agarlösung mit einem Indikatorstamm auf eine Agarplatte zu gießen. Der Indikatorstamm sollte ein Bakterienstamm sein, der von dem nachzuweisenden Phagen infiziert werden kann. Nachdem der Weichagar ausgehärtet ist, werden die Proben, die auf das Vorhandensein von Phagen getestet werden sollen, auf die Agarplatten aufgetragen. Die Platten werden dann über Nacht bebrütet und am nächsten Tag auf Klärungen (Plaques) überprüft. Wenn der Phage vorhanden ist, werden die Indikatorstämme infiziert und durchlaufen, während die Platten bebrütet werden, den normalen lysogenen Zyklus. Danach werden sie lysiert, wodurch die Plaquen entstehen. Die Titer der Plaques können durch Verdünnen der Proben und Zählen der plaquebildenden Einheiten (englisch plaque-forming units, PFUs) ermittelt werden.[27]
Es können auch biochemische Tests wie Southern Blots verwendet werden, um das vom Phagen aus dem speA-Gen produzierte speA-Toxin nachzuweisen. Dies wurde erstmals bei diesem Toxin von Johnson, Tomai und Schlievert im Jahr 1985 durchgeführt, indem die DNA von Streptokokkenstämmen isoliert und ein Restriktionsverdau mit dem Restriktionsenzym BglII durchgeführt wurde. Nachdem der Verdau abgeschlossen war, wurden die DNA-Proben auf ein Gel gegeben, um die DNA zu trennen. Die DNA aus diesem Gel wurde dann auf Nitrozellulosepapier übertragen und mit für speA spezifischen Sonden inkubiert[Anmerkung 5].[6]
Bakteriophagen werden sehr leicht verbreitet.[28] Bei geringer Exposition kann ultraviolettes Licht die Produktion sowohl des Phagen T12 als auch von speA steigern.[5] Längere UV-Expositionszeiten können den Phagen abtöten.UV-Licht stresst lysogene Bakterien, was dazu führt, dass sich die Phagen vermehren und die bakteriellen Wirtszellen aufplatzen (Lyse).[29] Im Fall von T12 erhöht die Exposition mit UV-Licht die Vermehrung des Bakteriophagen T12 bei 20 Sekunden Exposition. Nach 20 Sekunden Exposition beginnt das UV-Licht, den Bakteriophagen zu töten, indem es ihr Genom schädigt.[12]
Weitergehende Forschung
- Für das Erythrogene Toxin C (en. erythrogenic toxin C [en], speC oder SPE-C abgekürzt) scheint ein „Streptococcus-pyogenes-Phage CS112“ (ΦCS112) verantwortlich.[30][31] Eine ganze Reihe weiterer Erkrankungen kann auf (temperente) Bakteriophagen zurückgeführt werden, die nicht-pathogene Bakterienstämme in pathogene (ein Toxin erzeugende) verwandeln.[31]
Anmerkungen
- Man beachte, dass der Name des Gens „speA“ kursiv geschrieben ist. Der Name des Toxins „speA“ wird dagegen normal, nicht kursiv, geschrieben.
- Dieser Phage erzeugt in den Bakterienkolonien typische Plaques, die den lytischen Reproduktionszyklus anzeigen. Bei diesem platzen am Ende die Wirtszellen, um die neu gebildeten Virusteilchen freizusetzen.
- Integrase
- gelegentlich als T12cpl mit Buchstabe l am Ende verschrieben
- Einwirkenlassen von Enzymen, Antikörpern etc. auf ein Substrat
Einzelnachweise
- ICTV: ICTV Taxonomy history: Enterobacteria phage T4, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- NCBI: Bacteriophage T12 (species)
- W. Michael McShan, Y.-F. Tang, Joseph J. Ferretti: Bacteriophage T12 of Streptococcus pyogenes integrates into the gene encoding a serine tRNA. In: Molecular Microbiology. 23, Nr. 4, 1997, S. 719–28. doi:10.1046/j.1365-2958.1997.2591616.x. PMID 9157243.
- Dennis L. Stevens, Martha H. Tanner, Jay Winship, Jay, Raymond Swarts, Kristen M. Ries, Patrick M. Schlievert, Edward Kaplan: Severe Group A Streptococcal Infections Associated with a Toxic Shock-like Syndrome and Scarlet Fever Toxin A. In: New England Journal of Medicine. 321, Nr. 1, 6. Juli 1989, S. 1–7. doi:10.1056/NEJM198907063210101. PMID 2659990.
- Patrick L. Wagner, Matthew K. Waldor: Bacteriophage control of bacterial virulence. In: Infection and Immunity. 70, Nr. 8, 2002, S. 3985–3993. doi:10.1128/IAI.70.8.3985-3993.2002. PMID 12117903. PMC 128183 (freier Volltext).
- Lane P. Johnson, Mark Allen Tomai, Patrick M. Schlievert: Bacteriophage involvement in group a streptococcal pyogenic exotoxin a production. In: Journal of Bacteriology. 166, Nr. 2, 1986, S. 623–627. doi:10.1128/jb.166.2.623-627.1986. PMID 3009415. PMC 214650 (freier Volltext).
- W. Michael McShan, Joseph J. Ferretti: Genetic diversity in temperate bacteriophages of Streptococcus pyogenes: identification of a second attachment site for phages carrying the erythrogenic toxin A gene.. In: Journal of Bacteriology. 179, Nr. 20, Oktober 1997, S. 6509–6511. doi:10.1128/jb.179.20.6509-6511.1997. PMID 9335304. PMC 179571 (freier Volltext).
- J. Cantacuzène, O. Boncieu: Modifications subies par des streptococques d’origine non-scarlatineuse qu contact des produits scarlatineux filtrès. Comptes rendus de l'Académie des Sciences, 182: S. 1185–1187 (1926) (französisch).
- Joseph Ferretti, S. Kay Nida: Phage Influence on the Synthesis of Extracellular Toxins in Group A Streptococci. In: Infection and Immunity. 36, Nr. 2, Mai 1982, S. 745–750. PMID 7044976. PMC 351293 (freier Volltext).
- Claudia R. Weeks, Joseph J. Ferretti: The gene for type a streptococcal exotoxin (erythrogenic toxin) is located in bacteriophage T12. In: Infection and Immunity. 46, Nr. 2, 1984, S. 531–536. PMID 6389348. PMC 261567 (freier Volltext).
- Kurt Ferdinand Bingel: Neue Untersuchungen zur Scharlachätiologie. Deutsche medizinische Wochenschrift, 74, 1949, 703–706.
- John B. Zabriskie: The Role of Temperate Bacteriophage in the Production of Erythrogenic Toxin by Group a Streptococci. In: The Journal of Experimental Medicine. 119, Nr. 5, 1. Mai 1964, S. 761–780. doi:10.1084/jem.119.5.761. PMID 14157029. PMC 2137738 (freier Volltext).
- Lane P. Johnson, Patrick M. Schlievert: A physical map of the group A streptococcal pyrogenic exotoxin bacteriophage T12 genome.. In: Molecular & General Genetics : MGG. 189, Nr. 2, 1983, S. 251–255. doi:10.1007/BF00337813. PMID 6304466.
- Lane P. Johnson, Patrick M. Schlievert, Dennis W. Watson: Transfer of group A streptococcal pyrogenic exotoxin production to nontoxigenic strains of lysogenic conversion.. In: Infection and Immunity. 28, Nr. 1, April 1980, S. 254–7. PMID 6991440. PMC 550920 (freier Volltext).
- NCBI: Streptococcus phage A25 (species)
- NCBI: Streptococcus pyogenes phage H4489A (species)
- Larry McKane, Joseph J. Ferretti: Phage-host interactions and the production of type A streptococcal exotoxin in group A streptococci. In: Infection and Immunity. 34, Nr. 3, Dezember 1981, S. 915–919. PMID 7037644. PMC 350956 (freier Volltext). Im PDF ist T12cp1 von OCR als T12cpl missinterpretiert.
- Lane P. Johnson, Patrick M. Schlievert: Group a streptococcal phage T12 carries the structural gene for pyrogenic exotoxin type A. In: Molecular & General Genetics. 194, Nr. 1–2, 1984, S. 52–56. doi:10.1007/BF00383496. PMID 6374381.
- Martin E. Mulligan: Biochemistry 3107 - Fall 2001: Site-Specific Recombination (Memento vom 9. März 2011 im Internet Archive)
- James M. Musser, Alan R. Hauser, Michael H. Kim, Patrick M. Schlievert, Kimberlyn Nelson, Robert K. Selander: Streptococcus pyogenes causing toxic-shock-like syndrome and other invasive diseases: clonal diversity and pyrogenic exotoxin expression. In: Proceedings of the National Academy of Sciences of the United States of America. 88, Nr. 7, 1. April 1991, S. 2668–2672. bibcode:1991PNAS...88.2668M. doi:10.1073/pnas.88.7.2668. PMID 1672766. PMC 51299 (freier Volltext).
- Scarlet fever.
- Dennis L. Stevens: Streptococcus pyogenes (Group A β-hemolytic Streptococcus). Archiviert vom Original am 15. Dezember 2012.
- Streptococcal Infections (S. pyogenes — Group A streptococci).
- Streptococcal Infections (Invasive Group A Strep). Archiviert vom Original am 6. November 2012.
- S. P. Hackett, D. L. Stevens: Streptococcal toxic shock syndrome: synthesis of tumor necrosis factor and interleukin-1 by monocytes stimulated with pyrogenic exotoxin A and streptolysin O.. In: The Journal of Infectious Diseases. 165, Nr. 5, Mai 1992, S. 879–885. doi:10.1093/infdis/165.5.879. PMID 1569337.
- Dennis L. Stevens: Streptococcal toxic shock syndrome associated with necrotizing fasciitis.. In: Annual Review of Medicine. 51, 2000, S. 271–288. doi:10.1146/annurev.med.51.1.271. PMID 10774464.
- Marie Panec: Plaque Assay Protocols. In: Microbe Library. American Society for Microbiology. Archiviert vom Original am 30. November 12. Abgerufen am 28. November 2012.
- Maria Ester Ramírez Vázquez, Xavier Carbonell, Antonio P. Villaverde Corrales: Phage spread dynamics in clonal bacterial populations is depending on features of the founder cell. In: Microbiological Research. 156, Nr. 1, 2001, S. 35–40. doi:10.1078/0944-5013-00087. PMID 11372651.
- S. Atsumi, J. W. Little: Role of the lytic repressor in prophage induction of phage lambda as analyzed by a module-replacement approach. In: Proceedings of the National Academy of Sciences of the United States of America. 103, Nr. 12, 2006, S. 4558–4563. bibcode:2006PNAS..103.4558A. doi:10.1073/pnas.0511117103. PMID 16537413. PMC 1450210 (freier Volltext).
- S. C. Goshorn, P. M. Schlievert: Bacteriophage association of streptococcal pyrogenic exotoxin type C, in: J Bacteriol. 171(6), Juni 1989, S. 3068–3073, doi:10.1128/jb.171.6.3068-3073.1989, PMC 210016 (freier Volltext), PMID 2566595
- SIB: Viral exotoxin, auf: ViralZone