R-Loop
Ein R-Loop ist eine dreisträngige Nukleinsäure-Struktur, die aus einem DNA:RNA-Hybrid und der zugehörigen einzelsträngigen, nicht übersetzten DNA besteht. R-Loops können unter verschiedenen Umständen gebildet werden und von zellulären Komponenten toleriert oder freigegeben werden. Der Begriff "R-Loop" wurde verwendet, um die Ähnlichkeit dieser Strukturen mit D-Loops widerzuspiegeln; das "R" stellt in diesem Fall die Beteiligung einer RNA-Einheit dar.
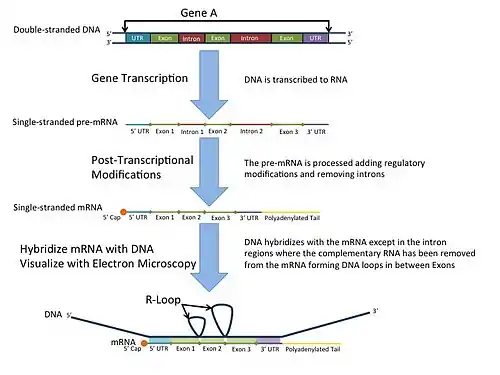
Im Labor können R-Loops auch durch Hybridisierung reifer mRNA mit doppelsträngiger DNA unter Bedingungen erzeugt werden, die die Bildung eines DNA-RNA-Hybrids begünstigen; in diesem Fall bilden die Intronbereiche (die aus der mRNA herausgespleißt wurden) einzelsträngige Schleifen, da sie nicht mit komplementärer Sequenz in der mRNA hybridisieren können.
Geschichte
Der R-Loop-Mechanismus wurde erstmals 1976 beschrieben.[1] Unabhängige Studien von R-Loops der Laboren von Richard J. Roberts und Phillip A. Sharp zeigten, dass ein Protein, das Adenovirus-Gene kodiert, DNA-Sequenzen enthielt, die in der reifen mRNA nicht vorhanden waren.[2][3] Roberts und Sharp erhielten 1993 den Nobelpreis für die unabhängige Entdeckung von Introns. Nach ihrer Entdeckung im Adenovirus wurden Introns in einer Reihe von eukaryontischen Genen gefunden, wie dem eukaryontischen Ovalbumin-Gen (zuerst vom O'Malley-Labor, dann von anderen Gruppen bestätigt),[4][5] Hexon-DNA[6] und extrachromosomalen rRNA-Genen von Tetrahymena thermophila.[6]
Mitte der 80er Jahre öffnete die Entwicklung eines Antikörpers, der spezifisch an die R-Loop-Struktur bindet, die Tür für Immunfluoreszenzstudien sowie die genomweite Charakterisierung der R-Schleifenbildung durch DRIP-Seq.[7]
R-loop Mapping
R-Loop-Mapping ist eine Labortechnik zur Unterscheidung von Introns und Exons in doppelsträngiger DNA.[8] Diese R-Loops werden elektronenmikroskopisch visualisiert und zeigen Intronbereiche der DNA, indem sie ungebundene Schleifen an diesen Bereichen erzeugen.[9]
R-loops in vivo
Das Potenzial von R-Loops als Primer für die Replikation wurde 1980 demonstriert.[10] 1994 wurde nachgewiesen, dass R-Loops in vivo vorhanden sind. Hierbei wurden Plasmide analysiert, die aus E. coli-Mutanten isoliert wurden, die Mutationen in der Topoisomerase[11] tragen. Diese Entdeckung endogener R-Loops in Verbindung mit den rasanten Fortschritten in der genetischen Sequenzierungstechnologie hat in den frühen 2000er Jahren zu einer Blütezeit der R-Loop-Forschung geführt, die bis heute anhält.[12]
Regelung der R-Loop-Bildung und -auflösung
RNase-H-Enzyme sind die wichtigsten Proteine, die für die Auflösung von R-Loops verantwortlich sind und den RNA-Teil abbauen, um die beiden komplementären DNA-Stränge hybridisieren zu lassen.[13] Die Forschung in den letzten zehn Jahren hat mehr als 50 Proteine identifiziert, die die Akkumulation von R-Loops zu beeinflussen scheinen. Während viele von ihnen vermutlich dazu beitragen, indem sie neu transkribierte RNA sequestrieren oder verarbeiten, um ein erneutes Hybridisieren an der DNA-Vorlage zu verhindern, müssen die Mechanismen der R-Loop-Interaktion für viele dieser Proteine noch bestimmt werden.[14]
Rolle von R-Loops bei der genetischen Regulation
Die Bildung von R-Loops ist ein wichtiger Schritt beim Wechsel der Immunglobulinklasse, einem Prozess, der es aktivierten B-Zellen ermöglicht, die Antikörperproduktion zu modulieren.[15] Sie scheinen auch eine Rolle beim Schutz einiger aktiver Promotoren vor Methylierung zu spielen.[16] Das Vorhandensein von R-Loops kann auch die Transkription hemmen.[17] Zusätzlich scheint die R-Loops mit dem "offenen" Chromatin verbunden zu sein, das für aktiv transkribierte Regionen charakteristisch ist.[18][19]
Genetische Schädigung durch R-Loops
Wenn sich R-Loops ungeplant bilden, können sie durch eine Reihe von verschiedenen Mechanismen Schaden anrichten.[20] Exponierte einzelsträngige DNA kann von endogenen Mutagenen angegriffen werden, einschließlich DNA-modifizierender Enzyme wie aktivierungsinduzierter Cytidin-Deaminase. Sie kann auch Replikationsgabeln blockieren, um deren Kollaps und nachfolgende Doppelstrangbrüche zu induzieren.[21] Außerdem können R-Loops eine ungeplante Replikation induzieren, indem sie als Primer fungieren.[22][19]
Die Akkumulation von R-Loops wurde mit einer Reihe von Krankheiten in Verbindung gebracht, darunter amyotrophe Lateralsklerose Typ 4 (ALS4), okulomotorische Apraxie Typ 2 (AOA2), Aicardi-Goutières-Syndrom, Angelman-Syndrom, Prader-Willi-Syndrom und Krebs.[12]
R-Loops, Introns und Beschädigungen der DNA
Introns sind nicht-kodierende Bereiche innerhalb von Genen, die zusammen mit den kodierenden Bereichen von Genen transkribiert werden, aber anschließend durch Spleißen aus dem primären RNA-Transkript entfernt werden. Aktiv transkribierte DNA-Bereiche bilden oft R-Loops, die für DNA-Schäden anfällig sind. Introns reduzieren die Bildung von R-Loops und DNA-Schäden in hochexprimierten Hefegenen.[23] Eine genomweite Analyse zeigte, dass intronhaltige Gene im Vergleich zu intronlosen Genen mit ähnlicher Expression in Hefe und Mensch einen verminderten R-Loop-Pegel und eine geringere DNA-Schädigung aufweisen.[23] Das Einsetzen eines Introns innerhalb eines für R-Loops anfälligen Gens kann auch die Bildung und Rekombination von R-Loops unterdrücken.[23] Bonnet et al. (2017) spekulierten, dass die Funktion von Introns bei der Aufrechterhaltung der genetischen Stabilität ihre evolutionäre Aufrechterhaltung an bestimmten Stellen erklären könnte, insbesondere bei hochexprimierten Genen.
Einzelnachweise
- Hybridization of RNA to double-stranded DNA: formation of R-loops. In: Proceedings of the National Academy of Sciences of the United States of America. 73, Nr. 7, July 1976, S. 2294–8. bibcode:1976PNAS...73.2294T. doi:10.1073/pnas.73.7.2294. PMID 781674. PMC 430535 (freier Volltext).
- Susan M. Berget, Claire Moore, Phillip A. Sharp: Spliced segments at the 5′ terminus of adenovirus 2 late mRNA. In: Proceedings of the National Academy of Sciences. Band 74, Nr. 8, August 1977, ISSN 0027-8424, S. 3171–3175, doi:10.1073/pnas.74.8.3171, PMID 269380, PMC 431482 (freier Volltext) – (pnas.org [abgerufen am 22. September 2019]).
- An amazing sequence arrangement at the 5' ends of adenovirus 2 messenger RNA. In: Cell. 12, Nr. 1, September 1977, S. 1–8. doi:10.1016/0092-8674(77)90180-5. PMID 902310.
- E. C. Lai, S. L. Woo, A. Dugaiczyk, J. F. Catterall, B. W. O'Malley: The ovalbumin gene: structural sequences in native chicken DNA are not contiguous. In: Proceedings of the National Academy of Sciences. Band 75, Nr. 5, 1. Mai 1978, ISSN 0027-8424, S. 2205–2209, doi:10.1073/pnas.75.5.2205, PMID 276861, PMC 392520 (freier Volltext) – (pnas.org [abgerufen am 22. September 2019]).
- K. O'Hare, R. Breathnach, C. Benoist, P. Chambon: No more than seven interruptions in the ovalbumin gene: comparison of genomic and double-stranded cDNA sequences. In: Nucleic Acids Research. Band 7, Nr. 2, 1979, ISSN 0305-1048, S. 321–334, doi:10.1093/nar/7.2.321, PMID 493147, PMC 328020 (freier Volltext) – (oup.com [abgerufen am 22. September 2019]).
- T. R. Cech, D. C. Rio: Localization of transcribed regions on extrachromosomal ribosomal RNA genes of Tetrahymena thermophila by R-loop mapping. In: Proceedings of the National Academy of Sciences. Band 76, Nr. 10, 1. Oktober 1979, ISSN 0027-8424, S. 5051–5055, doi:10.1073/pnas.76.10.5051, PMID 291921, PMC 413077 (freier Volltext) – (pnas.org [abgerufen am 22. September 2019]).
- Sophie J. Boguslawski, Dennis E. Smith, Mary A. Michalak, Kenneth E. Mickelson, Clifford O. Yehle: Characterization of monoclonal antibody to DNA · RNA and its application to immunodetection of hybrids. In: Journal of Immunological Methods. Band 89, Nr. 1, Mai 1986, S. 123–130, doi:10.1016/0022-1759(86)90040-2 (elsevier.com [abgerufen am 22. September 2019]).
- John L. Woolford, Michael Rosbash: The use of R-looping for structural gene identification and mRNA purification. In: Nucleic Acids Research. Band 6, Nr. 7, 1979, ISSN 0305-1048, S. 2483–2497, doi:10.1093/nar/6.7.2483, PMID 379820, PMC 327867 (freier Volltext) – (oup.com [abgerufen am 22. September 2019]).
- King RC, Stansfield WD, Mulligan PK (2007). A Dictionary of Genetics. Oxford University Press 7.
- T. Itoh, J. Tomizawa: Formation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. In: Proceedings of the National Academy of Sciences. Band 77, Nr. 5, 1. Mai 1980, ISSN 0027-8424, S. 2450–2454, doi:10.1073/pnas.77.5.2450, PMID 6156450, PMC 349417 (freier Volltext) – (pnas.org [abgerufen am 22. September 2019]).
- M. Drolet, X. Bi, L. F. Liu: Hypernegative supercoiling of the DNA template during transcription elongation in vitro. In: The Journal of Biological Chemistry. Band 269, Nr. 3, 21. Januar 1994, ISSN 0021-9258, S. 2068–2074, PMID 8294458.
- Matthias Groh, Natalia Gromak: Out of Balance: R-loops in Human Disease. In: PLoS Genetics. Band 10, Nr. 9, 18. September 2014, ISSN 1553-7404, S. e1004630, doi:10.1371/journal.pgen.1004630, PMID 25233079, PMC 4169248 (freier Volltext) – (plos.org [abgerufen am 22. September 2019]).
- Susana M. Cerritelli, Robert J. Crouch: Ribonuclease H: the enzymes in eukaryotes: Ribonucleases H of eukaryotes. In: FEBS Journal. Band 276, Nr. 6, März 2009, S. 1494–1505, doi:10.1111/j.1742-4658.2009.06908.x, PMID 19228196, PMC 2746905 (freier Volltext) – (wiley.com [abgerufen am 22. September 2019]).
- Yujia A. Chan, Maria J. Aristizabal, Phoebe Y. T. Lu, Zongli Luo, Akil Hamza: Genome-Wide Profiling of Yeast DNA:RNA Hybrid Prone Sites with DRIP-Chip. In: PLoS Genetics. Band 10, Nr. 4, 17. April 2014, ISSN 1553-7404, S. e1004288, doi:10.1371/journal.pgen.1004288, PMID 24743342, PMC 3990523 (freier Volltext) – (plos.org [abgerufen am 22. September 2019]).
- D. Roy, K. Yu, M. R. Lieber: Mechanism of R-Loop Formation at Immunoglobulin Class Switch Sequences. In: Molecular and Cellular Biology. Band 28, Nr. 1, 1. Januar 2008, ISSN 0270-7306, S. 50–60, doi:10.1128/MCB.01251-07, PMID 17954560, PMC 2223306 (freier Volltext) – (asm.org [abgerufen am 22. September 2019]).
- Paul A. Ginno, Paul L. Lott, Holly C. Christensen, Ian Korf, Frédéric Chédin: R-Loop Formation Is a Distinctive Characteristic of Unmethylated Human CpG Island Promoters. In: Molecular Cell. Band 45, Nr. 6, März 2012, S. 814–825, doi:10.1016/j.molcel.2012.01.017, PMID 22387027, PMC 3319272 (freier Volltext) – (elsevier.com [abgerufen am 22. September 2019]).
- Alicia D. D'Souza, Boris P. Belotserkovskii, Philip C. Hanawalt: A novel mode for transcription inhibition mediated by PNA-induced R-loops with a model in vitro system. In: Biochimica et Biophysica Acta (BBA) - Gene Regulatory Mechanisms. Band 1861, Nr. 2, Februar 2018, S. 158–166, doi:10.1016/j.bbagrm.2017.12.008, PMID 29357316, PMC 5820110 (freier Volltext) – (elsevier.com [abgerufen am 22. September 2019]).
- Maikel Castellano-Pozo, José M. Santos-Pereira, Ana G. Rondón, Sonia Barroso, Eloisa Andújar: R Loops Are Linked to Histone H3 S10 Phosphorylation and Chromatin Condensation. In: Molecular Cell. Band 52, Nr. 4, November 2013, S. 583–590, doi:10.1016/j.molcel.2013.10.006 (elsevier.com [abgerufen am 22. September 2019]).
- Lorenzo Costantino, Douglas Koshland: The Yin and Yang of R-loop biology. In: Current Opinion in Cell Biology. Band 34, Juni 2015, S. 39–45, doi:10.1016/j.ceb.2015.04.008, PMID 25938907, PMC 4522345 (freier Volltext) – (elsevier.com [abgerufen am 22. September 2019]).
- Boris P. Belotserkovskii, Silvia Tornaletti, Alicia D. D’Souza, Philip C. Hanawalt: R-loop generation during transcription: Formation, processing and cellular outcomes. In: DNA Repair. Band 71, November 2018, S. 69–81, doi:10.1016/j.dnarep.2018.08.009, PMID 30190235, PMC 6340742 (freier Volltext) – (elsevier.com [abgerufen am 22. September 2019]).
- Julie Sollier, Karlene A. Cimprich: Breaking bad: R-loops and genome integrity. In: Trends in Cell Biology. Band 25, Nr. 9, September 2015, S. 514–522, doi:10.1016/j.tcb.2015.05.003, PMID 26045257, PMC 4554970 (freier Volltext) – (elsevier.com [abgerufen am 22. September 2019]).
- T. Itoh, J. Tomizawa: Formation of an RNA primer for initiation of replication of ColE1 DNA by ribonuclease H. In: Proceedings of the National Academy of Sciences. Band 77, Nr. 5, 1. Mai 1980, ISSN 0027-8424, S. 2450–2454, doi:10.1073/pnas.77.5.2450, PMID 6156450, PMC 349417 (freier Volltext) – (pnas.org [abgerufen am 22. September 2019]).
- Amandine Bonnet, Ana R. Grosso, Abdessamad Elkaoutari, Emeline Coleno, Adrien Presle: Introns Protect Eukaryotic Genomes from Transcription-Associated Genetic Instability. In: Molecular Cell. Band 67, Nr. 4, August 2017, S. 608–621.e6, doi:10.1016/j.molcel.2017.07.002 (elsevier.com [abgerufen am 22. September 2019]).