Agrobacterium tumefaciens
Agrobacterium tumefaciens (lat. für „Tumor machendes Ackerbakterium“; neuerdings auch bezeichnet als Rhizobium radiobacter) ist ein pflanzenpathogenes, gramnegatives Bodenbakterium. Es gehört zur Alpha-Klasse der Proteobakterien.
| Agrobacterium tumefaciens | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|

Agrobacterium tumefaciens | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Agrobacterium tumefaciens | ||||||||||||
| (Smith & Townsend 1907) Conn 1942 |
A. tumefaciens ist ein Modellorganismus und verfügt über die Fähigkeit, DNA in pflanzliche Zellen zu übertragen. Dieser Vorgang wurde erstmals durch Jozef Schell und Marc Van Montagu im Jahr 1983 beschrieben.
Pathogenität
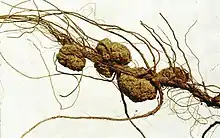 Durch A. tumefaciens hervorgerufene Gallen an den Wurzeln der Pekannuss (Carya illinoinensis).
Durch A. tumefaciens hervorgerufene Gallen an den Wurzeln der Pekannuss (Carya illinoinensis). Durch A. tumefaciens hervorgerufene Galle an einem Zweig der Forsythie (Forsythia × intermedia)
Durch A. tumefaciens hervorgerufene Galle an einem Zweig der Forsythie (Forsythia × intermedia)
Unter natürlichen Bedingungen kann das Bakterium nur verletzte, zweikeimblättrige Pflanzen infizieren. Die Infektion erfolgt meist an der Stängelbasis, es können aber auch andere oberirdische Pflanzenteile infiziert werden. Die Erkennung zwischen Bakterien- und Pflanzenzellen erfolgt mit Hilfe bestimmter Signalmoleküle (Pektine und Glucane) auf deren Oberfläche. Unter Laborbedingungen ist auch die Infektion von Monokotylen möglich.
Die Wucherungen, die A. tumefaciens durch Eingriff in den pflanzlichen Hormonhaushalt auslöst, haben bei Nutzpflanzen oft bestimmte Namen. Beim Befall der Weinrebe spricht man von Maukekrankheit, bei der Zuckerrübe von Wurzelkropf. Bei anderen Pflanzen wie etwa Meerrettich und Rhabarber werden die Tumoren auch als crown-galls (Wurzelhalsgallentumor) bezeichnet.
Ablauf der Infektion
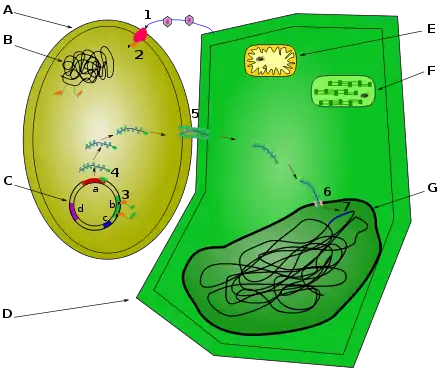
Pathogene Agrobakterien tragen stets ein Plasmid. Dieses Ti-Plasmid (Ti steht für tumor inducing – Tumor induzierend) enthält Gene, die für die spätere DNA-Übertragung in die Pflanzenzelle notwendig sind. Die Transkription dieser sogenannten „Virulenz-“ oder vir-Gene wird durch sekundäre Pflanzenstoffe (1), die aus verwundeten Pflanzenteilen austreten, aktiviert (2). Man spricht auf Seiten des Bakteriums auch von positiver Chemotaxis, da sich die Bakterien gezielt zur Quelle des Botenstoffs hin bewegen.
Mögliche Pflanzenstoffe, die Agrobacterium zu einer Infektion veranlassen:
- Acetosyringon
- Vanillin
- alpha-Hydroxyacetosyringon
- Sinapinsäure
- Syringasäure
- Ferulasäure
- Katechol
- 4-Hydroxybenzoesäure[1]
Das aktivierte vir-Genom des Bakteriums (3) befindet sich auf seinem Ti-Plasmid und codiert für Proteine (4), die in die Pflanze eindringen (5) und in der Pflanze Fremdgene installieren können. Mit Hilfe der vir-Genprodukte kann ein anderer Teil der DNA des Ti-Plasmides als so genannte „Transfer- oder t-DNA“ in die Pflanzenzelle übertragen werden. Dabei wird die t-DNA als einzelsträngiges DNA-Molekül mit Hilfe des bakteriellen Typ IV Sekretionssystems in die pflanzliche Wirtszelle eingeschleust. Diese t-DNA ist an ihrem 5'-Ende kovalent an das bakterielle VirD2-Protein gebunden, welches durch ein bestimmtes Motiv dazu beiträgt, dass die DNA in den pflanzlichen Zellkern transportiert wird (6, Kernlokalisierungssignal). Des Weiteren wird die t-DNA von VirE2-Proteinen gebunden und bildet nun den t-Komplex. Aus Arabidopsis thaliana ist bekannt, dass VirE2 mit dem pflanzlichen VIP1-Protein oder seinen homologen interagieren kann, nachdem dieses durch die pflanzliche MAP-Kinase 3 phosphoryliert wurde. Das aktivierte VIP1-Protein fungiert nun als Adaptor der pflanzlichen Kernimportmaschinerie (insbesondere Importin alpha) und dem bakteriellen VirE2-Protein und trägt nun ebenfalls zum Kerntransport des t-Komplexes bei. Im Zellkern muss der t-Strang von den VirE2 und VIP1-Proteinen befreit werden. Dies geschieht durch das bakterielle VirF-Protein, welches die Proteine dem Proteasom-vermittelten Abbau zuführt. Nun kann die t-DNA ins Wirtsgenom integrieren. In eukaryotischen Zellen ist DNA nur hier vor Abbau geschützt und kann repliziert und transkribiert werden.
Im Zellkern angekommen wird die DNA in das pflanzliche Genom integriert (7). Der Ort der Insertion im Wirtsgenom ist rein zufällig. Promotoren, die von der Pflanzenzelle erkannt werden, sorgen dafür, dass die Gene in der Pflanze auch aktiv sind. Die übertragenen Gene besitzen also eine typisch eukaryotische Struktur, obwohl sie aus einem Bakterium stammen. Die übertragenen Gene lösen Tumoren aus und veranlassen die pflanzliche Zelle, Opine (Octopin und Nopalin) zu produzieren, die den Bakterien als Nahrung dienen, für die Pflanze aber wertlos sind. Der Name Agrobacterium tumefaciens leitet sich von der tumorinduzierenden Eigenschaft ab.
Nutzen als Vektor
Die Aktivierung der vir-Gene kann dazu benutzt werden, gewünschte Gene in Pflanzen einzubringen, so genannte Transformation durch Agrobakterien. Agrobakterien können Pflanzenzellen also gezielt genetisch verändern. Wegen dieser Eigenschaft gilt es seit Beginn der Gentechnik als wichtiger Vektor, um Gene auf Pflanzen zu übertragen. Unter Laborbedingungen gelingt dies bei vielen Pflanzen und sogar einigen Pilzen. Hierfür werden die Gene der t-DNA, die normalerweise für den Opin-Metabolismus und die Tumor-Induktion notwendig sind, durch andere ersetzt. Transformationsziele sind neben zweikeimblättrigen Pflanzen wie Kartoffeln, Raps, Soja und Tabak auch einkeimblättrige Pflanzen aus der Familie der Süßgräser. In dieser Familie stehen die meisten der wichtigsten Getreidepflanzen, wie Mais, Weizen, Roggen, Reis und Gerste. Erfolgreiche Transformationen wurden für alle der genannten Monokotylen beschrieben.[2][3][4][5][6][7][8][9][10]
Genom
Das Genom von A. tumefaciens wurde 2001 sequenziert.
Ein weiterer bekannter Vertreter der Gattung ist Agrobacterium rhizogenes, der Pflanzen hauptsächlich an den Wurzeln infiziert und zu massiven Wucherungen der Wurzelhaare führt. Das Äquivalent des Ti-Plasmides ist hier das Ri-Plasmid.
Einzelnachweise
- U.S. Patent 6483013
- Cheng, M., J. E. Fry, et al. (1997). "Genetic transformation of wheat mediated by Agrobacterium tumefaciens." Plant Physiology 115(3): 971–980.
- Frame, B. R., H. X. Shou, et al. (2002). "Agrobacterium tumefaciens-mediated transformation of maize embryos using a standard binary vector system." Plant Physiology 129(1): 13–22.
- Ishida, Y., Y. Hiei, et al. (2007). "Agrobacterium-mediated transformation of maize." Nature Protocols 2(7): 1614–1621.
- Ishida, Y., H. Saito, et al. (1996). "High efficiency transformation of maize (Zea mays L) mediated by Agrobacterium tumefaciens." Nature Biotechnology 14(6): 745–750.
- Mooney, P. A., P. B. Goodwin, et al. (1991). "AGROBACTERIUM-TUMEFACIENS-GENE TRANSFER INTO WHEAT TISSUES." Plant Cell Tissue and Organ Culture 25(3): 209–218.
- Popelka, J. C. and F. Altpeter (2003). "Agrobacterium tumefaciens-mediated genetic transformation of rye (Secale cereale L.)." Molecular Breeding 11(3): 203–211.
- Raineri, D. M., P. Bottino, et al. (1990). "AGROBACTERIUM-MEDIATED TRANSFORMATION OF RICE (ORYZA-SATIVA-L)." Bio-Technology 8(1): 33–38.
- Sidorov, V., L. Gilbertson, et al. (2006). "Agrobacterium-mediated transformation of seedling-derived maize callus." Plant Cell Reports 25(4): 320–328.
- Tingay, S., D. McElroy, et al. (1997). "Agrobacterium tumefaciens-mediated barley transformation." Plant Journal 11(6): 1369–1376.