Bacillus-Virus Phi29
Bacillus-Virus Phi29 (wissenschaftlich Bacillus virus phi29, veraltet Bacillus phage phi29, Φ29) ist eine Spezies von Viren in der Gattung Salasvirus (früher Phi29virus) in der Unterfamilie Picovirinae. Diese gehört seit 2021 zur neu eingerichteten Familie Salasmaviridae der Ordnung Caudovirales (vorher zur Familie Podoviridae derselben Ordnung).
| Bacillus-Virus Phi29 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
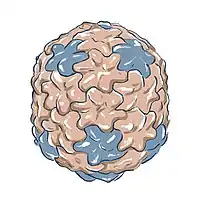
Eine Illustration des Kopfes von Bacillus-Phage Φ29 basierend auf EM-Daten | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| Bacillus virus phi29 | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| Φ29 | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
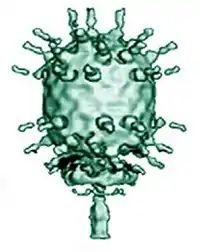
Das Genom von Bacillus-Virus Phi29 besteht aus doppelsträngiger DNA (dsDNA). Das DNA-Molekül liegt in linearer Form vor, ist also nicht kreisförmig geschlossen.
Die natürlichen Wirte sind Bakterien der Gattung Bacillus (Bazillen), daher werden diese Viren als Bakteriophagen (oder kurz Phagen) klassifiziert.
Zur selben Spezies gehört neben dem Referenzstamm Bacillus phage phi-29 auch der Bacillus phage phi-15,[2] zur selben Gattung gehört auch die Spezies Bacillus-Virus PZA und die Kandidaten Bacillus phage BS32, Bacillus phage M2 und Bacillus phage M2Y. Zur Schwestergattung Beecentumtrevirus gehören die Spezies Bacillus-Virus B103 und Bacillus-Phage Nf. Etwas entfernter verwandt ist die Spezies Bacillus-Virus GA1 (Unterfamilie Tatarstanvirinae).[3][4][1] Die Φ29-Phagen sind die kleinsten bisher isolierten Bacillus-Phagen und gehören zu den kleinsten bekannten dsDNA-Phagen.[5][6]
Aufbau
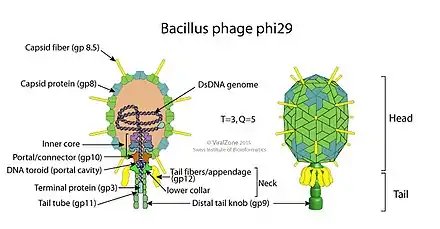
Die Virionen (Virusteilchen) von Φ29 haben eine Kopf-Schwanz-Struktur mit einem ikosaedrischen „Kopf“ und einem kurzen „Schwanz“, durch den nach der Infektion das viruseigene Erbmaterial (die lineare dsDNA) ins Zellinnere des Wirtsbakteriums gelangt. Der Kopf (Kapsid) hat eine leicht längliche Form von 45 × 54 nm, er hat eine Symmetrie mit Triangulationszahl T=3, Q=5. Der Schwanz ist nicht kontraktil mit einem „Kragen“ aus 25 Anhängseln in der Nackenregion. Am Kopf befinden sich 25 Fibrillen, die aber in manchen Laborstämmen verloren gegangen sind. Diese Strukturen dienen dazu, die Wirtszelle zuerkennen und sich an ihr anzuheften.[7]
Genom
Das Genom besteht wie allgemein in der Gattung Salsavirus aus einem einzigen linearen Stück (Segment) doppelsträngiger DNA mit einer Länge von 16 bis 20 kbp und kodiert etwa 20 bis 30 Gene.[7]
Vermehrungszyklus
Der folgende Ablauf folgt im Wesentlichen der Beschreibung auf ViralZone:[7]
- Zunächst lagert such der Phage mit Hilfe der Schwanzfibrillen an das Wirtsbakterium an.
- Danach wird die viruseigene DNA durch den Schwanz in das Zellplasma der Wirtszelle injiziert.
- Anschließend erfolgt Transkription (Ablesen der DNA und Erzeugen einer Messenger-RNA — mRNA) und Translation (Erzeugen eines Proteins aus der mRNA) einiger „früher“ Gene des Virus.
- Es folgt die Replikation des DNA-Genoms per Strangversetzung (englisch strand displacement)[8] durch die virale DNA-Polymerase (φ29-DNA-Polymerase).[9]
- Die Transkription und Translation von „späten“ Genen steht in der Schlussphase der Genexpression.
- In der darauf folgenden Assemblierungsphase werden zunächst die „Procapside“ zusammengesetzt. Dies sind die noch leeren, nicht mit Nukleinsäure — DNA oder RNA — bzw. Nukleoprotein gefüllten Kapside der
- Das Genom wird verpackt (englisch viral genome packaging), Details dazu siehe unten.
- Das Anheften des Schwanzes an die Kapside schließt die Assemblierung ab.
- Die reifen Virionen werden schließlich per Lyse (Platzen und Tod der Wirtszelle) freigesetzt.
Systematik
Äußere Systematik
- Unterfamilie Picovirinae
- Gattung Salasvirus (veraltet Phi29virus, Phi29likevirus, Phi29-like viruses)[6]
- Spezies Bacillus virus phi29 (Φ29, Typusspezies), Stämme siehe unten
- Spezies Bacillus virus B103 (B103)[10] mit den Stämmen Bacillus phage Nf (Nf) und Bacillus phage vB_BsuP-Goe1
- Spezies Bacillus virus GA1 (GA-1)[11]
- Spezies Kurthia virus 6 (veraltet Kurthia phage 6, K6, parasitiert Bakterien der Gattung Kurthia)[12]
- Bisher noch nicht vom ICTV bestätigte Kandidaten nach NCBI (Stand 3. Februar 2021):[13]
- Spezies Bacillus phage BS32 (BS32)
- Spezies Bacillus phage BSTP4 (BSTP4)
- Spezies Bacillus phage Gxv1 (Gxv1)
- Spezies Bacillus phage Karezi
- Spezies Bacillus phage M2 (M2)
- Spezies Bacillus phage M2Y (M2Y)
- Spezies Bacillus phage SF5 (SF5)
- Spezies Bacillus phage vB BveP-Goe6
- Spezies Bacillus virus SRT01hs
Synonyme
Inoffizielle Synonyme für die Spezies sind nach NCBI:[14]
- Bacillus phage phi-29
- Bacillus phage phi29
- Bacillus subtilis phage phi29
- Bacteriophage phi-29
- Phage phi-29
Bakteriophagen-pRNA
Während der Replikation wird wie bei anderen linearen dsDNA-Viren das virale Genom in das zuvor gebildete virale Procapsid verpackt. Die genauen Vorgänge wurden beispielhaft am Phagen Φ29 erforscht:
Die Verpackung von DNA in das Procapsid erfordert einen molekularen Motor, der Adenosintriphosphat (ATP) als Energie verwendet, um die eigentlich energetisch ungünstige Bewegung zu bewerkstelligen. Wie bei einigen anderen Bakteriophagen auch ist ein wesentlicher Bestandteil dieses Motors ein RNA-Molekül, das als pRNA (englisch packaging RNA) bezeichnet wird. Es handelt sich dabei um eine spezielle Form nicht-kodierender RNA (englisch non-coding RNA, ncRNA), da diese nicht wie sonst Proteine kodiert.[16] Strukturanalysen dieses Verpackungsmotors haben gezeigt, dass das pRNA-Molekül eine fünffache Symmetrie aufweist, wenn es an das Procapsid gebunden ist.[17][18][19] Es wird angenommen, dass die pRNA durch das Kapsid-Konnektor-Protein (englisch capsid connector protein) gebunden ist.[19]
Nur die ersten 120 Basen der pRNA sind für die Verpackung der viralen DNA essentiell.[20][21] Vermutlich besteht die pRNA aus zwei Abschnitten (Domänen), von denen der eine den ersten 120 Basen und der zweite den verbleibenden 50 Basen entspricht.[21]
Verwendung
Die φ29-DNA-Polymerase wird zu verschiedenen Methoden der isothermen DNA-Amplifikation wie dem Gibson Assembly und der Multidisplacement Amplification verwendet.
Einzelnachweise
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- NCBI: Phage phi-15 (no rank)
- S. Mc Grath, D. van Sinderen (Hrsg.): Bacteriophage: Genetics and Molecular Biology, 1. Auflage, Caister Academic Press, 2007, ISBN 978-1-904455-14-1.
- Ana Camacho, Fernando Jimenez, Javier Torre, Jose L. Carrascosa, Rafael P. Mellado, Eladio Vinuela, Margarita Salas, Cesar Vasquez: Assembly of Bacillus subtilis Phage Phi29. 1. Mutants in the Cistrons Coding for the Structural Proteins. In: European Journal of Biochemistry. 73, Nr. 1, Februar 1977, S. 39–55. doi:10.1111/j.1432-1033.1977.tb11290.x.
- W. J. J. Meijer, J. A. Horcajadas, M. Salas: phi29 Family of Phages. In: Microbiology and Molecular Biology Reviews. 65, Nr. 2, 1. Juni 2001, S. 261–287. doi:10.1128/MMBR.65.2.261-287.2001. PMID 11381102. PMC 99027 (freier Volltext).
- ICTV: ICTV Master Species List 2018b.v2
- SIB: Salasvirus, auf: ExPASy ViralZone
- Siehe φ29-DNA-Polymerase#Eigenschaften, Isotherme DNA-Amplifikation#Eigenschaften
- EMBL-EBI: PDBe > 2pyj – Phi29 DNA polymerase complexed with primer-template DNA and incoming nucleotide substrates (ternary complex), auf: Protein Data Bank in Europe, 2012
- ICTV: ICTV Taxonomy history: Bacillus virus B103
- ICTV: ICTV Taxonomy history: Bacillus virus GA1
- ICTV: ICTV Taxonomy history: Kurthia virus 6
- NCBI: unclassified Salasvirus
- NVBI: Salasvirus (genus)
- NCBI: Bacillus virus phi29 (species)
- P. Guo: Structure and function of phi29 hexameric RNA that drives the viral DNA packaging motor: Prog. Nucleic Acid Res. Mol. Biol. (Review) (= Progress in Nucleic Acid Research and Molecular Biology), Band 72 2002, ISBN 978-0-12-540072-5, S. 415–472, doi:10.1016/S0079-6603(02)72076-X. PMID 12206459
- M. C. Morais, Y. Tao, N. H. Olson et al.: Cryoelectron-microscopy image reconstruction of symmetry mismatches in bacteriophage phi29. In: J. Struct. Biol.. 135, Nr. 1, 2001, S. 38–46. doi:10.1006/jsbi.2001.4379. PMID 11562164. PMC 5595366 (freier Volltext).
- A. A. Simpson, Y. Tao, P. G. Leiman et al.: Structure of the bacteriophage phi29 DNA packaging motor. In: Nature. 408, Nr. 6813, 2000, S. 745–750. bibcode:2000Natur.408..745S. doi:10.1038/35047129. PMID 11130079. PMC 4151180 (freier Volltext).
- M. O. Badasso, P. G. Leiman, Y. Tao et al.: Purification, crystallization and initial X-ray analysis of the head-tail connector of bacteriophage phi29. In: Acta Crystallogr. D. 56, Nr. Pt 9, 2000, S. 1187–1190. doi:10.1107/S0907444900009239. PMID 10957642.
- P. X. Guo, S. Erickson, D. Anderson: A small viral RNA is required for in vitro packaging of bacteriophage phi 29 DNA. In: Science. 236, Nr. 4802, 1987, S. 690–694. bibcode:1987Sci...236..690G. doi:10.1126/science.3107124. PMID 3107124.
- S. Bailey, J. Wichitwechkarn, D. Johnson, B. E. Reilly, D. L. Anderson, J. W. Bodley: Phylogenetic analysis and secondary structure of the Bacillus subtilis bacteriophage RNA required for DNA packaging. In: J. Biol. Chem.. 265, Nr. 36, 1990, S. 22365–22370. PMID 2125049.