trp-Operon
Das Tryptophan-Operon, kurz trp-Operon, ist ein Operon, das für die intrazelluläre Synthese von Tryptophan in Bakterien wie z. B. Escherichia coli eine zentrale Rolle spielt.
Aktiv ist dieses Operon, wenn die Zelle Tryptophan selbst herstellen muss, weil diese Aminosäure nicht ausreichend im umgebenden Medium vorliegt. Bei hinreichend hoher intrazellulärer Konzentration an Tryptophan wird das Operon dagegen in der Regel durch einen spezifischen Repressor inaktiviert.
Die Strukturgene des Operons codieren für Polypeptide von Enzymen, mit deren Hilfe aus einer Vorstufe (Chorisminsäure) in mehreren Schritten die Aminosäure Tryptophan hergestellt werden kann. Der für die Proteinbiosynthese der enzymatisch wirksamen Proteine nötige Zugriff auf diese Gene wird über vorgeschaltete regulatorische Regionen im Operon geregelt, indem hier eine Transkription unterbunden oder unterbrochen wird.
trp-Operon in E. coli
Aufbau des Operons
Das trp-Operon in Escherichia coli-Bakterien besteht, ähnlich wie deren lac-Operon und andere Operons im Genom von prokaryoten Zellen, aus funktionell unterschiedlichen DNA-Abschnitten. Außer den Strukturgenen als solchen DNA-Abschnitten, deren Basensequenz die Struktur von enzymatisch wirksamen Proteinen bestimmt, wenn sie abgeschrieben (in RNA transkribiert) und anschließend übersetzt (in Aminosäurensequenz translatiert) wird, enthält das Operon noch andere DNA-Abschnitte als regulatorische Regionen, welche für die Regelung der Transkription von Bedeutung sind.[1]
Neben der Promotor-Region (P), an die ein RNA-Polymerase-Molekül für die Transkription binden kann, liegt die Operatoren-Region (O), an die ein Repressor-Protein binden und so den Transkriptionsstart verhindern kann (Repression). Im darauf folgenden untranslatierten Bereich liegt hier in der Leader-Sequenz noch eine weitere regulatorische Region vor (trpL), mit der ein begonnener Transkriptionsvorgang verzögert oder aber abgebrochen werden kann (Attenuation). Erst im Anschluss daran folgen downstream die Abschnitte der Strukturgene (trpE, trpD, trpC, trpB, trpA) mit offenen Leserahmen, die verschiedene Polypeptide für mehrere Proteine codieren. Wenn diese gemeinsam gebildet werden, können sie eine Abfolge von Reaktionsschritten der Tryptophan-Biosynthese katalysieren. Die dafür nötige polycistronische mRNA liefert das trp-Operon – sofern die Transkription gestartet werden kann und nicht abgebrochen wird.[2]
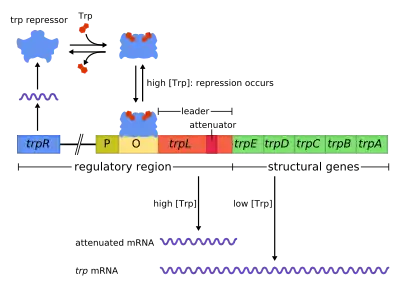
(P, O, trpL liegen als regulatorische DNA-Region vor den Strukturgenen trpE, trpD, trpC, trpB, trpA stromab)
im Zusammenhang mit zusätzlichen regulativen Elementen
(trpR-Gen für trp Repressor-Protein, das mit Tryptophan (Trp) als Corepressor aktiviert am Operator O binden kann und damit den Transkriptionsstart am Promoter P unterbindet).
Regulationsweisen
Die Transkription der Gene durch RNA-Polymerase wird in Abhängigkeit von der Tryptophan-Konzentration reguliert, vorrangig über den Operator durch Repression. Denn an den Operatorbereich kann ein blockierendes Protein als Repressor binden – doch nur dann, wenn es zuvor ein Molekül der Aminosäure Tryptophan als Liganden gebunden hat. Ist kein Tryptophan vorhanden, das diese reversible Bindung eingehen könnte, so bleibt der Repressor inaktiv und das Operon wird nicht reprimiert. Liegt dagegen hinreichend Tryptophan vor, sodass es als Cofaktor an das Protein bzw. als Corepressor an den Aporepressor gebunden wird, ist das Repressorprotein damit aktiviert und kann somit an den Operator binden, womit dann die Transkription blockiert ist.
Für die Biosynthese dieses Repressors ist eine mRNA nötig, die vom zugehörigen Regulatorgen trpR an anderer Stelle des DNA-Stranges codiert wird.
Mit vorliegendem Repressor kann die zelleigene Tryptophanbildung nun in Abhängigkeit von der Tryptophankonzentration im Zytosol selbsttätig geregelt werden, nach dem Prinzip der negativen Rückkopplung. Da hier Tryptophan auch das Produkt des regulierten Herstellungsprozesses ist, spricht man von einer Endproduktrepression: das Produkt einer Synthese wirkt selbst indirekt auf diese ein, indem es die weitere Genexpression für die beteiligten Enzyme behindert, reprimiert.
Neben dieser vorrangigen Regulationweise der Repression über den Operatorbereich, mit der ein Transkriptionsstart unterdrückt werden kann, ist für das trp-Operon eine zweite zusätzliche Regulationsweise der Attenuation bekannt, mit der eine begonnene Transkription vorzeitig abgebrochen werden kann. Hierbei spielen weitere regulatorische Sequenzen im untranslatierten Bereich (5′-UTR) eine Rolle; neben der – ein sogenanntes Leader-Peptid codierenden[3] – trpL-Sequenz sind es solche, die unterschiedliche alternative Faltungen in der entstehenden mRNA erlauben. Eine bestimmte dieser Haarnadelstrukturen stellt das sogenannte Terminationssignal der Attenuator-Sequenz dar: wird sie ausgebildet, so wird der bis dahin entstandene RNA-Strang von der RNA-Polymerase getrennt und diese von der DNA, womit die Transkription beendet ist. Dazu kommt es nicht, wenn die Konzentration an mit Tryptophan beladener tRNA (tRNATrp) sehr gering ist – also nur bei hohem Tryptophanspiegel. Auf diese Weise ist dämpfend eine feinere Abstimmung der Genexpression an die zytosolische Tryptophankonzentration möglich, und einer überhohen Tryptophansynthese wird zusätzlich entgegen gewirkt.[2]
Codierte Enzyme
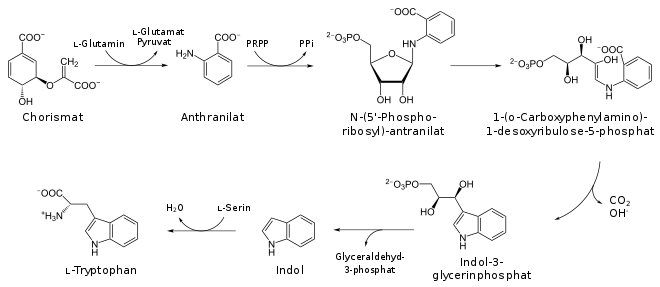
Die Strukturgene für die Tryptophan-Biosynthese im trp-Operon des Bakteriums Escherichia coli liegen stromab der Leader-Sequenz trpL und umfassen sieben Segmente trpEGDCFBA in fünf Abschnitten,
- trpE
- trpD(mit G)
- trpC(mit F)
- trpB
- trpA
wobei jedes der sieben genetischen Segmente je für eine Aminosäuresequenz im Polypetid codiert, die als Proteindomäne jeweils einen der Reaktionsschritte des Syntheseweges von Tryptophan aus Chorismat katalysiert. Chorismat wird über den Shikimisäureweg bereitgestellt.
Unter Kontrolle des trp-Operons werden seine Strukturgene durch RNA-Polymerase gemeinsam als polycistronische mRNA transkribiert und anschließend durch Ribosomen in Polypeptide translatiert. Da die Sequenzen von trpG mit trpD wie auch die von trpF mit trpC jeweils im gleichen offenen Leserahmen liegen, codieren diese beiden Abschnitte nur je für ein bifunktionelles Polypeptid, das zwei verschiedene enzymatisch wirksame Domänen enthält.
Zwischen den fünf getrennt aufgeführten Abschnitten dagegen befinden sich vier kurze intercistronische Bereiche. Hier ist jeweils neben einem Stopcodon und einem Startcodon als Ende des vorigen bzw. Anfang des nächsten Leserahmens auch die ribosomale Bindungsstelle (RBS) mit der Shine-Dalgarno-Sequenz zu finden, die ein Ribosom braucht, um am mRNA-Strang anzusetzen. Erst damit kann die codierende Nukleotidsequenz eines Leserahmens in die Aminosäuresequenz eines Polypeptids übersetzt werden.
Nach der Translation faltet ein naszierndes Polypeptid im salz-wässrigen Zellmilieu zu einem nativen Protein bestimmter Form auf, und kann mit anderen Molekülen assoziieren. Derart lagern sich hier nun oft die Proteine der Polypeptidarten trp-E und trp-D(mit G) aneinander und bilden aus vier Untereinheiten bestehende Proteinkomplexe, ebenso wie auch die von trp-B und trp-A zwei plus zwei ein Heterotetramer bilden. Diese Multienzymkomplexe entfalten ihren Untereinheiten entsprechend für unterschiedliche chemische Reaktionen des Biosyntheseweges katalytische Aktivität als
- Anthranilat-synthase (trp-G und trp-E): Chorismat → → Anthranilat
- Anthranilat-phosphoribosyl-transferase (trp-D): Anthranilat → N-(5-Phosphoribosyl)-anthranilat
- Phosphoribosyl-anthranilat-isomerase (trp-F): N-(5-Phosphoribosyl)-anthranilat → 1-(o-Carboxyphenylamino)-1-desoxyribulose-5-phosphat
- Indol-glycerolphosphat-synthase (trp-C): 1-(o-Carboxyphenylamino)-1-desoxyribulose-5-phosphat → Indol-3-glycerolphosphat
- Tryptophan-synthase (trp-A und trp-B): Indol-3-glycerolphosphat ↔ Indol → L-Tryptophan
Mit ihrer Hilfe kann aus Chorisminsäure dann L-Tryptophan aufgebaut werden. Nur einer dieser Schritte ist reversibel (↔).[2]
Einzelnachweise
- C. Yanofsky, T. Platt, I. P. Crawford, B. P. Nichols, G. E. Christie, H. Horowitz, M. VanCleemput, A. M. Wu: The complete nucleotide sequence of the tryptophan operon of Escherichia coli. In: Nucleic acids research. Band 9, Nummer 24, Dezember 1981, S. 6647–6668, PMID 7038627, PMC 327632 (freier Volltext) (Review).
- C. Yanofsky: RNA-based regulation of genes of tryptophan synthesis and degradation, in bacteria. In: RNA. Band 13, Nummer 8, August 2007, S. 1141–1154, Abbildung 2. doi:10.1261/rna.620507, PMID 17601995, PMC 1924887 (freier Volltext) (Review).
- siehe Eintrag UniProtKB – P0AD92 (LPW_ECOLI) in der bioinformatischen Datenbank für Proteine UniProt.
Literatur
- Nancy Trun, Janine Trempy: Gene expression and regulation. In: Nancy Trun, Janine Trempy: Fundamental Bacterial Genetics. Blackwell, Malden MA u. a. 2004, ISBN 0-632-04448-9, S. 191–212, online (PDF; 500 kB).