Terminator (Genetik)
Als Terminator oder Transkriptionsterminator wird jener Abschnitt einer genetischen Sequenz auf der DNA bezeichnet, der das Ende eines Gens oder Operons markiert, da er zur Beendigung (Termination) der Transkription führt.
Bei der Transkription wird auch diese terminierende Sequenz von RNA-Polymerasen in die Nukleotidsequenz des neugebildeten RNA-Strangs umgeschrieben. In dieser Form ist die Basenfolge eines Teminators auf dem RNA-Transkript dann das Signal – Terminationssignal – für jene Prozesse, die unmittelbar oder auch mithilfe zusätzlicher Faktoren – Terminationsfaktoren – dazu führen, dass der Transkriptionsvorgang beendet wird.
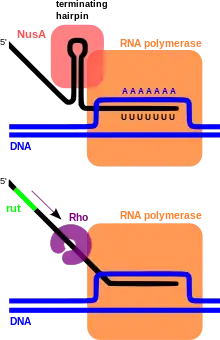
– einer intrinsischen (oben)
– einer rho-abhängigen (unten)
Termination der Transkription (schwarz dargestellt: RNA des Transkripts)
In Prokaryoten sind zwei Klassen von transkriptionalen Terminatoren bekannt:
- Intrinsische – durch unmittelbare RNA-Interaktion wirkend
- Rho-Faktor-abhängige – indirekt über Proteinfaktoren wirksam
Die DNA-Sequenz eines intrinsischen Terminators enthält zumeist kurze Folgen von (vier bis zehn) G/C-Basenpaaren und eine Folge gleicher Basen (T bzw. A). In RNA umgeschrieben finden sich diese stromab oft gleich nach einem Stopcodon, welches einen für die Translation des Transkripts offenen Leserahmen (ORF) beschließt.
Die DNA-Sequenz der längeren Erkennungsregion für den Terminationsfaktor (ρ) einer rho-abhängigen Termination enthält zumeist viele G- und wenig C-Basen; diese in RNA somit C-reiche Bindestelle (rut – rho utilization site genannt) liegt stromauf in einiger Distanz vor einem Stopcodon und der Terminationsstelle.
In Eukaryoten, wie dem Menschen, sind die Prozesse der transkriptionalen Termination weniger gut verstanden. Auch hier spielen Nukleotidsequenzen eine Rolle als Terminationssignal. Erkannt werden diese von verschiedenen Proteinen, die als Terminationsfaktor daran binden. In komplexem Zusammenspiel führen sie zum Pausieren der RNA-Synthese, zur Freisetzung des RNA-Transkripts und zur Ablösung der RNA-Polymerase von der DNA-Vorlage. Diese Teilprozesse sind in eukaryotischen Zellen oft zeitversetzt getrennte Abläufe, mit zwischengeschalteten Schritten der RNA-Prozessierung.
Als Antiterminatoren werden Proteine, RNAs oder (intrinsische) RNA-Strukturen bezeichnet, die eine Beendigung der Transkription verhindern.
Rho-unabhängige Termination (Intrinsische Termination)
Bei dieser Form der Termination kommt es zur Ausbildung einer haarnadelförmigen Sekundärstruktur im RNA-Transkript. Der Grund für diese Haarnadelstruktur liegt in der speziellen Basenfolge. Daneben liegt oft ein U-reicher Bereich vor. Für die optimale Struktur eines intrinsischen Terminators werden damit folgende Regionen im Transkript gebraucht:[1]
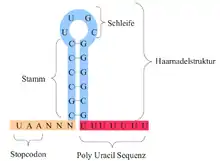
- Stammbildende GC-reiche Regionen a und c (6–8 Nukleotide)
- Schleifenbildende Region b (3–5 Nukleotide)
- Poly-Uracil Region d (3–8 Nukleotide)
Die beiden GC-reichen Regionen a und c lagern sich zum sogenannten Stamm (stem) zusammen. Die dazwischen liegende Region b formt die sogenannte Schleife (loop). Die entstandene Haarnadelstruktur (hairpin) bewirkt eine Verzögerung des Transkriptionsvorgangs, solange sie bestehen bleibt.[2] Dies wird begünstigt durch ein flexibles Protein mit besonderer Taschenbildung, NusA, das an die RNA-Polymerase gebunden ist.[3] Die RNA-Haarnadelstruktur führt darüber zu einer allosterischen Hemmung der Nukleotidaddition am aktiven Zentrum der RNA-Polymerase.[4] Mit der nachfolgenden Uracil-Reihe d verliert der transkriptionale Komplex an Stabilität.[5]
Nach der gängigen Theorie wird infolge der RNA-Haarnadel das RNA/DNA-Hybrid verkürzt und asymmetrisch verschoben; die schwache Bindung über U-A-Basenpaarungen zwischen RNA und DNA erlaubt dann jene Destabilisierung, mit der das Transkript aus dem Komplex entlassen wird.[6]
Eine rho-unabhängige Termination wird auch bei der Attenuation genutzt zur Regulation der Genexpression.
Rho-abhängige Termination
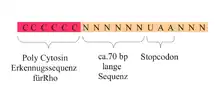
Bei der Rho-Faktor-abhängigen Termination sind keine destabilisierenden Sequenzelemente in der RNA nötig, denn die Termination wird durch den Terminationsfaktor Rho katalysiert, einen hexameren Proteinkomplex. Die Destabilisierung des transkriptionalen Komplexes ist Folge der Helikaseaktivität des Rho-Faktors.[7] Rho erkennt hierbei C-reiche (und G-arme) Abschnitte auf dem Transkript. Es bindet auf dem neusynthetisierten RNA-Strang an einen ca. 70 nt langen Bereich (rut), der stromauf der Terminationsstelle liegt. Mit ATPase-Aktivität bewegt sich Rho dann auf das 3’-Ende der RNA zu.
Zum Terminationsfaktor wird Rho durch seine Wirkung als Helikase, die zu einer Trennung des RNA/DNA-Hybrids führt. Die Folge ist eine Dissoziation des Komplexes und so die Freisetzung des RNA-Transkripts.[8]
Unterscheidung
Die Terminatorsequenzen im DNA-Doppelstrang sind nicht gleich denen auf dem RNA-Einzelstrang des Transkripts. Beide sind im Übrigen zu unterscheiden von den Stopcodons auf der mRNA, die zur Termination der Translation führen.
Literatur
- Rolf Knippers: Molekulare Genetik. 8. neubearbeitete Auflage. Georg Thieme Verlag, Stuttgart 2001, ISBN 3-13-477008-3.
Einzelnachweise
- Y. C Carafa, E. Brody, C. Thermes: Prediction of rho-independent Escherichia coli transcription terminators•:: A statistical analysis of their RNA stem-loop structures. In: Journal of molecular biology, 216(4), 1990, S. 835–858.
- I. Artsimovitch, R. Landick: Interaction of a nascent RNA structure with RNA polymerase is required for hairpin-dependent transcriptional pausing but not for transcript release. In: Genes & Development, 12(19), 1998, S. 3110–3122, doi:10.1101/gad.12.19.3110
- X. Guo, A. Myasnikov, J. Chen, C. Crucifix, G. Papai, M. Takacs, P. Schultz, A. Weixlbaumer: Structural Basis for NusA Stabilized Transcriptional Pausing. In: Molecular Cell, 69 (5), März 2018, S. 816–827.
- I. Toulokhonov, I. Artsimovitch, R. Landick: Allosteric control of RNA polymerase by a site that contacts nascent RNA hairpins. In: Science, 292(5517), 2001, S. 730–733.
- V. Brendel, G. H. Hamm, E. N. Trifonov: Terminators of transcription with RNA polymerase from Escherichia coli: what they look like and how to find them. In: Journal of biomolecular structure & dynamics, 3(4), 1986, S. 705–723, PMID 3078109
- P. H. von Hippel, T. D. Yager: Transcript elongation and termination are competitive kinetic processes. In: Proceedings of the National Academy of Sciences, 88(6), 1991, S. 2307–2311.
- K. M. Walstrom, J. M. Dozono, P. H. von Hippel: Kinetics of the RNA-DNA helicase activity of Escherichia coli transcription termination factor rho. 2. Processivity, ATP consumption, and RNA binding. In: Biochemistry, 36(26), 1997, S. 7993–8004, PMID 9201946
- D. J. Jin, R. R. Burgess, J. P. Richardson, C. A. Gross: Termination efficiency at rho-dependent terminators depends on kinetic coupling between RNA polymerase and rho. In: Proceedings of the National Academy of Sciences, 89(4), 1992, S. 1453–1457.