N-End Rule
Die N-End Rule beschreibt den Einfluss der N-terminalen Aminosäure eines Proteins auf seine Abbaugeschwindigkeit. Dieser Zusammenhang wurde erstmals 1986 von einer Arbeitsgruppe um den russisch-amerikanischen Biochemiker Alexander Varshavsky am Massachusetts Institute of Technology beschrieben.[1][2]
Eigenschaften
Die N-terminale Aminosäure hat Einfluss auf den proteolytischen Abbau eines Proteins. Bei der Proteinbiosynthese entstehen alle Proteine von Eukaryoten oder Archaeen mit einem Methionin als erster Aminosäure bzw. mit einem Formylmethionin bei Bakterien. Proteine mit einer anderen N-terminalen Aminosäure als Methionin können im Anschluss entweder durch Proteolyse entstehen oder durch Entfernung des Methionins durch Methionin-Aminopeptidasen, sofern die darauf folgende Aminosäure Valin, Glycin, Prolin, Alanin, Serin, Threonin oder Cystein ist.[3] Von diesen sieben Aminosäuren führt jedoch nur Cystein zu einem beschleunigten Abbau.[3]
Es werden zwei Typen N-terminaler Aminosäuren erkannt und einer Proteolyse zugeführt. Diese beiden Typen werden als Degrons bezeichnet. Das Typ-1-Degron umfasst basische Aminosäuren (Arginin, Lysin und Histidin) und das Typ-2-Degron besteht aus aromatischen und aliphatischen Aminosäuren (Phenylalanin, Tyrosin, Tryptophan, Leucin und Isoleucin).[3] In Bakterien erfolgt die Erkennung durch das Protein ClpS, welches ein Protein mit entsprechenden N-terminalen Aminosäuren dem Abbau durch ClpAP zuführt.[4] In Eukaryoten werden diese beiden Degron-Typen durch vier Typen von Recogninen erkannt, die ein gebundenes Protein der E3-Ubiquitin-Protein-Ligase und somit einem Abbau im Ubiquitin-Proteasom-System zuführen.[3]
Die einem Abbau vorangehende Arginylierung von N-terminalen Aspartat-, Glutamat- und Cysteinsulfonsäureresten eines Proteins erfolgt in Eukaryoten durch die Arginin-tRNA-Protein-Transferase Ate1.[3] Die Cysteinsulfonsäure ist ein oxidiertes Cystein und entsteht z. B. aufgrund der Oxidation durch Stickstoffmonoxid oder Superoxid während der Immunreaktion.[5] Asparaginsäure und Glutaminsäure können durch zwei verschiedene Amidohydrolasen zu Aspartat bzw. Glutamat desaminiert und anschließend mit Arginin versehen werden. Diese N-terminal arginylierten Proteine werden aufgrund des Typ-1-Degrons (Arginin) von den Recogninen erkannt und der Ubiquitinligase zugeführt.[3]
Die Oxidation des Cysteins zur Cysteinsulfonsäure ist ein Sensor für zellulären Stress. Durch den auf die Arginylierung folgenden Abbau des oxidierten Proteins im Proteasom werden proapoptotische Proteine und Peptide mit oxidierten Cysteinen abgebaut, die bei übermäßiger Anhäufung den Zelltod einleiten.[6] Bei Proteinen von Pathogenen des Menschen konnte ein verstärkter Abbau durch die N-terminalen Aminosäuren gezeigt werden, z. B. bei der Integrase von HIV und beim Listeriolysin O von Listeria monocytogenes.[3]
Biologische Halbwertszeiten N-terminaler Aminosäuren
Abhängig von der Spezies und der Aminosäure in N-terminaler Position besitzen diese verschiedene Halbwertszeiten:
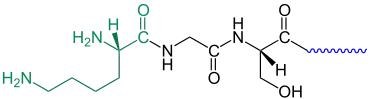
grün markierter N-terminaler α-Aminosäure L-Lysin und der blau markierten schematisch angedeuteten restlichen Proteinkette bis zur C-terminalen α-Aminosäure am Ende.

grün markierter N-terminaler α-Aminosäure L-Phenylalanin und der blau markierten schematisch angedeuteten restlichen Proteinkette bis zur C-terminalen α-Aminosäure am Ende.
| Spezies | Aminosäure | Halbwertszeit | ||
| Escherichia coli[7] |
|
| ||
| Saccharomyces cerevisiae (Backhefe)[7] |
|
| ||
| Säugetiere[8] |
|
| ||
Literatur
- J. H. Lee, Y. Jiang u. a.: Pharmacological Modulation of the N-End Rule Pathway and Its Therapeutic Implications. In: Trends in pharmacological sciences. Band 36, Nummer 11, November 2015, S. 782–797, doi:10.1016/j.tips.2015.07.004, PMID 26434644, PMC 4641009 (freier Volltext) (Review).
- A. Varshavsky: The N-end rule pathway and regulation by proteolysis. In: Protein science. Band 20, Nummer 8, August 2011, S. 1298–1345, doi:10.1002/pro.666, PMID 21633985, PMC 3189519 (freier Volltext) (Review).
- E. Graciet, F. Wellmer: The plant N-end rule pathway: structure and functions. In: Trends in plant science. Band 15, Nummer 8, August 2010, S. 447–453, doi:10.1016/j.tplants.2010.04.011, PMID 20627801 (Review).
- D. A. Dougan, K. N. Truscott, K. Zeth: The bacterial N-end rule pathway: expect the unexpected. In: Molecular microbiology. Band 76, Nummer 3, Mai 2010, S. 545–558, doi:10.1111/j.1365-2958.2010.07120.x, PMID 20374493 (Review).
- A. Varshavsky: The N-end rule and regulation of apoptosis. In: Nature cell biology. Band 5, Nummer 5, Mai 2003, S. 373–376, doi:10.1038/ncb0503-373, PMID 12724766 (Review).
- A. Varshavsky: The N-end rule: functions, mysteries, uses. In: Proceedings of the National Academy of Sciences. Band 93, Nummer 22, Oktober 1996, S. 12142–12149, PMID 8901547, PMC 37957 (freier Volltext) (Review).
- R. T. Baker, A. Varshavsky: Inhibition of the N-end rule pathway in living cells. In: PNAS. Band 88, Nummer 4, Februar 1991, S. 1090–1094, PMID 1899923, PMC 50962 (freier Volltext).
- D. K. Gonda, A. Bachmair u. a.: Universality and structure of the N-end rule. In: The Journal of biological chemistry. Band 264, Nummer 28, Oktober 1989, S. 16700–16712, PMID 2506181.
- A. Varshavsky, A. Bachmair, D. Finley: The N-end rule of selective protein turnover: mechanistic aspects and functional implications. In: Biochemical Society transactions. Band 15, Nummer 5, Oktober 1987, S. 815–816, PMID 3691950.
- Konstantin I. Piatkov, Christopher S. Brower, Alexander Varshavsky: The N-end rule pathway counteracts cell death by destroying proapoptotic protein fragments. In: Proceedings of the National Academy of Sciences. Band 109, Nr. 27, 2012, S. E1839–E1847, doi:10.1073/pnas.1207786109, PMID 22670058 (freier Volltext).
Einzelnachweise
- Andreas Bachmair, Daniel Finley, Alexander Varshavsky: In vivo half-life of a protein is a function of its amino-terminal residue. In: Science. Band 234, Nummer 4773, Oktober 1986, S. 179–186, PMID 3018930.
- Quick or slow ends for proteins. In: New Scientist. vom 30. Okt. 1986, ISSN 0262-4079, Band 112, Nr. 1532, S. 25 (eingeschränkte Vorschau in der Google-Buchsuche).
- S. M. Sriram, R. Banerjee, R. S. Kane, Y. T. Kwon: Multivalency-assisted control of intracellular signaling pathways: application for ubiquitin- dependent N-end rule pathway. In: Chem Biol. (2009), Band 16(2), S. 121–131. doi:10.1016/j.chembiol.2009.01.012. PMID 19246002; PMC 2665046 (freier Volltext).
- D. A. Dougan, K. N. Truscott, K. Zeth: The bacterial N-end rule pathway: expect the unexpected. In: Mol Microbiol. (2010), Band 76(3), S. 545–558. doi:10.1111/j.1365-2958.2010.07120.x. PMID 20374493.
- R. G. Hu, J. Sheng, X. Qi, Z. Xu, T. T. Takahashi, A. Varshavsky: The N-end rule pathway as a nitric oxide sensor controlling the levels of multiple regulators. In: Nature (2005), Band 437(7061), S. 981–986. PMID 16222293. PDF.
- K. I. Piatkov, C. S. Brower, A. Varshavsky: The N-end rule pathway counteracts cell death by destroying proapoptotic protein fragments. In: Proc Natl Acad Sci U S A (2012), Band 109(27), S. E1839-47. doi:10.1073/pnas.1207786109. PMID 22670058; PMC 3390858 (freier Volltext).
- A. Varshavsky: The N-end rule pathway of protein degradation. In: Genes Cells. (1997), Band 2(1), S. 13–28. PMID 9112437. PDF.
- D. K. Gonda et al.: Universality and Structure of the N-end Rule. In: Journal of Biological Chemistry (1989), Band 264:28, S. 16700–16712, PMID 2506181.
