Mitochondriale Eva
Die mitochondriale Eva ist ein Begriff aus der Archäogenetik und bezeichnet eine Frau, aus deren mitochondrialer DNA (mtDNA) die mitochondriale DNA aller heute lebenden Menschen durch eine direkte Abstammungslinie hervorgegangen ist. Ihr männliches Gegenstück ist der Adam des Y-Chromosoms.

Theorie
Betrachtet man verschiedene Gene (oder auch andere abgrenzbare Abschnitte des Genoms) bei verschiedenen Individuen, stellt man in der Regel fest, dass es hier bei unterschiedlichen Individuen leicht unterschiedliche Varianten gibt, die Allele genannt werden (Polymorphismus des Genoms). Wenn es sich um bei den Individuen einander entsprechende, kurz homologe Gene oder DNA-Abschnitte handelt, können diese Varianten nur dadurch entstanden sein, dass sich eine ursprüngliche Sequenz durch Mutationen nach und nach verändert hat – dies entspricht bereits der Definition für homologe Gene. Jeder (homologe) DNA-Abschnitt lässt sich also gedanklich auf eine Ursprungssequenz zurückführen, aus der sich die heutige Vielfalt nach und nach entwickelt hat. Verfolgt man diesen Vorgang im zeitlichen Ablauf, ergibt sich ein Muster aus Aufspaltungsvorgängen, die jeweils auf eine Mutation zurückgehen. Nimmt man stattdessen die heutigen Sequenzen und versucht deren Entstehung zu rekonstruieren, entspricht jede dieser Aufspaltungen im Rückblick einem „Zusammenfließen“ (englisch coalescence) der jeweiligen Sequenzen. Entsprechende Analysen werden deshalb als Koaleszenzanalysen bezeichnet.
Beim Menschen liegt nun jedes Chromosom, und damit jedes Gen, im Prinzip in zwei Kopien vor, von denen jeweils eine von der Mutter und eine vom Vater stammt (Diploidie). Ausnahmen davon sind, neben den Geschlechtschromosomen, die eigenständigen Gene der Mitochondrien – das sind Organellen, die vor allem als Energielieferanten („Kraftwerke“) der Zelle dienen. Aufgrund des besonderen Baus der Spermien stammen alle Mitochondrien, sowohl bei Männern als auch bei Frauen, aus der Eizelle und tragen somit das mütterliche Genom. Die mitochondrialen Gene jedes Individuums besitzen also alle denselben Haplotyp. Durch Koaleszenzanalyse der mitochondrialen DNA kann man rechnerisch eine DNA-Sequenz bestimmen, auf die die heutige Variationsbreite zurückgeführt werden kann. Die Trägerin dieser bestimmten Sequenz wird, etwas plakativ, als „mitochondriale Eva“ bezeichnet.
Die Existenz dieser Vorfahrin an sich ist trivial und mit keiner besonderen Erkenntnis verbunden – sie ergibt sich automatisch daraus, dass alle Menschen, ja überhaupt alle Lebewesen, letztlich miteinander verwandt sind und damit zwingend irgendwann einmal einen gemeinsamen Vorfahren gehabt haben müssen. Von der Logik her müsste die „mitochondriale Eva“ nicht einmal unserer Art angehört haben, weil schon die Artbildung auf eine Population zurückgeht, die bereits einen erheblichen Polymorphismus besessen haben könnte, so dass die gemeinsame Vorfahrin, von der die mitochondriale Sequenz herrührt, bereits einer Vorgängerart entstammen würde.[1] Schon da Polymorphismus und Mutationsrate bei unterschiedlichen Genen verschieden sind, aber auch einfach aus Zufall, ergibt sich bei Betrachtung anderer Gene auch jeweils ein anderer gemeinsamer Vorfahre, der wesentlich jünger oder älter als die „mitochondriale Eva“ sein kann. Wissenschaftlich interessant wird die Beschäftigung damit nur, wenn dieser trivialen Grundaussage weitere Erkenntnisse, beispielsweise zum Ort oder Zeitpunkt der Aufspaltung, hinzugefügt werden können.
Unter idealen Bedingungen (unendliche Populationsgröße, unbeschränkte Mischung, adaptiv gleichwertige Allele, keine Mutationen) würde jedes in der Population vorhandene Allel in völlig unveränderter Frequenz auf Dauer erhalten bleiben (Hardy-Weinberg-Gleichgewicht). In realen Populationen ist das freilich nie der Fall. Allele werden bei begrenzter Populationsgröße einfach per Zufall, durch unterschiedliche Nachkommenzahl einzelner Individuen, häufiger oder seltener werden; dieses Phänomen wird als Gendrift bezeichnet. Dadurch ist in realen Populationen die Lebensdauer von Allelen durch Gendrift auch dann begrenzt, wenn diese untereinander vollkommen gleichwertig (neutral) sind. Dabei ist leicht einzusehen, dass die Gendrift umso stärker sein muss, je kleiner die Populationsgröße ist. Der Zufallsweg (mathematisch eine Markow-Kette) führt unabhängig von der Ausgangsgröße jedes in einer Population vorhandene Allel über kurz oder lang zum Aussterben – in Abwesenheit von weiteren Mutationen solange, bis nur noch eines übrig ist (genannt „Fixierung“ des Allels). Bei haploidem Erbgang (wie beim Mitochondrium) ist die Wahrscheinlichkeit einer Koaleszenz in der vorangehenden Generation 1 geteilt durch die Populationsgröße N (eigentlich der „effektiven“ Populationsgröße, die neben der Anzahl der Individuen durch abweichende Paarungswahrscheinlichkeiten beeinflusst wird). Dadurch ergibt sich ein Erwartungswert für die Koaleszenzzeit von zweimal der Populationsgröße.[2] Bei einer Populationsgröße von 100 Individuen wäre demnach ein gemeinsamer Vorfahr vor etwa 200 Generationen zu erwarten. In realen Populationen muss allerdings unbedingt der Einfluss von Mutationen, die neue Allele hervorbringen, berücksichtigt werden.
Die mitochondriale Eva war weder die erste Frau noch die einzige Frau zu einem bestimmten Zeitpunkt der Vergangenheit. Eva hatte viele Zeitgenossinnen; die mitochondrialen Erblinien der anderen Frauen starben aber aus, während die von Eva überlebte (im größten Teil ihrer Nachkommen allerdings mit mehr oder weniger vielen Mutationen). Lebenszeit und -ort dieser Vorfahrin lassen sich mit Hilfe der Analyse der mtDNA einer repräsentativen Anzahl heute lebender Individuen recht genau eingrenzen.
Bedeutung
Eine Reihe von Eigenschaften machen die mtDNA zu einem wertvollen Werkzeug für die Erforschung der menschlichen Abstammung:
- Im Vergleich zur DNA des Zellkerns zeigt die mtDNA eine höhere und konstantere Mutationsrate.
- Da mtDNA nur von der Mutter weiter vererbt wird, ist die effektive Populationsgröße nur ¼ so groß wie bei der autosomalen DNA, bei der beide Elternteile je zwei Kopien tragen. Dementsprechend erfolgt die Fixierung von Allelen auch etwa viermal so schnell. Die Allele menschlicher mtDNA sind also wesentlich jünger als bei der autosomalen DNA und eignen sich sehr gut für die Erforschung der jungen Menschheitsgeschichte, wie zum Beispiel der Besiedlung Eurasiens.
- Die im Vergleich zur autosomalen DNA höhere Mutationsrate und Gendrift der mtDNA führen dazu, dass die Häufigkeit der Allele von einer Teilpopulation zur anderen viel stärker schwankt. Aus diesen Unterschieden können Aussagen über Abstammung, Migration, Verdrängung oder Vermischung von Populationen viel einfacher abgeleitet werden als mit der geographisch homogeneren autosomalen DNA.
- Da eine Zelle viele Mitochondrien enthält und in jeder mehrere Kopien der mtDNA vorliegen, lässt sich oft auch aus Fossilien (z. B. Neandertaler-Knochengewebe) genug mtDNA für die Analyse extrahieren, während die DNA des Zellkerns weitaus seltener ausreichend vollständig überliefert ist.
- Das Fehlen von Rekombination bei der Vererbung der mtDNA ermöglicht Aussagen über spezifische Eigenschaften der weiblichen Erblinie.
Wo und wann lebte Eva?
Erste Untersuchungen der Variation humaner mitochondrialer DNA wurden bereits 1983 durchgeführt.[3][4] Bekannt wurde die Theorie durch eine Publikation von Rebecca L. Cann, Allan Wilson und Mark Stoneking (1987).[5]
Cann et al. (1987) extrahierten mtDNA aus der Plazenta von Frauen aus unterschiedlichen Teilen der Welt. Sie sequenzierten die mtDNA nicht, sondern führten eine Untersuchung mittels Restriktionsfragmentlängenpolymorphismus (RFLP) durch. Sie ordneten die mtDNAs entsprechend ihrer Ähnlichkeit auf einem Stammbaum an und ermittelten schließlich die Wurzel des Stammbaums. Von der ermittelten Wurzel zweigten zwei Hauptäste ab: Auf dem einen fanden sich nur Afrikaner, auf dem anderen Personen aus allen Erdteilen. Daraus schlossen die Autoren, dass die mitochondriale Eva in Afrika gelebt haben muss.
Zudem versuchten sie, mit Hilfe einer molekularen Uhr zu ermitteln, wann die mitochondriale Eva gelebt hat. 1987 lagen bereits Daten über die mitochondriale DNA (mtDNA) von Fischen, Vögeln und einigen Säugetierarten vor. Aus diesen Daten ging hervor, dass mtDNA sich etwa um 2–4 % pro Million Jahren verändert. Diese Daten wurden zur Kalibrierung der molekularen Uhr verwendet. Da die menschlichen mitochondrialen DNAs in der Studie sich durchschnittlich nur um 0,57 % unterschieden, ergab sich daraus, dass die mitochondriale Eva vor nur etwa 200.000 Jahren gelebt haben muss.
Da die Fixierung einer Erblinie in einer expandierenden Population unwahrscheinlich ist – so das Argument der Autoren –, muss die mitochondriale Eva gelebt haben, bevor die Vorfahren aller heute lebenden Menschen Afrika verlassen hatten. Für die Autoren ein klarer Hinweis für die Out-of-Africa-Theorie. Die Hypothese vom „multiregionalen Ursprung des modernen Menschen“ wurde von den Autoren zurückgewiesen, da die mitochondriale Eva hierfür sehr viel älter hätte sein müssen (Homo erectus hatte Afrika vor fast 2 Millionen Jahren verlassen).
Die Publikation wurde von Anfang an stark kritisiert. Viele Kritikpunkte erschienen durchaus berechtigt:
- Von den 147 Personen in der Studie stammten nur 2 der 20 „Afrikaner“ wirklich aus Afrika südlich der Sahara; die anderen 18 waren Afroamerikaner.
- Die Methode, den Stammbaum zu generieren, lieferte nicht unbedingt den statistisch günstigsten Baum.
- Um die Wurzel des Baums zu finden, setzte man die Wurzel in die Mitte des längsten Astes (midpoint rooting). Das kann zu einer falschen Position der Wurzel führen, z. B. wenn die Evolutionsgeschwindigkeit in Afrika höher ist.
- RFLP ist wenig geeignet, um Mutationsraten zu bestimmen, was für eine Molekulare Uhr wichtig ist.
- Schlechte statistische Analyse.
Die (seriöse) Kritik war also nicht allgemein gegen das Konzept der mitochondrialen Eva gerichtet. Jedem, der mit der Materie vertraut war, war klar, dass diese Frau irgendwann und irgendwo gelebt haben muss. Nur das wissenschaftliche Vorgehen der Autoren wurde kritisiert.
Spätere, verbesserte Studien bestätigten und untermauerten jedoch die wichtigsten Aussagen von Cann et al. (1987). Zum Beispiel führten Ingman et al. (2000) eine neue, verbesserte Studie durch:
- Sie nahmen Proben von 53 Personen, 32 von ihnen aus unterschiedlichen Teilen Afrikas südlich der Sahara.
- Sie sequenzierten die kompletten mtDNAs, aber für die Analyse schlossen sie die schnell evolvierende D-Loop-Region aus.
- Die Wurzel des Stammbaums wurde mit Hilfe der mtDNA eines Schimpansen bestimmt (outgroup rooting).
Die Ergebnisse dieser verbesserten Studie waren noch eindeutiger als bei der Studie 1987:
- Vollständige Separation von Afrikanern und Nichtafrikanern.
- Die ersten drei Äste des Stammbaums führten nur zu Afrikanern, der vierte führte zu Afrikanern und Nichtafrikanern.
- Lange Äste in Afrika, aber sternförmige Struktur außerhalb (charakteristisch für kürzliche Expansion).
- 175.000 ± 50.000 Jahre bis zum gemeinsamen Vorfahren (mitochondriale Eva aller Menschen in der Studie).
- 52.000 ± 28.000 Jahre zur Verzweigung zwischen dem letzten afrikanischen und dem nichtafrikanischen Zweig (mitochondriale Eva aller Nichtafrikaner in der Studie).
- Signal für eine Expansion im nichtafrikanischen Ast vor etwa 1925 Generationen, also etwa vor 38.500 Jahren, wenn man von einer Generationszeit von 20 Jahren ausgeht.
2013 wurde schließlich in Science eine weitere Studie publiziert, der zufolge die „mitochondriale Eva“ vor 99.000 bis 148.000 Jahren lebte und der sogenannte Adam des Y-Chromosoms vor 120.000 bis 156.000 Jahren.[6]
Haplotypen
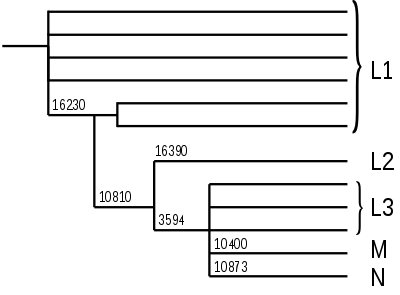
Die Zahlen geben die Position der Mutationen an.
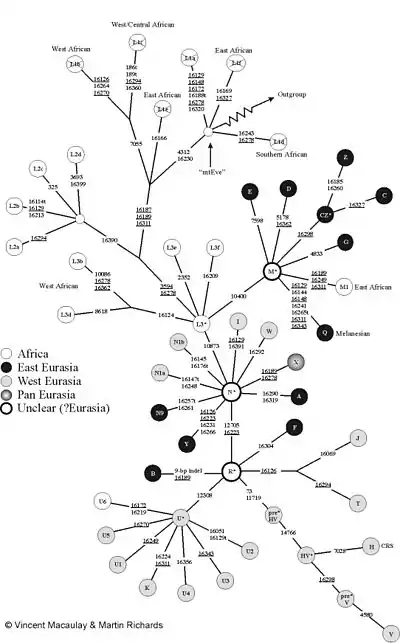
Die Zahlen geben die Position der Mutationen an.
„mtEve“ ist die mitochondriale Eva. „Outgroup“ führt zu mtDNA anderer Primaten (z. B. Schimpansen). Die Abbildung benutzt die übliche (falsche) Nomenklatur mit der „L1 Haplogruppe“: L1 bildet jedoch die Wurzel (L1a ist mit L1f nicht näher verwandt als mit V!). Daher wurden die L1-Felder durchgestrichen.[7]
Die mitochondriale DNA der Menschen kann in sogenannte Haplogruppen unterteilt werden. Eine Haplogruppe kann ihrerseits weitere Unter-Haplogruppen enthalten, die sich ihrerseits weiter unterteilen lassen. Man versucht, bei der Nomenklatur der Haplogruppen diese Baumstruktur abzubilden und verwendet abwechselnd Buchstaben und Zahlen. Zwei mtDNAs einer Haplogruppe sind dabei stets monophyletisch. Für die Zuordnung verwendet man charakteristische Mutationen in den Gensequenzen der mtDNA, außerhalb des D-Loops.
Eine Person kann z. B. die Haplogruppe C1a3b2 haben. Ihre mtDNA ist dann eng mit der einer anderen Person verwandt, die z. B. C1a3b4 hat. Natürlich teilt ihre mtDNA auch eine gemeinsame Vorfahrin mit einer dritten Person, die C1a3c5 hat, aber diese gemeinsame Vorfahrin hatte früher gelebt, noch bevor sich die C1a3-Linie aufgespalten hatte. Das heißt, C1a3b4 und C1a3b2 sind gegenüber C1a3c5 monophyletisch. Ebenso sind C1a3b2 und C1a3c5 monophyletisch gegenüber allen H-Haplotypen usw.
Leider ist die Nomenklatur relativ inkonsequent realisiert. Viele Buchstaben wurden benutzt, um die wichtigsten nichtafrikanischen Haplogruppen zu benennen. Viele alte Haplogruppen kommen jedoch in Afrika vor. Diese bezeichnet man zusammen als „L“ und geht bereits für die Unterteilung der Hauptgruppen zu Ziffern über. Über die Zuordnung mancher afrikanischer Haplotypen (in L1 und L3) besteht bis heute noch kein wissenschaftlicher Konsensus.
Wenn man von der Wurzel anfängt, besteht der mitochondriale Stammbaum des Menschen zunächst aus einer Reihe tiefer Äste. Diese genetischen Linien werden heute L1 genannt. Anders als früher gedacht, ist L1 keine monophyletische Haplogruppe, sondern bildet die Wurzel. L1 sind also eigentlich ein ganzes Paket afrikanischer Haplogruppen, die ähnlich alt sind wie die mitochondriale Eva und deren genaue verwandtschaftliche Beziehung untereinander noch nicht genau geklärt ist.
Von diesen alten L1-Ästen zweigt ein Ast durch eine Mutation an der Position 10810 ab. Von diesem spaltet sich seinerseits die Haplogruppe L2 durch eine Mutation an der Position 16390 ab. Auch L2 kommt praktisch nur bei Afrikanern südlich der Sahara vor.
Eine Mutation an der Position 3594 bildet den Ast, auf dem die großen Haplogruppen M und N sowie noch zahlreiche weitere afrikanische Haplogruppen, die man heute noch unter L3 zusammenfasst, liegen. L3 ist, wie L1, keine echte (monophyletische) Haplogruppe. Die Haplogruppen M und N kommen beim allergrößten Teil der Nichtafrikaner vor. Sie sind in Afrika südlich der Sahara sehr selten, wo L1, L2 und L3 dominieren.
Die Haplogruppe M wird in die großen Haplogruppen M1, Z, C, D, E, G und Q unterteilt. Die Haplogruppe N in N1a, N1b, N9, A, I, W, X und Y, sowie in die Haplogruppe R, die die Unter-Haplogruppen B, F, H, P, T, J, U und K bildet.
Die derzeit umfangreichste Untersuchung von mitochondrialer DNA wurde vom Genographic Consortium durchgeführt (s. a. The Genographic Project). In diesen Vergleich wurden 78.590 genotypische Proben einbezogen und die mitochondrialen Haplogruppen (und deren Untergruppen) wurden in einem phylogenetischen Baum dargestellt.[8]
Geographische Verteilung
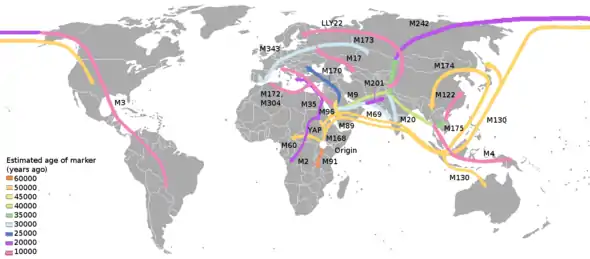
Die „alten“ Haplotypen aus den L-Ästen dominieren in Afrika südlich der Sahara. Es bestehen keine Zweifel, dass sie ihren Ursprung dort haben. Diese Haplotypen finden sich auch in Nordafrika (ca. 50 % Häufigkeit) und, in geringer Häufigkeit, in Europa und Westasien.
Die Haplogruppen M und N dominieren im Rest der Welt und sind in Afrika südlich der Sahara selten. Spezielle Varianten der Haplogruppe M (M1) kommen mit einer Häufigkeit von etwa 20 % in Äthiopien vor. Entweder ist M dort bereits entstanden oder es handelt sich um eine semitische Süd-Rückwanderung.
Bei Amerikanischen Ureinwohnern kommen die Haplogruppen A, B, C, D und X vor; davon entstanden A, B, und X aus einem Ostzweig der Haplogruppe N, C und D dagegen aus Haplogruppe M.
In Europa und Westasien ist Haplogruppe M extrem selten. Die häufigsten Untergruppen gehören in die Untergruppe R: H, V, T, J, U und K. Daneben kommen auch die Haplogruppen I, W und X mit einer signifikanten Häufigkeit vor. In Europa, dem Kaukasus und dem Nahen Osten finden sich praktisch die gleichen Haplogruppen, nur die Häufigkeiten der einzelnen Haplogruppen schwanken. Vor allem die Haplogruppe H ist im Nahen Osten und im Kaukasus deutlich seltener als in Europa (≈25 % versus ≈45 %), während die Haplogruppe K deutlich häufiger ist. Innerhalb Europas schwanken die Häufigkeiten der Haplogruppen je nach Region geringfügig.
Süd- und Ostasien unterscheiden sich bei den Haplogruppen sehr stark von Westasien. Hier kommen, aus der Haplogruppe M, die Haplogruppen C, D, E, G, Z und Q vor. Die Haplogruppe N kommt hier auch vor, allerdings ist sie vor allem durch die Haplogruppen A, B, F, Y und X vertreten.
Die Haplogruppe X ist bemerkenswert, da sie in ganz Eurasien und Nordamerika vorkommt, wenn auch nur mit relativ geringer Häufigkeit. Früher wurde angenommen, dass die Haplogruppe X in Europa entstand und nur in Europa vorkommt. Als die Haplogruppe bei amerikanischen Ureinwohnern entdeckt wurde, kam die Hypothese auf, sie sei vor Jahrtausenden von Europa aus auf dem Seeweg durch europäische Emigranten nach Amerika gelangt. Mittlerweile wurde Haplogruppe X jedoch auch in Asien entdeckt (Derneko et al., 2001).
| Evolutionsbaum Haplogruppen Mitochondriale DNA (mtDNA) | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Alte mitochondriale DNA
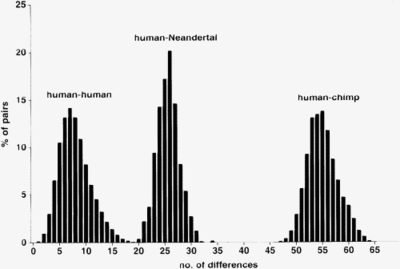
Jede Zelle enthält viele Mitochondrien, die ihrerseits mehrere Kopien der mitochondrialen DNA enthalten. Dies ermöglicht oft eine DNA-Sequenzierung der mtDNA aus Fossilien, die nicht zu alt sind (weniger als 100.000 Jahre). Bis 2006 wurden Teile der mtDNA von zwölf Neandertalern aus Deutschland, Kroatien, Russland, Frankreich, Belgien, Italien und Spanien sequenziert.[9] Das Ergebnis:
- Die mtDNA eines Neandertalers unterscheidet sich etwa dreimal so stark von der eines modernen Menschen, wie die mtDNA zweier moderner Menschen untereinander.
- Die mtDNA der Neandertaler unterscheiden sich untereinander nur wenig. Die mtDNA moderner Menschen unterscheiden sich untereinander wesentlich stärker.
- Die mtDNA der Neandertaler zeigt keine größere Ähnlichkeit zu der mtDNA heutiger Europäer als zu jener heutiger Afrikaner oder Asiaten.
Die mtDNA des Neandertalers hat sich also relativ früh von der Linie, die zum modernen Menschen geführt hat, abgespalten. Man schätzt, dass die gemeinsame mitochondriale Eva von modernem Menschen und Neandertaler vor 550.000 bis 690.000 Jahren gelebt hat, also deutlich früher als die mitochondriale Eva des modernen Menschen (Krings et al., 1997).
Laut Nordborg (1998) und anderen kann man trotz dieser Daten nicht ausschließen, dass es eine Vermischung zwischen Neandertalern und modernen Menschen gab. Currat & Excoffier (2004) meinten hingegen, dass man anhand dieser mtDNA-Daten eine Vermischung praktisch ausschließen kann. Forschungsergebnisse aus den Jahren 2012 bis 2014 zu den Fossilien von Peștera cu Oase in Rumänien und Ust-Ischim wiesen allerdings Hybridisierungen mit Neandertalern nach.[10][11]
Die mtDNA aus allen fossilen Überresten anatomisch moderner Menschen (Homo sapiens), die bis jetzt untersucht wurden, sind hingegen eng mit denen heute lebender Menschen verwandt (Serre et al. 2004). Die mtDNA von „Ötzi“ zum Beispiel gehörte zur mitochondrialen Haplogruppe K1 (Rollo et al. 2006), genauer der Untertyp K1f.[12]
Genetische Diversität der mtDNA
Die Diversität der mitochondrialen DNA-Sequenzen ist in Afrika am größten. Insgesamt ist die genetische Diversität beim Menschen im Vergleich zu den Menschenaffen gering. In einem bestimmten Segment der mtDNA zeigen Schimpansen die drei- bis vierfache genetische Diversität im Vergleich zu Menschen. Allein die mtDNAs von 19 untersuchten Schimpansen aus dem Taï-Nationalpark (Elfenbeinküste) zeigen eine höhere Diversität als die aller Menschen, obwohl jene Affen zu einer kleinen, genetisch durchmischten Gruppe gehören (Gagneux et al., 1999).
Bonobos, Gorillas und Orang-Utans zeigen ebenfalls eine höhere genetische Diversität der mtDNA als der Mensch (Jobling et al., 2004).
| Locus | Schimpanse vs. Mensch | Bonobo vs. Mensch | Gorilla vs. Mensch | Orang-Utan vs. Mensch | Referenz | Anmerkung |
|---|---|---|---|---|---|---|
| mtDNA | 3- bis 4-mal so hoch | höher | höher | höher | Gagneux et al. (1999) | – |
| Y-Chromosom | höher | höher | geringer | höher | Stone et al. (2002) | siehe Adam |
| X-Chromosom | 3-mal so hoch | keine Daten | 2-mal so hoch | 3,5-mal so hoch | Kaessmann et al. (2001) | – |
| Locus | Afrika | Asien | Europa | Referenz | Anmerkung |
|---|---|---|---|---|---|
| mtDNA (paarweise Unterschiede) | 2,08 | 1,75 | 1,08 | Vigilant et al. (1991) | – |
| Y-Chromosom (43 Marker) | 0,841 | 0,904 | 0,852 | Hammer et al. (2001) | siehe Adam |
| X-Chromosom (Nukleotid Diversität) | 0,035 | 0,025 | 0,034 | Kaessmann et al. (1999) | – |
| Autosomen (Nukleotid Diversität) | 0,115 | 0,061 | 0,064 | Yu et al. (2002) | – |
Eva und die Out-of-Africa-Theorie
Die Theorie der mitochondrialen Eva ist mittlerweile anerkannt und etabliert, aber was ihre Aussagen über die Fragestellung von Out-of-Africa-Theorie versus „multiregionaler Ursprung des modernen Menschen“ betrifft, besteht immer noch kein wissenschaftlicher Konsens. Klar ist, dass die Theorie der mitochondrialen Eva nicht im Widerspruch zur Out-of-Africa-Theorie steht. Aber sie widerlegt auch nicht zwangsläufig andere Modelle. Schließlich macht sie nur Aussagen über einen einzigen Locus. Die Loci im Zellkern können andere, dank Rekombination wesentlich kompliziertere, Geschichten haben.
Zum Beispiel argumentierte Nordborg (1998), dass das vollständige Fehlen von Neandertaler-mtDNA bei heutigen Europäern durchaus das Ergebnis von Gendrift sein könnte.
Nordborg (1998) nahm in einem einfachen Modell an, dass der moderne Mensch sich zunächst über Europa ausbreitete, ohne sich mit Neandertalern zu mischen. Erst nachdem die Expansion abgeschlossen war, mischten sich moderne Menschen mit Neandertalern. Nach diesem Modell könnten die Neandertaler bis zu 25 % Beitrag am Genpool der daraus entstanden Mischpopulation gehabt haben. Durch Gendrift starben die mitochondrialen Erblinien der Neandertaler in der Zwischenzeit aus (siehe auch Theorie). Heutige Europäer könnten also in ihrem Zellkern durchaus noch DNA mit Neandertaler-Ursprung haben.
Currat & Excoffier (2004) entwickelten ein wesentlich komplizierteres Modell für die Durchmischung von modernem Menschen und Neandertaler. Die Besiedlung von Europa und die Verdrängung der Neandertaler durch moderne Menschen dauerte etwa 12.000 Jahre (≈500 Generationen). Wenn man bei den Berechnungen davon ausgeht, dass Expansion und Vermischung gleichzeitig abgelaufen sind, bekommen die Neandertaler einen großen Vorteil, weil sie Europa vorher besiedelt hatten.
Wenn z. B. moderne Menschen, von Afrika kommend, Anatolien besiedeln und sich teilweise mit den Neandertalern dort mischen, findet die mtDNA der Neandertaler Eingang in den Genpool der modernen Menschen. Expandiert nun die Population der modernen Menschen, so expandiert auch die importierte Neandertaler-DNA. Wenn jetzt z. B. der Balkan von den „Misch-Nachkommen“ besiedelt wird und es wieder zu einer Vermischung mit den dort lebenden Neandertalern kommt, steigt der Anteil der Neandertaler-DNA im Genpool noch weiter. Je weiter die Expansionswelle moderner Menschen nach Westen und Norden vordringt und sich dabei immer wieder mit den dort lebenden Neandertalern mischt, desto mehr reichert sich Neandertaler-DNA im Genpool an.
Nach diesem Modell würde selbst eine kleine Mischquote ausreichen, damit die mitochondriale DNA des Neandertalers die des modernen Menschen vollständig verdrängt (was nicht der Fall war). Currat und Excoffier (2004) ermittelten, dass es in den 12.000 Jahren, in denen moderne Menschen und Neandertaler Europa bewohnten, nur maximal 120 „Mischkinder“ geben konnte. Die Autoren vermuteten wegen dieser extrem geringen Zahl, dass moderne Menschen und Neandertaler verschiedene Arten sind, die zu keiner gemeinsamen Fortpflanzung fähig waren.
Auch große Teile Asiens waren bereits von Vertretern der Gattung Homo (Homo erectus) bewohnt, als sie von modernen Menschen aus Afrika besiedelt wurden. Auch hier finden sich keine mitochondrialen Erblinien, die auf eine Vermischung hindeuten würden.
Zusammenfassend kann gesagt werden, dass die mitochondriale Eva die Out-of-Africa-Theorie durch folgende Fakten stützt:
- Die mitochondriale DNA beim Menschen weist eine geringe genetische Diversität auf (Gagneux u. a., 1999).
- Die mitochondriale Eva hat mit nur ≈175.000 Jahren ein relativ junges Alter (Ingman u. a., 2000).
- Der mitochondriale Stammbaum zeigt tiefe Äste in Afrika aber sternförmige Struktur außerhalb (Ingman u. a., 2000).
- Die mitochondriale DNA der Neandertaler ist eindeutig verschieden von der heutiger Menschen (Serre u. a., 2004).
- Eine Vermischung von modernen Menschen mit Neandertalern wurde bis zum Jahre 2004 noch für unwahrscheinlich gehalten (Currat & Excoffier, 2004).
Untersuchungen an anderen Loci auf dem X-Chromosom, Y-Chromosom und Autosomen deuten ebenfalls auf einen jungen, afrikanischen Ursprung des Menschen hin (Takahata u. a., 2001).
In den Jahren 2013 bis 2015 wurden von dem Forscherteam um den schwedischen Wissenschaftler Svante Pääbo neue Erkenntnisse zur Vermischung veröffentlicht:
- Verbesserte Analysemethoden zeigten, dass Genfluss stattgefunden habe mit einem Beitrag von bis zu 4 % Neandertaler-Genen zum Genpool heutiger Europäer und Asiaten.[13]
- Forschungsdaten zu den Homo-sapiens-Fossilien von Peștera cu Oase in Rumänien und Ust-Ischim in Sibirien untermauerten diese Aussagen.[10][11]
- Allerdings wurde Genfluss bislang nur in eine Richtung nachgewiesen, nämlich Verpaarung von Homo-sapiens-Männern mit Neandertaler-Frauen.[14]
„Eva“ in der populären Rezeption
- Bryan Sykes hat ein Buch namens Die sieben Töchter Evas geschrieben.
- In River Out of Eden beschreibt Richard Dawkins die menschlichen Vorfahren im Kontext eines Flusses aus Genen und zeigt, dass die mitochondriale Eva eine von vielen gemeinsamen Ahnen ist, die wir auf Grund der verschiedenen genetischen Pfade zurückverfolgen können.
- Der Discovery Channel hat eine Dokumentation namens Die wirkliche Eva ausgestrahlt.
- Der japanische Roman, Horrorfilm und die Videospielserien Parasite Eve benutzen die mitochondriale Eva-Theorie als Basis für eine Fantasiegeschichte über einen Wissenschaftler, der seine Frau mit Hilfe von regenerierten Zellen wiederbelebt, was fatale Folgen hat.
- Greg Egan schrieb eine Kurzgeschichte mit dem Titel Mitochondriale Eva.
- In Ronald D. Moores Science-Fiction-Serie Battlestar Galactica ist Hera als Tochter eines Menschen und einer Zylonin die mitochondriale Eva.
- Lynn Okamoto benutzte die Theorie der mitochondrialen Eva für seinen Manga Elfen Lied. In diesem ist Lucy die mitochondriale Eva einer neuen Rasse namens Diclonius.
Literatur
- Fachliteratur
- M. Currat, L. Excoffier: Modern humans did not admix with Neanderthals during their range expansion into Europe. In: PLoS Biology. Lawrence 2.2004,12, e421. doi:10.1371/journal.pbio.0020421 ISSN 1544-9173.
- M.V. Derenko, T. Grzybowski, B.A. Malyarchuk, J. Czarny, D.M. Sliwka, I.A. Zakharov: The presence of mitochondrial haplogroup x in Altaians from South Siberia. In: American Journal of Human Genetics(Am J Hum Genet). New York 69.2001,1, 237-241. PMID 11410843 ISSN 0002-9297.
- P. Gagneux, C. Wills, U. Gerloff, D. Tautz, P.A. Morin, C. Boesch, B. Fruth, G. Hohmann, O.A. Ryder, D.S. Woodruff: Mitochondrial sequences show diverse evolutionary histories of African hominoids. In: Proceedings of the National Academy of Sciences of the United States of America (PNaS U S A). Washington 96.1999,9, 5077-5082. doi:10.1073/pnas.96.9.5077 ISSN 0027-8424.
- M.F. Hammer, T.M. Karafet, A.J. Redd, H. Jarjanazi, S. Santachiara-Benerecetti, H. Soodyall, S.L. Zegura: Hierarchical patterns of global human Y-chromosome diversity. In: Molecular biology and evolution (Mol Biol Evol). Oxford 18.2001,7, 1189–1203. PMID 11420360 ISSN 0737-4038.
- M. Ingman, H. Kaessmann, S. Pääbo, U. Gyllensten: Mitochondrial genome variation and the origin of modern humans. In: Nature. London 408.2000,6813, 708-713. doi:10.1038/35047064 ISSN 0028-0836.
- Mark A.Jobling, Chris Tyler-Smith, Matthew Hurles: Human Evolutionary Genetics. Origins, Peoples and Disease. ISBN 0-8153-4185-7.
- H. Kaessmann, F. Heissig, A. von Haeseler, S. Pääbo: DNA sequence variation in a non-coding region of low recombination on the human X chromosome. In: Nature Genetics (Nat Genet). New York 22.1999,1, 78–81. doi:10.1038/8785 ISSN 1061-4036.
- H. Kaessmann, V. Wiebe, G. Weiss, S. Pääbo: Great ape DNA sequences reveal a reduced diversity and an expansion in humans. In: Nature Genetics (Nat Genet). New York 27.2001,2, 155–156. doi:10.1038/84773 ISSN 1061-4036.
- M. Krings, A. Stone, R.W. Schmitz, H. Krainitzki, M. Stoneking, S. Pääbo: Neandertal DNA sequences and the origin of modern humans. In: Cell. Cambridge 90.1997,1, 19-30. doi:10.1016/S0092-8674(00)80310-4 ISSN 0092-8674.
- M. Nordborg: On the probability of Neanderthal ancestry. In: American Journal of Human Genetics (Am J Hum Genet). New York 63.1998,4, 1237-1240. PMID 9758610 ISSN 0002-9297.
- F. Rollo, L. Ermini, S. Luciani, I. Marota, C. Olivieri, D. Luiselli: Fine characterization of the Iceman’s mtDNA haplogroup. in: American journal of physical anthropology (Am J Phys Anthropol). New York 130.2006,4, 557-564. doi:10.1002/ajpa.20384 ISSN 0002-9483.
- Serre, D.; Langaney, A.; Chech, M.; Nicola, M.T.; Paunovic, M.; Mennecier, P.; Hofreiter, M.; Possnert, G. & Pääbo, S. (2004): No evidence of Neandertal mtDNA contribution to early modern humans. PLoS Biol 2(3), E57. doi:10.1371/journal.pbio.0020057.
- Stone, A. C.; Griffiths, R. C.; Zegura, S. L. & Hammer, M. F. (2002): High levels of Y-chromosome nucleotide diversity in the genus Pan. Proc Natl Acad Sci U S A 99(1), 43–48. doi:10.1073/pnas.012364999.
- Vigilant, L.; Stoneking, M.; Harpending, H.; Hawkes, K. & Wilson, A. C. (1991): African populations and the evolution of human mitochondrial DNA. Science 253(5027), 1503–1507. doi:10.1126/science.1840702.
- Takahata, N.; Lee, S.H. & Satta, Y. (2001): Testing multiregionality of modern human origins. Mol Biol Evol 18(2), 172-183. PMID 11158376.
- Yu, N.; Chen, F.; Ota, S.; Jorde, L. B.; Pamilo, P.; Patthy, L.; Ramsay, M.; Jenkins, T.; Shyue, S. & Li, W. (2002): Larger genetic differences within africans than between Africans and Eurasians. Genetics 161(1), 269–274. PMID 12019240.
- Rezeption
- Bryan Sykes: Die sieben Töchter Evas. Lübbe-Verlag, Bergisch Gladbach 2001, ISBN 3-7857-2060-2.
Weblinks
Einzelnachweise
- vgl. J. Hein, M.H. Schierup, C. Wiuf: Gene genealogies, variation and evolution: a primer in coalescent theory. Oxford University Press, Oxford 2005.
- Alan R. Templeton: Haplotype trees and modern human origins. Yearbook of physical Anthropology 48, 2005, S. 33–59.
- C.F. Aquadro, B.D. Greenberg: Human mitochondrial DNA variation and evolution, analysis of nucleotide sequences from seven individuals. (PDF; 1,7 MB) In: Genetics. Jg. 103, Bethesda 1983, S. 287–312. PMID 6299878 ISSN 0016-6731
- M.J. Johnson u. a.: Radiation of human mitochondria DNA types analyzed by restriction endonuclease cleavage patterns. In: Journal of molecular evolution. Jg. 19, New York 1983, S. 255–271. PMID 6310133 doi:10.1007/BF02099973 ISSN 0022-2844
- R.L. Cann u.a: Mitochondrial DNA and human evolution. In: Nature. Jg. 325, London 1987, S. 31–36. PMID 3025745 doi:10.1038/325031a0 ISSN 0028-0836
- G. David Poznik et al.: Sequencing Y Chromosomes Resolves Discrepancy in Time to Common Ancestor of Males Versus Females. In: Science. Band 341, Nr. 6145, 2013, S. 562–565, doi:10.1126/science.1237619
- Macaulay und Richards
- D.M. Behar u. a.: The Genographic Project public participation mitochondrial DNA database. In: PLoS Genet. Jg. 3, San Francisco 2007, S.e104. PMID 17604454 doi:10.1371/journal.pgen.0030104 ISSN 1553-7390
- R.E. Green u. a.: Analysis of one million base pairs of Neanderthal DNA. In: Nature. Jg. 444, London 2006, S. 330–336. PMID 17108958 doi:10.1038/nature05336 ISSN 0028-0836
- Frühe Europäer haben sich mit Neandertalern vermischt. Abgerufen auf mpg.de am 12. Juli 2015, mit einer Abbildung des Unterkiefers Oase1
- Erbgut des bisher ältesten modernen Menschen entschlüsselt. Max-Planck-Gesellschaft vom 22. Oktober 2014.
- European Academy of Bozen/Bolzano (EURAC), 15. Januar 2016 – NPO
- Genfluss vom Neandertaler zum Homo sapiens. Abgerufen auf mpg.de am 28. Juni 2015.
- Neandertaler-Genom beim Homo sapiens nachgewiesen, noch kein Nachweis von sapiens-Genom beim Neandertaler. Abgerufen auf mpg.de am 20. Juli 2015.