Kaumoebavirus
Die vorgeschlagene Spezies „Kaumoebavirus“[3] von Riesenviren ist morphologisch und genetisch ähnlich zwei anderen vorgeaschlagenen Taxa von Riesenviren, „Faustovirus“ und „Pacmanvirus“, sowie den Viren der Familie Asfarviridae mit dem Asfivirus (ASFV), dem Erreger der Afrikanischen Schweinepest. Kaumoebaviren haben ein ikosaedrisches Kapsid von etwa 250 nm Größe, das Genom besteht aus einem dsDNA-Molekül mit einer Länge von 350 kbp. Es enthält vermutlich 465 Gene, die Proteine kodieren.[1]
| „Kaumoebavirus“ | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
_Clumps.png.webp)
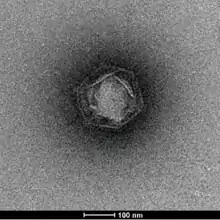
Kaumoebavirus in Vermamoeba vermiformis, die Virionen teilweise in Klumpen von 2–4.[1][Anm. 1] | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| „Kaumoebavirus“ | ||||||||||||||||
| Links | ||||||||||||||||
|
Das Virus wurde 2016 bei der Kultivierung von Amöben der Gattung Vermamoeba vermiformis (Tubulinea) aus einer Abwasserprobe aus dem Süden Saudi-Arabiens gefunden. Der Name ist ein Akronym für englisch King Abdulaziz University amoeba virus (König-Abdulaziz-Universität-Amöbenvirus).[1]
Bisher (Stand April 2020) ist die Gattung „Kaumoebavirus“ noch nicht in der Datenbank des International Committee on Taxonomy of Viruses (ICTV) registriert[2]
Aufbau
Wie oben erwähnt, haben die Viruspartikel (Virionen) von „Kaumoebavirus“ ein ikosaedrisches Kapsid von etwa 250 nm Größe. Sein Genom wird durch ein zirkuläres dsDNA-Molekül dargestellt, das ungefähr 351.731 bp enthält. Es gibt im Genom keine repetitiven Sequenzen (englisch repetitive sequences) mit Ausnahme einer langen invertierten Tandem-Wiederholung (englisch inverted tandem repeat) von 1.407 bp Länge. Der GC-Gehalt des Genoms beträgt 43,7 %. Entsprechend den bioinformatischen Daten enthält das Genom 465 Gene, die für Proteine kodieren, welche eine Länge von 113 bis 6209 Aminosäure-Einheiten haben. In seinem Genom wurden keine tRNA-Gene identifiziert. Was Homologie der Gene anbelangt, so sind
- 74 (59 %) der Kaumoebavirus-Proteine anderen viralen Proteinen nahestehend,
- 19 (15 %) sind homolog zu Bakterienproteinen, 2 (2 %) zu Archaeengenen,
- 31 (25 %) zu eukaryotischen Proteinen.
Wie bei „Faustovirus“ kommt es bei „Kaumoebavirus“ während der Reifung der mRNA zur Spleißung (englisch splicing).[1]
Vermehrungszyklus
_VF.png.webp)
In einem frühen Stadium der Infektion (2 Stunden nach dem Eindringen in die Wirtszellen der Amöbe Vermamoeba vermiformis) werden Phagozytose-Vakuolen, die Virionen (Virusteilchen) von „Kaumoebavirus“ enthalten, im Zytoplasma der Zelle beobachtet. Nach weiteren drei Stunden sind bereits Cluster von 2 bis 4 Viruspartikeln in den Vakuolen gebildet. 4 Stunden nach der Penetration tritt virale DNA in das Zytoplasma ein, und innerhalb der nächsten zwei Stunden können Virusfabriken in der Nähe des Zellkerns beobachtet werden, in denen das virale Genome repliziert und sich neue Virionen ansammeln; 8 bis 16 Stunden nach der Infektion werden die neu gebildeten Viruspartikel in der Virusfabrik nachgewiesen. Während der Replikation verändert „Kaumoebavirus“ die Morphologie des Zellkerns nicht. Schließlich tritt eine Zelllyse auf und die Virionen treten aus. Die Gesamtdauer des Vermehrungszyklus beträgt bei „Kaumoebavirus“ wie bei „Faustovirus“ 16 bis 20 Stunden.[1]
Systematik
De Gattung „Kaumoebavirus“ hat die Spezies, „Kaumoebavirus LCC10“ und „Kaumoebavirus Sc“.[4]
Unter den Virusproteinen ähneln Proteine anderer Viren des Phylums der Nucleocytoviricota (veraltet Nucleocytoviricoa, NCLDV, früher auch als Ordnung „Megavirales“ vorgeschlagen), den „Kaumoebavirus“-Proteinen am nächsten, insbesondere gilt dies für ein weiteres Riesenvirus, das „Faustovirus“, und die Mitglieder der Familie der Asfarviridae. Zur weiteren Verwandtschaft von „Kaumoebavirus“ gehören die Familien Mimiviridae, Phycodnaviridae, Poxviridae, Marseilleviridae, sowie der nicht-NCLDV-Familie Baculoviridae. Aus evolutionärer Sicht nimmt „Kaumoebavirus“ eine Zwischenposition zwischen „Faustovirus“ und den Asfarviridae ein.[1]
2018 wurden die Ergebnisse der Analyse veröffentlicht, die die enge Verwandtschaft von „Kaumoebavirus“ mit „Faustovirus“ und den Asfarviridae weiter bestätigten. Die Studie prüfte, ob die Sequenzen, die für die zuvor identifizierten Asfarviridae-Promotoren charakteristisch sind, ebenfalls für die riesigen Kaumoebaviren und Faustoviren charakteristisch sind. Zuvor wurde bereits gezeigt, dass die Vertreter der Asfarviridae durch das Vorhandensein von AT-reichen Sequenzen in Promotoren charakterisiert sind. Es stellte sich dann heraus, dass „Kaumoebavirus“ und „Faustovirus“ von AT-reichen TATTT- und TATATA-Motiven in Promotoren dominiert werden, was sie zusätzlich näher an die Asfarviridae bringt.[5]
Auf Basis der Gensequenz von Genen der DNA-Polymerase-B-Familie bilden „Pacmanvirus“, „Kaumoebavirus“, „Faustovirus“ und die Asfarviridae einen phylogenetischen Baum (Klade), offenbar hatten diese Viren einen gemeinsamen Vorfahren. In Bezug auf die Kapsid-Architektur steht Faustovirus dem Pacmanvirus am nächsten.[6]
Die meisten Autoren schlagen vor, die Gattung Faustovirus als Prototyp einer neuen Familie Faustoviridae[1] innerhalb der Nucleocytoplasmic large DNA viruses (NCLDV, vom ICTV im März neu eingerichtetes Phylum Nucleocytoviricota)[2] aufzufassen, die der Familie Asfarviridae mit Gattung Asfivirus (ASFV) nahesteht, aber von ihr immer noch verschieden ist.[7][8][9] Das ICTV hat im März für die nähere Verwandtschaft (englisch extended Afarviridae[4]) der Asfarviridae die neue Ordnung Asfuvirales eingerichtet, womit ein Taxon für die gemeinsame Klade der Asfaviridae und „Faustoviridae“ zur Verfügung steht. Schulz et al (2018) schlagen für diese Klade eine Systematik wie folgt vor:[10]
| Asfuvirales[2] |
| ||||||||||||||||||
Im Vergleich dazu sehen Guglielmini et al. (2019), Fig. 2, die Positionen von Asfarviridae und „Kaumoebavirus“ vertauscht.[12] Als mögliches weiteres Mitglied dieser erweiterten Asfarviridae-Gruppe wurde das Dinodnavirus vorgeschlagen.[13]
Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- Leena H. Bajrai, Samia Benamar, Esam I. Azhar, Catherine Robert, Anthony Levasseur, Didier Raoult, Bernard La Scola; Eric O. Freed (Hrsg.): Kaumoebavirus, a New Virus That Clusters with Faustoviruses and Asfarviridae. In: Viruses Volume 8, Nr. 11, 28. Oktober 2016, Special Edition: Viruses of Protozoa; 278, doi: 10.3390/v8110278, PMC 5127008 (freier Volltext), PMID 27801826.
 CC-BY icon.svg
CC-BY icon.svg - ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- NCBI: Kaumoebavirus (species)
- Khalil Geballa-Koukoulas, Julien Andreani, Bernard La Scola, Guillaume Blanc: The Kaumoebavirus LCC10 Genome Reveals a Unique Gene Strand Bias among “Extended Asfarviridae”, in: Viruses, Band 13, Nr. 2, Section Viruses of Plants, Fungi and Protozoa, 148, Viruses of Plants, Fungi and Protozoa, 15. Januar 2021, doi:10.3390/v13020148.

- Oliveira G. P., de Aquino I. L. M., Luiz A. P. M. F., Abrahão J. S.: Putative Promoter Motif Analyses Reinforce the Evolutionary Relationships Among Faustoviruses, Kaumoebavirus, and Asfarvirus. In: Frontiers In Microbiology, Band 9 vom 23. Mai 2018, S. 1041, eCollection 2018, doi:10.3389/fmicb.2018.01041, PMID 29875752
- J. Andreani, J. Y. B. Khalil, M. Sevvana, S. Benamar, F. Di Pinto, I. Bitam, F. Colson, T. Klose, M. G. Rossmann, D. Raoult, B. La Scola: Pacmanvirus, a New Giant Icosahedral Virus at the Crossroads between Asfarviridae and Faustoviruses. In: Journal Of Virology 91(14), 26. Juni 2017, pii: e00212-17. Print 15. Juli 2017. doi:10.1128/JVI.00212-17, PMID 28446673
- D. G. Reteno, S. Benamar, J. B. Khalil, J. Andreani, N. Armstrong, T. Klose, M. Rossmann, P. Colson, D. Raoult, B. La Scola: Faustovirus, an asfarvirus-related new lineage of giant viruses infecting amoebae. In: Journal of virology. Band 89, Nr. 13, Juli 2015, S. 6585–6594, doi:10.1128/JVI.00115-15, PMID 25878099, PMC 4468488 (freier Volltext).
- S. Benamar, D. G. Reteno, V. Bandaly, N. Labas, D. Raoult, B. La Scola: Faustoviruses: Comparative Genomics of New Megavirales Family Members. In: Frontiers in microbiology. Band 7, 2016, S. 3, doi:10.3389/fmicb.2016.00003, PMID 26903952, PMC 4742530 (freier Volltext).
- T. Klose, D. G. Reteno, S. Benamar, A. Hollerbach, P. Colson, B. La Scola, M. G. Rossmann: Structure of faustovirus, a large dsDNA virus. In: Proceedings of the National Academy of Sciences. Band 113, Nr. 22, Mai 2016, S. 6206–6211, doi:10.1073/pnas.1523999113, PMID 27185929, PMC 4896704 (freier Volltext).
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1038/s41467-018-07335-2
- Pierre-Philippe Dechant: Recent developments in mathematical virology. (PDF; 24 MB) ICERM, York St. John University, 15. November 2018
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS 116(39), 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- H. Ogata, K. Toyoda, Y. Tomaru, N. Nakayama, Y. Shirai, J. M. Claverie, K. Nagasaki: Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus. In: Virol J., 6, 2009, S. 178.
Weblinks
- Kaumoebavirus im National Center for Biotechnology Information (NCBI), Zugriffsdatum 7. Juli 2019.
- Taxonomy - Kaumoebavirus, auf: UniProt, Zugriffsdatum 7. Juli 2019.
- dsDNA Viruses > Asfarviridae ICTV Report vom März 2018, Fig. 4
- Leena H. Bajrai, Samia Benamar, Esam I. Azhar, Catherine Robert, Anthony Levasseur, Didier Raoult, Bernard La Scola; Eric O. Freed (Hrsg.): Kaumoebavirus, a New Virus That Clusters with Faustoviruses and Asfarviridae. In: Viruses Volume 8, Nr. 11, November 2016, S. 278, doi:10.3390/v8110278, PMC 5127008 (freier Volltext), PMID 27801826
- Vermamoeba (Wikimedia)
- Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy, in: Virol J., 16, 126, 4. November 2019, doi:10.1186/s12985-019-1244-3, PDF
_Mature.png.webp)