Faustovirus
„Faustovirus“ („FauV“ oder „FSTV“) bzw. „Faustoviridae“ ist die vorgeschlagene Bezeichnung für eine Gattung bzw. Familie von Doppelstrang-DNA-Viren.[2][3][4][5]
| „Faustovirus“ | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|

| ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| „Faustovirus“ | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| „FauV“/„FSTV“ | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Eigenschaften
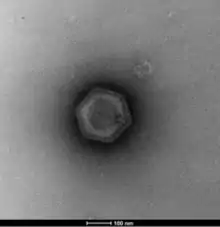
Die „Faustoviren“ sind Riesenviren aus dem Phylum Negarnaviricota (veraltetNucleocytoplasmic large DNA viruses, NCLDV; früher gelegentlich auch als Ordnung Megavirales eingestuft). Sie infizieren Amöben.[6] Sie wurden erstmals 2015 in Vermamoeba verformis (Arcellinida) beschrieben, die vor allem in der Umgebung von Menschen vorkommt.[7]
Struktur
Die größte Ähnlichkeit zu anderen Viren besteht mit denen der Familie Asfarviridae, wobei die Virusteilchen (Virionen) von „Faustovirus“ (wie auch das nicht näher verwandte Coltivirus der Familie Reoviridae) eine doppelte Proteinhülle zur Verpackung des Genoms aufweist.[5] Faustoviren besitzen einen Durchmesser von 2.400 Ångström.[5] Das Kapsid ist ungefähr ikosaedrisch.[5] Symmetrie T=277.[9]
Genom
Das Genom der Faustoviren besteht aus dsDNA und hat eine Länge von 466 kbp (Faustovirus ST1: 470.659 bp, Faustovirus D6: 462011 bp) und kodiert vorhergesagt 478 (respektive 495) virale Proteine, von denen etwa zwei Drittel keine bekannten Homologe aufweisen.[5] Der GC-Gehalt liegt bei 37 % (respektive 38 %).[10] Ungewöhnlich für Viren enthält das Genom der Faustoviren mehrere Introns.[5] Das Gen des Haupt-Kapsidproteins MCP (englisch major Capsid protein) kodiert für 652 Aminosäuren, hat aber eine ungewöhnliche Länge von 17 kB.[5]
Replikationszyklus
Der Replikationszyklus der Faustoviren dauert etwa 24 Stunden und endet mit der Zelllyse zur Freisetzung der neugebildeten Virionen.[5]
Systematik
Innere Systematik
In der Klade der „Faustoviren“ (Gattung „Faustovirus“ oder Familie „Faustoviridae“) gibt es nach Geballa-Koukoulas et al. (2020), Rolland et al. (2019), Borges et al. (2019), Andreani et al. (2018) und Reteno et al. (2015) folgende Untergruppen und einzelne Vertreter (nach NCBI: Spezies):[11][8][12][13][14]
- Gattung: „Faustoviren“ Gattung „Faustovirus“ oder Familie „Faustoviridae“[3]
- „Klade D+E9“
- „Faustoviru-Klades D“
- „Faustovirus D3“
- „Faustovirus D5b“
- „Faustovirus D6“
- „Faustovirus VV10“
- „Faustovirus-Klade E9“
- „Faustovirus E9“
- „Faustovirus LC9“
- „Faustovirus LCD7“
- „Faustovirus M6“
- „Faustovirus S17“
- „Faustovirus VV57“
- „Faustovirus VV63“
- „Klade L+M“
Äußere Systematik
Viele Autoren schlagen vor, die „Faustoviren“ in einer neuen Familie Faustoviridae[4] innerhalb der Nucleocytoplasmic large DNA viruses (NCLDV) zusammenzufassen. Diese Gruppe wurde im März 2020 vom ICTV offiziell als Phylum Nucleocytoviricota bestätigt und umfasst außer den Pockenviren alle bekannten Riesenviren. Die Faustoviren stehen innerhalb der NCLDV der Familie Asfarviridae mit Gattung Asfivirus (ASFV) am nächsten, auch wenn sie von diesen Viren verschieden sind.[7][6][5] Das ICTV hat im März für die nähere Verwandtschaft der Asfarviridae die neue Ordnung Asfuvirales eingerichtet, womit ein Taxon für die gemeinsame Klade der Asfaviridae und „Faustoviridae“ zur Verfügung steht. Geballa-Koukoulas et al (2020) und Schulz et al (2018) schlagen für diese Klade eine Systematik wie folgt vor:[11][16]
| Asfuvirales[1] |
| ||||||||||||||||||||||||
Im Vergleich dazu sehen Guglielmini et al. (2019), Fig. 2, die Positionen von Asfarviridae und „Kaumoebavirus“ vertauscht,[18] und nach Rolland et al. (2019) bilden diese beiden eine gemeinsame Schwesterklade zur Klade der „Faustoviren“ und „Pacmanvirus“. Als mögliches weiteres Mitglied dieser erweiterten Asfarviridae-Gruppe wurde die Gattung Dinodnavirus vorgeschlagen.[19]
Weblinks
- Centre national de la recherche scientifique (CNRS):
- List of the main “giant” viruses known as of today. (PDF; 334 kB) Université Aix Marseille, Centre national de la recherche scientifique (CNRS), 18. April 2018
- List of the main “giant” viruses known as of today (March 2019), Université Aix Marseille, März 2019
- dsDNA Viruses > Asfarviridae ICTV Report vom März 2018, Fig. 4
- Gabriel Augusto Pires de Souza, Victória Fulgêncio Queiroz, Maurício Teixeira Lima, Erik Vinicius de Sousa Reis, Luiz Felipe Leomil Coelho, Jônatas Santos Abrahão: Virus goes viral: an educational kit for virology classes, in: Virology Journal, Band 17, Nr. 13, 31. Januar 2020, doi:10.1186/s12985-020-1291-9

Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy, in: Virol J 16, 126, 4. November 2019, doi:10.1186/s12985-019-1244-3, PDF
- NCBI: Faustovirus (list)
- Leena H. Bajrai, Samia Benamar, Esam I. Azhar, Catherine Robert, Anthony Levasseur, Didier Raoult, Bernard La Scola; Eric O. Freed (Hrsg.): Kaumoebavirus, a New Virus That Clusters with Faustoviruses and Asfarviridae. In: VirusesVolume 8, Nr. 11, 28. Oktober 2016, S. 278, doi: 10.3390/v8110278, PMC 5127008 (freier Volltext), PMID 27801826
- T. Klose, D. G. Reteno, S. Benamar, A. Hollerbach, P. Colson, B. La Scola, M. G. Rossmann: Structure of faustovirus, a large dsDNA virus. In: Proceedings of the National Academy of Sciences. Band 113, Nummer 22, Mai 2016, S. 6206–6211, doi:10.1073/pnas.1523999113, PMID 27185929, PMC 4896704 (freier Volltext).
- S. Benamar, D. G. Reteno, V. Bandaly, N. Labas, D. Raoult, B. La Scola: Faustoviruses: Comparative Genomics of New Megavirales Family Members. In: Frontiers in microbiology. Band 7, 2016, S. 3, doi:10.3389/fmicb.2016.00003, PMID 26903952, PMC 4742530 (freier Volltext).
- D. G. Reteno, S. Benamar, J. B. Khalil, J. Andreani, N. Armstrong, T. Klose, M. Rossmann, P. Colson, D. Raoult, B. La Scola: Faustovirus, an asfarvirus-related new lineage of giant viruses infecting amoebae. In: Journal of virology. Band 89, Nummer 13, Juli 2015, S. 6585–6594, doi:10.1128/JVI.00115-15, PMID 25878099, PMC 4468488 (freier Volltext).
- Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere, auf: Viruses Band 11, Nr. 4, doi:10.3390/v11040312.
 Siehe insbes. Fig. 4A.
Siehe insbes. Fig. 4A. - Genki Yoshikawa, Romain Blanc-Mathieu, Chihong Song, Yoko Kayama, Tomohiro Mochizuki, Kazuyoshi Murata, Hiroyuki Ogata, Masaharu Takemura: Medusavirus, a novel large DNA virus discovered from hot spring water. In: Journal of Virology, ISSN 0022-538X. 93, Nr. 8, 2019. doi:10.1128/JVI.02130-18. PMID 30728258. Abgerufen am 2. Juli 2019. PDF
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Khalil Geballa-Koukoulas, Hadjer Boudjemaa, Julien Andreani, Bernard la Scola, Guillaume Blanc: Comparative Genomics Unveils Regionalized Evolution of the Faustovirus Genomes, in: Viruses 12(5):577, Mai 2020, doi:10.3390/v12050577
- Iara Borges, Rodrigo Araújo Lima Rodrigues, Fábio Pio Dornas, Gabriel Almeida, Isabella Aquino, Cláudio Antônio Bonjardim, Erna Geessien Kroon, Bernard La Scola, Jônatas Santos Abrahão; Joanna L. Shisler (Hrsg.): Trapping the Enemy: Vermamoeba vermiformis Circumvents Faustovirus Mariensis Dissemination by Enclosing Viral Progeny inside Cysts, in: Journal of Virology Band 93, Nr. 14, doi:10.1128/JVI.00312-19, PMID 31019058
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses, in: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643
- Dorine Gaëlle Reteno, Samia Benamar, Jacques Bou Khalil, Julien Andreani, Nicholas Armstrong, Thomas Klose, Michael Rossmann, Philippe Colson, Didier Raoult, Bernard La Scola; G. McFadden (Hrsg.): Faustovirus, an Asfarvirus-Related New Lineage of Giant Viruses Infecting Amoebae, in: Journal of Virology Band 89, Nr. 13, 3. Juni 2015, doi:10.1128/JVI.00115-15, PMID 25878099
- Amina Cherif Louazani, Emeline Baptiste, Anthony Levasseur, Philippe Colson, Bernard La Scola: Faustovirus E12 Transcriptome Analysis Reveals Complex Splicing in Capsid Gene, in: Front. Microbiol., 23. Oktober 2018, doi:10.3389/fmicb.2018.02534
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1038/s41467-018-07335-2.

- Pierre-Philippe Dechant: Recent developments in mathematical virology. (PDF; 23,6 MB) ICERM, York St. John University, 15. November 2018
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- H. Ogata, K. Toyoda, Y. Tomaru, N. Nakayama, Y. Shirai, J. M. Claverie, K. Nagasaki: Remarkable sequence similarity between the dinoflagellate-infecting marine girus and the terrestrial pathogen African swine fever virus. In: Virol J. 6, 2009, S. 178.