Coccolithovirus
Coccolithovirus ist eine Gattung doppelsträngiger DNA-Riesenviren in der Familie Phycodnaviridae. Als natürliche Wirte dienen Algen, speziell Emiliania huxleyi, eine Spezies der Isochrysidales [en] (zusammen mit den Coccolithales [en] gelegentlich als Coccolithophorida bzw. Coccolithophores [en] bezeichnet).[2][3][4][5][6]
| Coccolithovirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
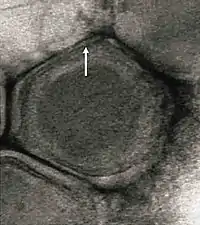
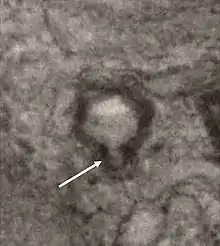
Virion von EhV-86 mit der interner Lipidmembran (Pfeil) | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Coccolithovirus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| EhV-86 | ||||||||||||||||||
| Links | ||||||||||||||||||
|
In dieser Gattung gibt es mit Stand März 2019 nur eine einzige vom International Committee on Taxonomy of Viruses (ICTV) bestätigte Spezies, das Emiliania huxleyi-Virus 86 (EhV-86).[7][8][9] Als Gattung von DNA-Riesenviren wird Coccolithovirus dem Phylum Nucleocytoviricota (veraltet Nucleocytoplasmic large DNA viruses, NCLDV) zugerechnet.
Aufbau
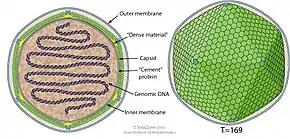
Coccolithoviren sind umhüllt, ikosaedrisch und haben einen Durchmesser im Bereich von 100–220 nm. Ihr Genom ist linear mit einer Größe 410–415 Kilobasen. Das bedeutet, dass ihre DNA für ungefähr 472 Proteine kodieren sollte.[7]
Vermehrungszyklus
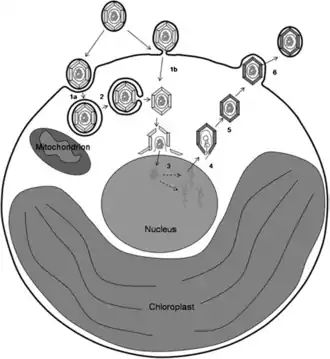
Die Riesenviren der NCLDV replizieren entweder ausschließlich im Zytoplasma der Wirtszelle oder beginnen ihren Lebenszyklus im Wirtskern, vervollständigen ihn jedoch im Zytoplasma. Im Fall von EhV-86 ist die Infektionsstrategie nicht vollständig verstanden, aber Mackinder et al. haben 2009 folgendes Modell vorgeschlagen:[10]
Das Virus dringt durch Endozytose in die Wirtszelle ein, gefolgt von der Fusion der Lipidmembran des Virus mit der Vakuolenmembran des Wirts und der Freisetzung des Virus-Nukleoproteinkerns in das Zytoplasma. Alternativ könnte die Virusmembran direkt mit der Wirtsplasmamembran fusionieren. Das Virusgenom wird dann vom Kapsid in den Zellkern freigesetzt, wo es von der viralen DNA-Polymerase repliziert wird. Das replizierte Genom wird in zusammengebaute Kapside im Zytoplasma gepackt, und die neu gebildeten (etwa 400–1000) Virionen (Viruspartikel) werden zur Plasmamembran transportiert und durch einen kontrollierten Knospungsmechanismus (en.: budding, siehe auch Replikation des Rabiesvirus) freigesetzt, der zum Zerfall der Wirtszelle führt.
Wie man weiß, kann E. huxleyi saisonale Algenblüten hervorrufen, die 250.000 km² erreichen können, wobei die Zelldichte in den oberen 200 m von 103 auf 105 Zellen pro ml Meerwasser steigt.[11] Diese Algenblüte kollabiert normalerweise nach 5–8 Tagen. Mehrere Studien haben gezeigt, dass der Zusammenbruch der Algenblüte intrinsisch mit der Infektion durch Coccolithoviren zusammenhängt.[12]
Die Übertragung der Viren zwischen den Algenwirten erfolgt durch passive Diffusion. Darüber hinaus wurde EhV-DNA auch in Ruderfußkrebsen (Copepoda) nachgewiesen, was vermuten lässt, dass virentragendes Zooplankton zu einer verstärkten Verbreitung der Viren beiträgt.[13]
Genom
Alleine zwischen 1999 und 2008 wurden 14 EhV-Stämme vor allem vom Ärmelkanal, aber auch von der norwegischen und schottischen Küste isoliert.[14][15][16][17][18]
Obwohl aufgrund der sich stark wiederholenden Natur des Genoms Teilsequenzen aller dieser 14 Stämme verfügbar sind, ist EhV-86 der einzige Stamm, der vollständig sequenziert und vom ICTV offiziell als Spezies anerkannt wurde (Stand März 2019).[19][9]
Die Sequenzierung von EhV-86 ergab ein zirkuläres Genom mit einer Länge von 407.339 bp bei einem GC-Gehalt von 40,2 % und vorhergesagten 472 kodierenden Sequenzen (CDS, entspricht in etwa: Genen).[20] Bemerkenswerterweise haben 80 % dieser mutmaßlichen Gene noch keine Datenbank-Homologe. Zu den Funktionen, denen aufgrund von Sequenzähnlichkeit oder Übereinstimmungen mit der Proteindomäne eine Funktion zugewiesen werden konnte, gehören DNA- und RNA-Polymerase-Untereinheiten, acht Proteasen sowie mindestens vier Gene, die für an der Sphingolipid-Biosynthese beteiligte Proteine kodieren. Es wurde gezeigt, dass diese durch horizontalen Gentransfer vom Wirt erhalten wurden.[21][22]
Systematik
Innere Systematik
Neben der vom International Committee on Taxonomy of Viruses (ICTV) bestätigten Spezies Emiliania huxleyi virus 86 wurden noch eine etliche weitere Spezies, wie „Emiliania huxleyi virus 145“, vorgeschlagen.[23] Insgesamt nennt das NCBI mehr als 17 vorgeschlagene Mitglieder der Gattung:[24][25]
- Spezies Emiliania huxleyi virus 86 (EhV-86, ICTV-bestätigt)[26][27]
- Spezies „Emiliania huxleyi virus 18“ (EhV-18)
- Spezies „Emiliania huxleyi virus 84“ (EhV-84)
- Spezies „Emiliania huxleyi virus 88“ (EhV-88)
- Spezies „Emiliania huxleyi virus 99B1“ (EhV-99B1)
- Spezies „Emiliania huxleyi virus 145“ (EhV-145)
- Spezies „Emiliania huxleyi virus 156“ (EhV-156)
- Spezies „Emiliania huxleyi virus 163“ (EhV-163)
- Spezies „Emiliania huxleyi virus 164“ (EhV-164)
- Spezies „Emiliania huxleyi virus 201“ (EhV-201)
- Spezies „Emiliania huxleyi virus 202“ (EhV-202)
- Spezies „Emiliania huxleyi virus 203“ (EhV-203)
- Spezies „Emiliania huxleyi virus 204“ (EhV-204)
- Spezies „Emiliania huxleyi virus 205“ (EhV-205)
- Spezies „Emiliania huxleyi virus 206“ (EhV-2061)
- Spezies „Emiliania huxleyi virus 207“ (EhV-207)
- Spezies „Emiliania huxleyi virus 208“ (EhV-208)
- Spezies „Emiliania huxleyi virus 209“ (EhV-209)
- Spezies „Emiliania huxleyi virus V2“ (EhV-V2)
Phylogenetische Beziehungen nach Guglielmini et al. (2019), Fig. 2:[28]
| Coccolithovirus |
| |||||||||||||||
Äußere Systematik
Die folgende Systematik folgt Schulz et al., (2018)[29] erweitert (und leicht berichtigt) nach Hao et al. (2018),[30] stimmt überein mit Guglielmini et al. (2019):[28]
| Phycodnaviridae [31] |
| ||||||||||||||||||||||||||||||||||||||||||||||||
Koonin und Yutin (2018) geben eine andere Darstellung. Die Phycodnaviridae sind hier nicht mehr monophyletisch, insbesondere nimmt Raphidovirus mit HaV eine Zwischenstellung zwischen den beiden Zweigen der restlichen Phycodnaviridae und den erweiterten Mimiviridae ein, ohne selbst – anders als die „OLPG“-Mitglieder, AaV und TetV-1 – diesen anzugehören:[33]
| Phycodnaviridae-Mimiviridae-Klade |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
YSLPV = „Yellowstone Lake Phycodnavirus“
DSLPV = „Dishui Lake Phycodnavirus“
Das zweite Szenario würde die Mollivirus-Klade wie vorgeschlagen als Familie Molliviridae,[37][38][39] und die Pandoraviren wie vorgeschlagen ebenfalls als „Pandoraviridae“ erlauben.[40], die Chlorovirus-Klade wäre Phycodnaviridae s. s.. Unter Verwendung der vom ICTV im März 2020 mit der Master Species List #35 eingeführten Bezeichnungen kann im obigen Kladogramm ‚erweiterte Mimiviridae‘ durch die Ordnung Imitervirales und die ‚Phycodnaviridae-Mimiviridae-Klade‘ durch die Klasse Megaviricetes ersetzt werden. Dazu passt ein Vorschlag von Filée (2018), die Mimiviridae-Erweiterung — hier als Unterfamilie Mesomimivirinae eingestuft — stattdessen als Mesomomoviridae in den Rang einer Familie zu erheben. Diese wäre dann Schwestertaxon der Mimiviridae.[41]
Eine weitere alternative Sicht findet sich bei Johannessen et al. (2015), Fig. S2.[42]
Greiner et al. (2018) sehen YLPV2 (alias YSLPV2, Yellowstone Phycodnavirus 2) jedoch nicht in der Klade der Viren vom Chlorovirus-Typ.[43]
Forschungsgeschichte
Willie H. Wilson et al. an der Marine Biological Association (MBA) der University of East Anglia und des Plymouth Marine Laboratory (PML) beobachteten das Virus erstmals im Jahr 1999. Im Sommer 2005 untersuchten Forscher am Plymouth Marine Laboratory (wieder Willie Wilson et al.) und am Sanger Institute (M. T. G. Holden et al.) sequenzierten das Genom für den Stamm EhV-86, wobei 472 Proteingene gefunden wurden. Nach dem Genom ist daher EhV-86 ein Riesenvirus und das größte bis dato bekannte Meeresvirus.
Bei der ersten Untersuchung des Coccolithoviren-Genoms wurde eine Gensequenz entdeckt, die für die Produktion von Ceramid verantwortlich ist.[5] Ceramid ist ein kontrollierender Faktor für den Zelltod (Apoptose). Es wird angenommen, dass Coccolithovirus das Leben von Emiliania huxleyi verlängert, während es die Wirtszelle für seine Replikation benutzt. Dies ist eine einzigartige Fähigkeit, die bisher in keinem anderen viralen Genom zu sehen war.
Siehe auch
- Mimivirus – das prominenteste Riesenvirus
- Mycoplasma genitalium, Pelagibacter ubique – einige der kleinsten bekannten Bakterien
- Nanoarchaeum – das kleinste der bekannten Archaeen
- Parvovirus – das kleinste bekannte Virus
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Jean-Michel Claverie: Giant viruses in the oceans: the 4th Algal Virus Workshop In: Virology Journal. 2005.
- Sanger institute home for Emiliania huxleyi virus 86 (Memento des Originals vom 14. Juni 2006 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- Giantviruses.org top viruses by genome size
- Plymouth Marine Laboratory press release
- Niyaz Yoosuf, Natalya Yutin, Philippe Colson, Svetlana A. Shabalina, Isabelle Pagnier, Catherine Robert, Said Azza, Thomas Klose, Jimson Wong, Michael G. Rossmann, Bernard La Scola, Didier Raoult, Eugene V. Koonin: Related Giant Viruses in Distant Locations and Different Habitats: Acanthamoeba polyphaga moumouvirus Represents a Third Lineage of the Mimiviridae That Is Close to the Megavirus Lineage, in: Genome Biol Evol. 4(12), 4. Dezember 2012, S. 1324–1330, doi:10.1093/gbe/evs109, PMC 3542560 (freier Volltext), PMID 23221609
- Viral Zone. ExPASy, abgerufen am 19. Dezember 2015.
- ICTV: Virus Taxonomy. Abgerufen am 19. Dezember 2018.
- ICTV Master Species List 2018b.v2
- L. C. M. Mackinder, C. A. Worthy, G. Biggi, M. Hall, K. P. Ryan, A. Varsani, G. M. Harper, W. H. Wilson, C. Brownlee, D. C. Schroeder: A unicellular algal virus, Emilienia huxleyi virus 86, exploits an animal-like infection strategy. In: Journal of general Virology. 90, 2009, S. 2306–2316.
- D. C. Schroeder, J. Oke, M. Hall, G. Malin, W. H. Wilson: Viral succession observed during an Emilinaia huxleyi bloom. In: Applied and Environmental Microbiology. 69, 2003, S. 2484–2490, PMC 154549 (freier Volltext).
- W. H. Wilson, G. A. Tarran, D. Schroeder, M. Cox, J. Oke, G. Malin: Isolation of viruses responsible for the demise of an Emiliania huxleyi bloom in the English Channel. In: Journal of the Marine Biological Association of the United Kingdom. 82, 2002, S. 369–377.
- M. J. Frada, D. Schatz, V. Farstey, J. E. Ossolinski, H. Sabanay, S. Ben-Dor, I. Koren, A. Vardi: Zooplankton May Serve as Transmission Vectors for Viruses Infecting Algal Blooms in the Ocean. In: Current Biology. 24, 2014, S. 2592–2597.
- J. I. Nissimov, J. A. Napier, S. A. Kimmance, M. J. Allen: Permanent draft genomes of four new coccolithoviruses: EhV-18, EhV-145, EhV-156 and EhV-164. In: Marine Genomics. 15, 2014, S. 7–8.
- J. I. Nissimov, C. A. Worthy, P. Rooks, J. A. Napier, S. A. Kimmance, M. R. Henn, H. Ogata, M. J. Allen: Draft genome sequence of the Coccolithovirus EhV-84. In: Standards in Genomic Science. 5, 2011, S. 1–11.
- J. I. Nissimov, C. A. Worthy, P. Rooks, J. A. Napier, S. A. Kimmance, M. R. Henn, H. Ogata, M. J. Allen: Draft Genome Sequence of Four Coccolithopviruses: Emiliania huxleyi Virus EhV-88, EhV-201, EhV-207 and EhV-208. In: Journal of Virology, 86(5), 2012, S. 2896–2897, PMC 3302265 (freier Volltext).
- J. I. Nissimov, C. A. Worthy, P. Rooks, J. A. Napier, S. A. Kimmance, M. R. Henn, H. Ogata, M. J. Allen: Draft Genome Sequence of the Coccolithopvirus Emiliania huxleyi Virus 202. In: Journal of Virology. 86(4), 2012, S. 380–2381.
- J. I. Nissimov, C. A. Worthy, P. Rooks, J. A. Napier, S. A. Kimmance, M. R. Henn, H. Ogata, M. J. Allen: Draft Genomic Sequence of the Coccolithovirus Emiliania huxleyi Virus 203. In: Journal of Virology, 85(24), 2011, S. 13468–13469, PMC 3233127 (freier Volltext).
- W. H. Wilson, D. C. Schroeder, M. J. Allen, M. T. G. Holden, J. Parkhill, B. G. Barrell, C. Churcher, N. Hamlin, K. Mungall, H. Norbertczak, M. A. Quail, C. Price, E. Rabbinowitsch, D. Walker, M. Craigon, D. Roy, P. Ghazal: Complete Genome Sequence and Lytic Phase Transcription Profile of a Coccolithovirus. In: Science. Volume 309, Issue 5737, 2005, S. 1090–1092. doi:10.1126/science.1113109
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- A. Monier, A. Pagarete, C. De Vargas, M. J. Allen, B. Read, J. Claverie, H. Ogata, C. De Vargas: Horizontal gene transfer of an entire metabolic pathway between a eukaryotic alga and its DNA virus. In: Genome Research. 19, 2009, S. 1441–1449.
- Jean-Michel Claverie, Chantal Abergel: Mimiviridae: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes. In: Viruses. 2018 Sep; 10(9), 18. September 2018, S. 506, doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528, Tab. 2
- Julien Andreani, Jacques Y. B. Khalil, Emeline Baptiste, Issam Hasni, Caroline Michelle, Didier Raoult, Anthony Levasseur, Bernard La Scola: Orpheovirus IHUMI-LCC2: A New Virus among the Giant Viruses, in: Front. Microbiol., 22. Januar 2018, doi:10.3389/fmicb.2017.02643
- NCBI: unclassified Coccolithovirus (genus)
- NCBI: Coccolithovirus (genus)
- Graziele Oliveira, Bernard La Scola, Jônatas Abrahão: Giant virus vs amoeba: fight for supremacy, in: Virol J 16, 126, 4. November 2019, doi:10.1186/s12985-019-1244-3, PDF
- Ben A. Wagstaff, Iulia C. Vladu, J. Elaine Barclay, Declan C. Schroeder, Gill Malin, Robert A. Field: Isolation and Characterization of a Double Stranded DNA Megavirus Infecting the Toxin-Producing Haptophyte Prymnesium parvum, in: Viruses 2017, Band 9, Nr. 3, Special Issue Marine Viruses 2016, 40; doi:10.3390/v9030040
- Julien Guglielmini, Anthony C. Woo, Mart Krupovic, Patrick Forterre, Morgan Gaia: Diversification of giant and large eukaryotic dsDNnA viruses predated the origin of modern eukaryotes, in: PNAS, Band 116, Nr. 39, 10./24. September 2019, S. 19585–19592, doi:10.1073/pnas.1912006116, PMID 31506349, Fig. 2
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1007/s00705-016-2853-4
- Hao Chen, Weijia Zhang, Xiefei Li, Yingjie Pan, Shuling Yan, Yongjie Wang: The genome of a prasinoviruses-related freshwater virus reveals unusual diversity of phycodnaviruses, in: BMC Genomics, Dezember 2018, doi:10.1186/s12864-018-4432-4
- NCBI: Phycodnaviridae (family)
- Fumito Maruyama, Shoko Ueki: Evolution and Phylogeny of Large DNA Viruses, Mimiviridae and Phycodnaviridae Including Newly Characterized Heterosigma akashiwo Virus. In: Front. Microbiol. 30. November 2016, doi:10.3389/fmicb.2016.01942, PMC 5127864 (freier Volltext), PMID 27965659.
- Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses, in: F1000 Research. 22. November 2018, doi:10.12688/f1000research.16248.1, version 1.
- Michael J. Allen, Declan C. Schroeder, Matthew T. G. Holden, William H. Wilson: Evolutionary History of the Coccolithoviridae. In: Molecular Biology and Evolution. Volume 23, Issue 1, 1. Januar 2006, S. 86–92, doi:10.1093/molbev/msj010
- List of the main “giant” viruses known as of today (PDF; 334 kB) Centre National de la Recherche Scientifique, Université Aix Marseille, vom 18. April 2018.
- Neues Riesenvirus entdeckt - Vor Hawaii aufgespürtes Virus ist bisher größter Zellparasit pflanzlicher Organismen, auf: scinexx vom 4. Mai 2018
- Chantal Abergel: Giant viruses physiology. 24. März 2017, Laboratoire Information Génomique et Structurale (IGS), Institut de Microbiologie de la Méditerranée (IMM), Marseille, France
- Claire Bertelli, Linda Mueller, Vincent Thomas, Trestan Pillonel, Nicolas Jacquier, Gilbert Greub: Cedratvirus lausannensis – digging into Pithoviridae diversity. In: Environmental Microbiology. 19 (10), 2017, S. 4022–4034,doi:10.1111/1462-2920.13813
- Bruno Guigliarelli, Bénédicte Burlat: PhD proposal: Structure and functional role of a new iron-sulfur protein family in giant viruses. (PDF) Ecole Doctorale des Sciences Chimiques ED250, 2017.
- Natalya Yutin, Yuri I. Wolf, Eugene V. Koonin: Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. In: Virology, 0, Okt 2014, S. 38–52, PMID 25042053, doi:10.1016/j.virol.2014.06.032, PMC 4325995 (freier Volltext), siehe Supplement 04
- Jonathan Filée: Giant viruses and their mobile genetic elements: the molecular symbiosis hypothesis, in: Current Opinion in Virology, Band 33, Dezember 2018, S. 81–88; bioRxiv: 2018/04/11/299784 (Preprint-Volltext)
- Torill Vik Johannessen, Gunnar Bratbak, Aud Larsenb, Hiroyuki Ogatac, Elianne S.Egged, Bente Edvardsen, Wenche Eikremd, Ruth-Anne Sandaaa: Characterisation of three novel giant viruses reveals huge diversity among viruses infecting Prymnesiales (Haptophyta), in: Virology, Band 476, Februar 2015, S. 180–188, doi:10.1016/j.virol.2014.12.014, PMID 25546253, Fig. S2
- Timo Greiner, Anna Moroni, James L. Van Etten, Gerhard Thiel: Genes for Membrane Transport Proteins: Not So Rare in Viruses, in: MDPI Viruses Band 10, Nr. 9, Special Issue Algae Virus, 26 August 2018, 456; doi:10.3390/v10090456
Literatur
- M. J. Allen, D. C. Schroeder, M. T. Holden, W. H. Wilson: Evolutionary History of the Coccolithoviridae. In: Mol Biol Evol. 23, 2006, S. 86–92.
- E. Paasche: A review of the coccolithophorid Emiliania huxleyi (Prymnesiophyceae), with particular reference to growth, coccolith formation, and calcification-photosynthesis interactions. In: Phycologia. 40(6), 2001, S. 503–552. doi:10.2216/i0031-8884-40-6-503.1
- M. J. Allen, D. C. Schroeder, W. H. Wilson: Preliminary characterisation of repeat families in the genome of EhV-86, a giant algal virus that infects the marine microalga Emiliania huxleyi. In: Archives of Virology. 151, 2006, S. 525–535. doi:10.1007/s00705-005-0647-1
- M. J. Allen, T. Forster, D. C. Schroeder, M. Hall, D. Roy, P. Ghazal, W. H. Wilson: Locus specific gene expression pattern suggests a unique propagation strategy for a giant algal virus. In: Journal of Virology, 80, 2006, S. 7699–7705, PMC 1563701 (freier Volltext).
- M. J. Allen, J. A. Howard, K. S. Lilley, W. H. Wilson: Proteomic analysis of the EhV-86 virion. In: Proteome Science. 6(11), 2008. doi:10.1186/1477-5956-6-11
- Fumito Maruyama, Shoko Ueki: Evolution and Phylogeny of Large DNA Viruses, Mimiviridae and Phycodnaviridae Including Newly Characterized Heterosigma akashiwo Virus. In: Front. Microbiol. 30. November 2016. doi:10.3389/fmicb.2016.01942
- Dickson Kinyanyi, George Obiero, Peris W Amwayi, Stephen Mwaniki, Mark Wamalwa: In silico structural and functional prediction of African swine fever virus protein-B263R reveals features of a TATA-binding protein, in: PeerJ 6(4):e4396 (2018), doi:10.7717/peerj.4396, S. 13, insbes. Fig. 7
- Weijia Zhang, Jinglie Zhou, Taigang Liu, Yongxin Yu, Yingjie Pan, Shuling Yan, Yongjie Wang: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes, in: Scientific Reports volume 5, Artikel Nr. 15131, 2015, doi:10.1038/srep15131, Abstract