Phaeovirus
Phaeovirus ist eine Gattung von Viren in der Familie der Phycodnaviridae und gehört damit zum Phylum Nucleocytoviricota (alias Nucleocytoplasmic large DNA viruses, NCLDV).[1] Als natürliche Wirte dienen Algen der Ordnung Ectocarpales. Es gibt derzeit (Stand 19. Juni 2021) elf Spezies in dieser Gattung,[2] einschließlich der (ehemaligen) Typusspezies Ectocarpus siliculosus virus 1.[3][4][5] Der Wortbestandteil Phaeo- ist abgeleitet von griechisch φαιός phaiós: „dunkel“.
| Phaeovirus | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| Phaeovirus | ||||||||||||||||
| Links | ||||||||||||||||
|
Systematik
Innere Systematik
Das International Committee on Taxonomy of Viruses (ICTV) hat mit Stand November 2018 folgende Spezies anerkannt:
- Gattung: Phaeovirus
- Spezies: Ectocarpus siliculosus virus 1 (EsV-1, Typusspezies, Wirt Ectocarpus siliculosus, Ectocarpales: Ectocarpaceae)[6]
- Spezies: Ectocarpus siliculosus virus a (EsV-a, auch EsV-1a)
- Spezies: Ectocarpus fasciculatus virus a (EfV-a, Wirt Ectocarpus fasciculatus)
- Spezies: Feldmannia species virus (FLSV, FsV, FspV, Wirt Feldmannia sp., Ectocarpales: Acinetosporaceae)[7][8]
- Spezies: Feldmannia species virus a (FsV-a)
- Spezies: Feldmannia irregularis virus a (FirrV-a, auch FirrV-1)
- Spezies: Hincksia hinckiae virus a (HhV-a, auch HhV-1, Wirt Hincksia hinckiae, Ectocarpales: Acinetosporaceae)
- Spezies: Myriotrichia clavaeformis virus a (McV-a, auch McV-1, Wirt Myriotrichia clavaeformis, Ectocarpales: Chordariaceae)
- Spezies: Pilayella littoralis virus 1 (PlV-1, Wirt Pylaiella littoralis — sic! siehe WoRMS[9] und AlgaeBase,[10] Ectocarpales: Acinetosporaceae)
Eine Liste, die auch einzelne Stämme (Isolate) und Abkürzungen aufführt findet man bei ScienceDirect.[11] Oft – nicht immer – bezeichnet der Zusatz -a eine Spezies, -1 den zugehörigen Typus-Stamm (Isolat).
Äußere Systematik
Die folgende Systematik folgt Schulz et al. (2018) mit Korrekturen und Ergänzungen nach Hao et al. (2018):[12][13]
| Phycodnaviridae |
| ||||||||||||||||||||||||||||||||||||||||||||||||
YSLPV = „Yellowstone Lake Phycodnavirus“
DSLPV = „Dishui Lake Phycodnavirus“
Greiner et al. (2018) sehen YLPV2 (alias YSLPV2, Yellowstone Phycodnavirus 2) jedoch nicht in der Klade der Viren vom Chlorovirus-Typ.[16]
Aufbau
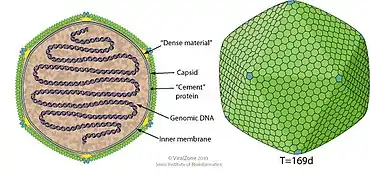
Die Virionen (Virusteilchen) der Phaeoviren haben eine Hülle mit ikosaedrischer oder sphärischer Geometrie mit der Triangulationszahl T=169. Der Durchmesser beträgt ca. 120–150 nm.
Genom
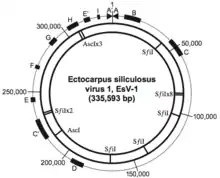
Das Genom ist linear und hat eine Länge von etwa 150–350 Kbp (Kilobasenpaaren).[3]
Bei der Typusspezies EsV-1 haben die Virionen einen Durchmesser von 200 nm, die Genomlänge beträgt 335.593 bp, es werden vorhergesagt 240 Proteine kodiert und der GC-Gehalt liegt bei 51,7 %.[17][18]
Vermehrungszyklus
Die Virusreplikation geschieht nukleo-cytoplasmatisch (vom Zellkern ins Zytoplasma). Das Virus verlässt die Wirtszelle durch Lyse vermittels lytischer Phospholipide. Die Übertragung geschieht durch passive Diffusion.[3]
Literatur
- Dickson Kinyanyi, George Obiero, Peris W Amwayi, Stephen Mwaniki, Mark Wamalwa: In silico structural and functional prediction of African swine fever virus protein-B263R reveals features of a TATA-binding protein, in: PeerJ 6(4):e4396 (2018), doi:10.7717/peerj.4396, S. 13, insbes. Fig. 7
- Weijia Zhang, Jinglie Zhou, Taigang Liu, Yongxin Yu, Yingjie Pan, Shuling Yan, Yongjie Wang: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes, in: Scientific Reports volume 5, Artikel Nr. 15131, 2015, doi:10.1038/srep15131, Abstract
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- ICTV: ICTV Master Species List 2020.v1, New MSL including all taxa updates since the 2019 release, March 2021 (MSL #36)
- Viral Zone. ExPASy. Abgerufen am 21. Dezember 2018.
- ICTV: Virus Taxonomy. Abgerufen am 22. Dezember 2015.
- ICTV Master Species List 2018a v1, ICTV MSL including all taxa updates since the 2017 release.
- Nicolas Delaroque, Wilhelm Boland: The genome of the brown alga Ectocarpus siliculosus contains a series of viral DNA pieces, suggesting an ancient association with large dsDNA viruses, in: BMC Evolutionary Biology, Band 8, Nr. 110, 12. April 2008, doi:10.1186/1471-2148-8-110, PMID 18405387, PMC 2373305 (freier Volltext) (mit Genomkarte) – CC Attribution License 2.0
- William H. Wilson, Ilana C. Gilg, Mohammad Moniruzzaman, Erin K. Field, Sergey Koren, Gary R. LeCleir, Joaquín Martínez Martínez, Nicole J. Poulton, Brandon K. Swan, Ramunas Stepanauskas, Steven W. Wilhelm: Genomic exploration of individual giant ocean viruses, in: ISME Journal, Band 11, Nr. 8, August 2017, S. 1736–1745, doi:10.1038/ismej.2017.61, PMC 5520044 (freier Volltext), PMID 28498373
- Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea, in: eLife 7, 27. März 2018, e33014, doi:10.7554/eLife.33014. Hier insbes.: Fig. 6 Supplement 1
- WoRMS: Pilayella littoralis (Linnaeus) Kjellman, 1872 (x Status: unaccepted), und Pylaiella littoralis (Linnaeus) Kjellman, 1872 (✔︎ Status: accepted)
- Guiry & Guiry: Pylaiella littoralis, AlgaeBase, World-wide electronic publication, National University of Ireland, Galway 2018 (Aufgerufen am 21. Dezember 2018)
- Phycodnaviridae: List of species in the genus Phaeovirus, Ninth Report of the International Committee on Taxonomy of Viruses, in: Virus Taxonomy (ScienceDirect), 2012, S. 249-262, doi:10.1016/B978-0-12-384684-6.00024-0
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses, in: Nature Communicationsvolume 9, Article number: 4881 (2018) vom 19. November 2018, doi:10.1007/s00705-016-2853-4
- Hao Chen, Weijia Zhang, Xiefei Li, Yingjie Pan, Shuling Yan, Yongjie Wang: The genome of a prasinoviruses-related freshwater virus reveals unusual diversity of phycodnaviruses, in: BMC Genomics, Dezember 2018, 19:49 (online 15. Januar 2018), doi:10.1186/s12864-018-4432-4
- Fumito Maruyama, Shoko Ueki: Evolution and Phylogeny of Large DNA Viruses, Mimiviridae and Phycodnaviridae Including Newly Characterized Heterosigma akashiwo Virus. In: Front. Microbiol. 30. November 2016, doi:10.3389/fmicb.2016.01942, PMC 5127864 (freier Volltext), PMID 27965659.
- Eugene V. Koonin, Natalya Yutin: Multiple evolutionary origins of giant viruses, in: F1000 Research. 22. November 2018, doi:10.12688/f1000research.16248.1, version 1.
- Timo Greiner, Anna Moroni, James L. Van Etten, Gerhard Thiel: Genes for Membrane Transport Proteins: Not So Rare in Viruses, in: MDPI Viruses Band 10, Nr. 9, Special Issue Algae Virus, 26 August 2018, 456; doi:10.3390/v10090456
- Jean-Michel Claverie, Chantal Abergel: Mimiviridae: An Expanding Family of Highly Diverse Large dsDNA Viruses Infecting a Wide Phylogenetic Range of Aquatic Eukaryotes]. In: Viruses. 2018 Sep; 10(9), 18. September 2018, S. 506, doi:10.3390/v10090506, PMC 6163669 (freier Volltext), PMID 30231528, Tab. 2
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)