Stille Mutation
Eine stille Mutation (synonym stumme Mutation) ist eine Mutation in einer codierenden Abfolge von Nukleinsäuren, die sich nicht auf die Proteinbiosynthese eines neu entstehenden Proteins auswirkt.[1]
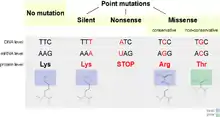
Eigenschaften
Eine stille Mutation ist meistens eine Punktmutation durch Substitution. Dabei wird ein Nukleotid gegen ein anderes getauscht. Das betroffene Codon wird dabei geändert, aber die codierte Aminosäure bleibt bei einer stillen Mutation gleich. Eine stille Mutation ohne Auswirkungen wird auch als neutrale Mutation bezeichnet. Eine stille Mutation in einem Exon wird als synonyme Mutation bezeichnet. Selbst eine nichtsynonyme Mutation kann eine stille Mutation sein, wenn die Auswirkung der Änderung einer codierten Aminosäure hinreichend gering ausfällt, d. h. wenn eine Aminosäure gegen eine ähnliche Aminosäure ausgetauscht wurde, die keinen Einfluss auf die Funktionsfähigkeit des Proteins hat.[2]
Allerdings ist nicht jede synonyme Mutation eine stille Mutation.[3][4][5] Eine synonyme Mutation kann die Sekundärstruktur einer mRNA ändern, die sich wiederum auf die Initiation und die Termination der Translation auswirkt.[6] Bei einer synonymen Mutation kann sich die Kinetik der Verwendung der verschiedenen Codons für die gleiche Aminosäure bei der Translation am Ribosom auf die Proteinfaltung und somit auf Sekundär- und Tertiärstruktur sowie die biologische Aktivität eines neu entstehenden Proteins auswirken und dadurch auch einen veränderten Phänotyp bewirken, ohne dass sich die Aminosäuresequenz ändert.[7][8][9][10][11] Manche Codons werden aufgrund der vergleichsweise langsamen Bindung von tRNA dann auch langsam während der Translation in eine Aminosäure übersetzt,[12] was sich entsprechend auf die bevorzugte Codonverwendung sowie auf die Sekundärstruktur und die Abbaustabilität von mRNA auswirkt.[13] Beispiele für Mutationen, die sich trotz korrekter Aminosäuresequenz auf den Phänotyp auswirken, sind das p-Glykoprotein (synonym MDR1)[12] und das CFTR,[14] wo es bei manchen synonymen Mutationen durch Verzögerungen oder veränderte RNA-Strukturen während der Translation zu einer fehlerhaften Proteinfaltung kommen kann. Daneben können in seltenen Fällen auch Mutationen in untranslatierten Regionen die Lesart eines Codons beeinflussen, ohne dass dieses verändert ist. So wirkt das Basentriplett UGA normalerweise als Stopcodon, kann aber in bestimmten Kontexten – abhängig von Sekundärstrukturen der mRNA – als Codon für Selenocystein interpretiert werden.
Anwendungen
Durch eine Codon-Optimierung kann die Genexpressionsrate gesteigert werden, indem nur diejenigen 20 Aminosäurecodons verwendet werden, die in der jeweiligen Art am stärksten exprimiert werden.[15] Eine gehäufte Verwendung suboptimaler Codons ist eine Methode zur Attenuierung von viralen Lebendimpfstoffen, die am Poliovirus demonstriert wurde.[16] Daneben werden stille Mutationen bei einer Klonierung eingeführt, um neue Restriktionsstellen für Restriktionsenzyme zu erzeugen.
Literatur
- Z. Zhang, M. A. Miteva, L. Wang, E. Alexov: Analyzing effects of naturally occurring missense mutations. In: Computational and mathematical methods in medicine. Band 2012, 2012, S. 805827, doi:10.1155/2012/805827, PMID 22577471, PMC 3346971 (freier Volltext).
Weblinks
- WatCut, ein Online-Tool zur Restriktionsanalyse, zur Suche nach stillen Mutationen und für RFLP-Simulationen.
Einzelnachweise
- Julia E. Richards: The Human Genome. Academic Press, 2010, ISBN 978-0-080-91865-5, S. 571.
- S. Teng, T. Madej, A. Panchenko, E. Alexov: Modeling effects of human single nucleotide polymorphisms on protein-protein interactions. In: Biophysical Journal. Band 96, Nummer 6, März 2009, S. 2178–2188, doi:10.1016/j.bpj.2008.12.3904, PMID 19289044, PMC 2717281 (freier Volltext).
- J. V. Chamary, J. L. Parmley, L. D. Hurst: Hearing silence: non-neutral evolution at synonymous sites in mammals. In: Nature Reviews Genetics. Band 7, Nummer 2, Februar 2006, S. 98–108, doi:10.1038/nrg1770, PMID 16418745.
- Patrick Goymer: Synonymous mutations break their silence. In: Nature Reviews Genetics. 8, 2007, S. 92, doi:10.1038/nrg2056.
- T. Zhou, E. A. Ko, W. Gu, I. Lim, H. Bang, J. H. Ko: Non-silent story on synonymous sites in voltage-gated ion channel genes. In: PLOS ONE. Band 7, Nummer 10, 2012, S. e48541, doi:10.1371/journal.pone.0048541, PMID 23119053, PMC 3485311 (freier Volltext).
- S. A. Shabalina, A. Y. Ogurtsov, N. A. Spiridonov: A periodic pattern of mRNA secondary structure created by the genetic code. In: Nucleic acids research. Band 34, Nummer 8, 2006, S. 2428–2437, doi:10.1093/nar/gkl287, PMID 16682450, PMC 1458515 (freier Volltext).
- Mary K. Campbell: Biochemistry. Cengage Learning, 2016, ISBN 978-1-337-51435-4, S. 391.
- Czech A, Fedyunin I, Zhang G, Ignatova Z: Silent mutations in sight: co-variations in tRNA abundance as a key to unravel consequences of silent mutations. In: Mol Biosyst. 6, Nr. 10, Oktober 2010, S. 1767–72. doi:10.1039/c004796c. PMID 20617253.
- Komar AA: Genetics. SNPs, silent but not invisible. In: Science. 315, Nr. 5811, Januar 2007, S. 466–7. doi:10.1126/science.1138239. PMID 17185559.
- Komar AA: Silent SNPs: impact on gene function and phenotype. In: Pharmacogenomics. 8, Nr. 8, August 2007, S. 1075–80. doi:10.2217/14622416.8.8.1075. PMID 17716239.
- Z. Zhang, M. A. Miteva, L. Wang, E. Alexov: Analyzing effects of naturally occurring missense mutations. In: Computational and mathematical methods in medicine. Band 2012, 2012, S. 805827, doi:10.1155/2012/805827, PMID 22577471, PMC 3346971 (freier Volltext).
- C. Kimchi-Sarfaty, J. M. Oh, I.-W. Kim, Z. E. Sauna, A. M. Calcagno, S. V. Ambudkar, M. M. Gottesman,: A "Silent" Polymorphism in the MDR1 Gene Changes Substrate Specificity. In: Science. 315, Nr. 5811, 26. Januar 2007, S. 525–8. doi:10.1126/science.1135308. PMID 17185560.
- Angov E: Codon usage: nature's roadmap to expression and folding of proteins. In: Biotechnol J. 6, Nr. 6, Juni 2011, S. 650–9. doi:10.1002/biot.201000332. PMID 21567958. PMC 3166658 (freier Volltext).
- R. Bartoszewski, J. Króliczewski, A. Piotrowski, A. J. Jasiecka, S. Bartoszewska, B. Vecchio-Pagan, L. Fu, A. Sobolewska, S. Matalon, G. R. Cutting, S. M. Rowe, J. F. Collawn: Codon bias and the folding dynamics of the cystic fibrosis transmembrane conductance regulator. In: Cellular & molecular biology letters. Band 21, 2016, S. 23, doi:10.1186/s11658-016-0025-x, PMID 28536625, PMC 5415761 (freier Volltext).
- E. Kotsopoulou, V. N. Kim, A. J. Kingsman, S. M. Kingsman, K. A. Mitrophanous: A Rev-independent human immunodeficiency virus type 1 (HIV-1)-based vector that exploits a codon-optimized HIV-1 gag-pol gene. In: J Virol. (2000), Bd. 74(10), S. 4839–52. PMID 10775623; PMC 112007 (freier Volltext).
- S. Mueller, J. R. Coleman, E. Wimmer: Putting synthesis into biology: a viral view of genetic engineering through de novo gene and genome synthesis. In: Chemistry & biology. Band 16, Nummer 3, März 2009, S. 337–347, doi:10.1016/j.chembiol.2009.03.002, PMID 19318214, PMC 2728443 (freier Volltext).