Pfannenstielstruktur
Die Pfannenstielstruktur (englisch panhandle structure) ist eine Sekundärstruktur bei Nukleinsäuren. Sie entsteht dadurch, dass sich die beiden Enden eines Nukleinsäure-Einzelstranges, dessen Basenfolgen sich terminal umgekehrt wiederholen (inverted terminal repeat, ITR), komplementär zusammenfügen. Die dazwischen liegenden nicht komplementären Abschnitte bleiben einzelsträngig.
Vorkommen
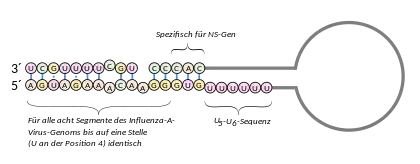
Die für Basenpaarung in Frage kommenden Nukleotide sind mit Linien verbunden dargestellt, die weniger stabile G-U-Paarung ist mit Punkten markiert. Die endständigen Nukleotide (zwölf am 3′-Ende, dreizehn am 5′-Ende) sind für alle acht RNA-Segmente identisch – bis auf eine Ausnahme: anstelle des Uridin-Nukleotids an der Position 4 befindet sich in einigen Genen ein Cytidin-Nukleotid. Die anschließenden drei Nukleotidpaare sind spezifisch für das NS-Gen. Daneben befindet sich das U5─U6 Terminations-/Polyadenylierungs-Signal.
Im Jahr 1987 durchgeführte Untersuchungen an Influenza-A-Viren[1] zeigten für deren genomische RNA eine zirkuläre Konformation mit einer ungefähr 15 Nukleotide langen stielförmigen Struktur der Enden. An jedem der acht RNA-Segmente sind die Sequenzen an den Enden über einen kurzen Bereich komplementär und hoch konserviert. Die Forscher fanden, dass diese Struktur im intrazellulären Pool nur während der Transkription vorkommt, jedoch nicht während der Assemblierung, also dem Zusammenbau der einzelnen Proteine und Nukleinsäuren zu einem fertigen Viruspartikel. Sie postulierten verschiedene Funktionen der Pfannenstielstruktur im viralen Replikationsprozess der Influenza-A-Viren: als Signal für das Umschalten zwischen Transkription und Replikation, als Terminationssignal bei der mRNA-Synthese und als Initiationsstelle für die Verpackung der Nukleoproteinkomplexe in den viralen Partikel.[1]
Terminale umgekehrte Wiederholungssequenzen wurden auch im Genom anderer negativsträngiger Viren (Negarnaviricota) einschließlich Arenaviren[2] und Bunyaviren[3][4] gefunden, bei denen ebenfalls zirkuläre Nukleinsäurestrukturen beobachtet wurden;[5][6][7] die Nukleotidfolge ermöglicht Pfannenstiellängen von 23 (PICV) bzw. 25 (LACV, SSHV) Basenpaaren.
Adenoviren besitzen ein doppelsträngiges DNA-Genom, in welchem nach Strangtrennung die entstehenden Einzelstränge sich in einer Pfannenstielstruktur konformieren.[8][9][10]
Siehe auch
Einzelnachweise
- M.T. Hsu, J.D. Parvin, S. Gupta, M. Krystal, P. Palese: Genomic RNAs of influenza viruses are held in a circular conformation in virions and in infected cells by a terminal panhandle. In: Proceedings of the National Academy of Sciences of the United States of America. Band 84, 1987, S. 8140─8144, doi:10.1073/pnas.84.22.8140.
- D.D. Auperin, V. Romanowski, M. Galinski, D.H. Bishop: Sequencing studies of pichinde arenavirus S RNA indicate a novel coding strategy, an ambisense viral S RNA. In: Journal of Virology. Band 52, 1984, S. 897─904, PMID 6492264.
- J.F. Obijeski, J. McCauley, J.J. Skehel: Nucleotide sequences at the terminal of La Crosse virus RNAs. In: Nucleic Acids Research. Band 8, 1980, S. 2431─2438, doi:10.1093/nar/8.11.2431.
- C.D. Cabradilla, B.P. Holloway, J.F. Obijeski: Molecular cloning and sequencing of the La Crosse virus S RNA. In: Virology. Band 128, 1983.
- A.C. Vezza, J.P. Clewley, G.P. Gard, N.Z. Abraham, R.W. Compans, D.H. Bishop: Virion RNA species of the arenaviruses Pichinde, Tacaribe, and Tamiami. In: Journal of Virology. Band 26, 1978, S. 485─497, doi:10.1128/JVI.26.2.485-497.1978.
- E.L. Palmer, J.F. Obijeski, P.A. Webb, K.M. Johnson: The circular, segmented nucleocapsid of an arenavirus-Tacaribe virus. In: The Journal of General Virology. Band 3, 1977, S. 541─545, doi:10.1099/0022-1317-36-3-541.
- R.F. Pettersson, C.H. von Bonsdorff: Ribonucleoproteins of Uukuniemi virus are circular. In: Journal of Virology. Band 15, 1975, S. 386─392, doi:10.1128/JVI.15.2.386-392.1975.
- P.A .Leegwater, R.F. Rombouts, P.C. van der Vliet: Adenovirus DNA replication in vitro: duplication of single-stranded DNA containing a panhandle structure. In: Biochimica et Biophysica Acta. Band 951, 1988, S. 403─410, doi:10.1016/0167-4781(88)90113-3.
- K. Wang, F.Y. Xu, K.G. Ahern, G.D. Pearson: Inverted repeats direct repair of adenovirus minichromosome ends. In: Virology. Band 183, 1991, doi:10.1016/0042-6822(91)90116-s.
- Viren mit doppelsträngigem Genom. In: S. Modrow, D. Falke, U. Truyen (Hrsg.): Molekulare Virologie. 2. Auflage. Spektrum, 2003, S. 513 ff.