Backbone (Biochemie)
In der Chemie, insbesondere der Biochemie, sowie der Molekularbiologie versteht man unter einem Backbone (englisch backbone „Rückgrat“), einer Hauptkette[1] oder einem Rückgrat[2] eine durchgehende Reihe kovalent gebundener Atome, die als Kette das „Rückgrat“ eines Makromoleküls bilden. Nicht zum Backbone gehören die daran gebundenen Molekülanteile, beispielsweise Seitenketten.
_2.png.webp)
Proteine
Bei Proteinen verläuft das Backbone über die Peptidbindungen, die die Aminosäuren zur Polypeptidkette verknüpfen:
- [H3N+–(CαR)–(C=O)–[NH–(CαRn)–(C=O)]n–O−

Die Torsionswinkel des Backbones von Proteinen werden mit φ, ψ und ω bezeichnet; erstere beiden werden im Ramachandran-Plot dargestellt. Die Torsionswinkel in den Seitenketten werden vom Backbone ausgehend mit χ1, χ2 usw. bezeichnet. Bei Darstellungen von Proteinen wird häufig nur der Verlauf des Backbones gezeigt, wobei Sekundärstrukturen oft verschiedenfarbig dargestellt werden (siehe Abbildung).
Nukleinsäuren
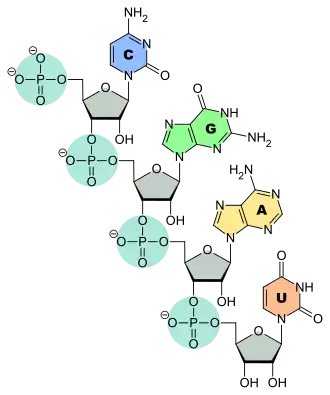
Bei Nukleinsäuren wird das Backbone von den alternierend verknüpften Phosphorsäure- und Pentose-Untereinheiten gebildet. An dieses Rückgrat sind die Nukleinbasen jeweils über die Pentose gebunden.
Bei Ribonukleinsäure (RNA) bilden Atome der Phosphorsäure- und der Ribose-Untereinheiten das Backbone.
Bei Desoxyribonukleinsäure (DNA) bilden Atome der Phosphorsäure- und Desoxyribose-Untereinheiten das Backbone. Das doppelsträngige DNA-Molekül besitzt im Unterschied zu anderen Biopolymeren zwei gegenläufige Backbones.
Polysaccharide
Alle Polysaccharide besitzen ein Backbone aus ihren Zucker-Untereinheiten. Bei unverzweigten Polysacchariden wie z. B. Cellulose trägt dieses Rückgrat keine Seitenketten.
Bei verzweigten Polysacchariden werden die Hauptkette des Rückgrats und die hieran gebundenen Seitenketten unterschieden:
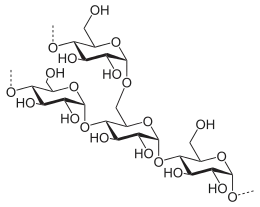
Amylopectin enthält eine Hauptkette aus 1,4-α-glycosidisch gebundenen D-Monosaccharid-Einheiten von Glucose und davon ausgehend an etwa jedem 25. Glucoserest eine α-1,6-glykosidisch verknüpfte Seitenkette.
Bei Galactoglucomannan besteht das Backbone aus 1,4-α-glycosidisch verknüpften D-Monosaccharid-Einheiten von Glucose und von Mannose, ungefähr im Verhältnis von ungefähr 2:1.[4][5] Diese Hauptkette nimmt analog zur Amylose der Stärke eine helikale Schraubenstruktur ein. An diese Hauptkette sind α-1,6-glycosidisch Monomere oder kurze Seitenketten aus Zuckerresten gebunden, welche etwa 8 % der Molekülmasse ausmachen. Damit liegt ein komplexes verzweigtkettiges Polymer vor, welches jedoch im Wesentlichen wie ein unverzweigtes erscheint (Zahlenangaben beziehen sich auf Glucomannan aus Konjak).[6] Die „Seitenketten“ bestehen überwiegend aus einzelnen Galactoseresten.[7]
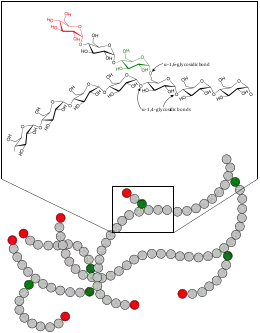
Im Glykogen sind Glucosebausteine α-1,4-glykosidisch verknüpft, jeder 8. bis 12. dieser Glucosereste tragen zusätzlich eine Seitenkette an einer 1,6-glycosidische Verknüpfung. Da hier „Seitenketten“ und „Hauptkette“ gleichermaßen aufgebaut sind, kann das Gesamtmolekül eher als Netz denn als Backbone betrachtet werden.
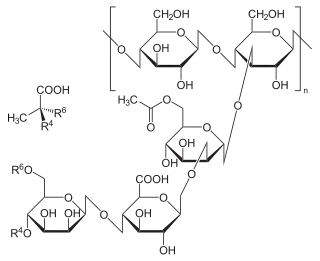
Bei Xanthan besteht das Backbone aus β-(1→4)-verknüpften D-Glucoseeinheiten. An jede zweite Glucoseeinheit ist α-(1→3)-glycosidisch eine β-D-Mannopyranosyl-(1→4)-β-D-glucuronopyranosyl-(1→2)-6-O-acetyl-α-D-mannopyranosyl-Seitenkette geknüpft.
Einzelnachweise
- R. Merkl & S. Waack: Bioinformatik Interaktiv. 2. Auflage, Wiley-VCH, Weinheim 2009, S. 13, ISBN 978-3-527-32594-8.
- Jeremy M. Berg, John L. Tymoczko, Lubert Stryer, Gregory J. Gatto jr.: Stryer Biochemie. Springer-Verlag, 2014, ISBN 978-3-8274-2988-9, S. 34.
- Hans-Dieter Jakubke und Hans Jeschkeit: Aminosäuren, Peptide, Proteine, Verlag Chemie, Weinheim, 1982, S. 96–97, ISBN 3-527-25892-2.
- Kaname Katsuraya, Kohsaku Okuyamab, Kenichi Hatanakab, Ryuichi Oshimab, Takaya Satoc, and Kei Matsuzakic (2003). Constitution of konjac glucomannan: chemical analysis and 13C NMR spectroscopy. In: Carbohydrate Polymers. 53 (2), S. 183–189; doi:10.1016/S0144-8617(03)00039-0.
- E. Gruber: Grundlagen der Zellstofftechnologie, Abb. 8 und 9.
- Eintrag zu (1-6)-alpha-glucomannan bei ChemicalBook, abgerufen am 30. Dezember 2011.
- Tea Hannuksela, Catherine Hervé du Penhoat: NMR structural determination of dissolved O-acetylated galactoglucomannan isolated from spruce thermomechanical pulp. In: Carbohydrate Research. Band 339, Nr. 2, Januar 2004, S. 301–312, doi:10.1016/j.carres.2003.10.025.