Pyrimidin-de-novo-Synthese
Unter der de-novo-Synthese von Pyrimidin versteht man die biochemische Synthese von Pyrimidin-Nukleotiden aus einfacheren Molekülen. Im Gegensatz zum salvage pathway werden hier keine Nukleotide oder Nukleotidderivate umgebaut und wiederverwertet, sondern unter höherem Energieaufwand neu aufgebaut. Bei der de-novo-Synthese entstehen Ribonukleotide, nicht aber Desoxyribonukleotide.
| Übergeordnet |
| Nukleosidsynthese Nukleotidsynthese |
| Untergeordnet |
| UMP-Synthese CMP-Synthese TMP-Synthese |
| Gene Ontology |
|---|
| QuickGO |
Der Ablauf der de-novo-Synthese ist hochkonserviert.[1]
Syntheseweg
Bei der de-novo-Synthese von Pyrimidin wird zuerst die Pyrimidinbase aufgebaut und dann auf ein Ribosephosphat aufgesetzt, so dass schließlich ein Pyrimidinnukleotid entsteht. Alle bis auf einen Schritt finden dabei im Cytosol statt.
Carbamoylphosphatsynthese (Schritt 1)
Hydrogencarbonat und Ammoniak bilden die Ausgangsstoffe, die vom Enzym Carbamoylphosphat-Synthetase II (CPS2, EC 6.3.5.5) unter Aufwendung zweier Moleküle ATP zu Carbamoylphosphat umgesetzt werden. Das Ammoniak stammt hierbei aus L-Glutamin.
Anbindung von Aspartat (Schritt 2)
Carbamoylphosphat (2) und die Aminosäure L-Aspartat (1) werden zu N-Carbamoylaspartat (3) umgesetzt, was eine Aspartat-Transcarbamoylase (ATCase, EC 2.1.3.2) katalysiert.
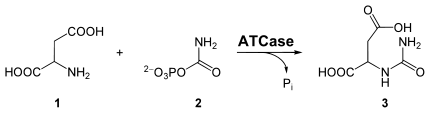
Ringschluss (Schritt 3)
Das Enzym Dihydroorotase (EC 3.5.2.3) katalysiert die intramolekulare Kondensation von Carbamoylaspartat zu Dihydroorotat (4)
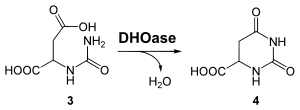
Oxidation zu Orotat (Schritt 4)
Das Enzym Dihydroorotat-Dehydrogenase (DHODH, EC 1.3.3.1) oxidiert Dihydroorotat zu Orotat (5), wobei NAD+ als Oxidationsmittel zu NADH reduziert wird.
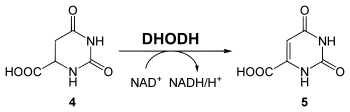
Ribosetransfer und Decarboxylierung (Schritt 5 und 6)
Die Übertragung auf eine Ribosegruppe erfolgt mit Hilfe der Orotat-Phosphoribosyltransferase (OPRTase EC 2.4.2.10). Dabei reagiert Orotat mit Phosphoribosylpyrophosphat (6) unter Abspaltung von Pyrophosphat zu Orotidinmonophosphat (OMP, 7). Dieses wiederum wird von Orotidin-5′-Phosphat-Decarboxylase (auch Orotidylat-Decarboxylase, EC 4.1.1.23) zu Uridinmonophosphat (UMP, 8) decarboxyliert.
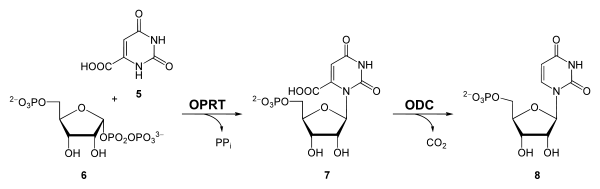
UMP ist das Ausgangsprodukt aller weiteren Pyrimidinnukleotide wie Uridindiphosphat (UDP), UDP-Glucose, Uridintriphosphat (UTP) und Cytidintriphosphat (CTP). Desoxyribonukleotide, Bausteine in der DNA, werden aus den entsprechenden Ribonukleotiden gewonnen.
Genomische Organisation in verschiedenen Spezies
Obwohl die de-novo-Synthese quasi ubiquitär ist, gibt es zwischen den Arten doch Unterschiede in der Organisation der Enzyme und der dafür codierenden Gene. Die Zahl der Gene, welche an den sechs enzymatischen Schritten beteiligt sind, nimmt von Prokaryoten zu Eukaryoten ab, jedoch nimmt die Komplexität der Enzyme in gleicher Weise zu. Es bilden sich sogenannte Clustergene, die für multifunktionelle Enzyme codieren.[2]
Prokaryoten
| Gen | Enzym | Name |
|---|---|---|
| pyrA | CPSase | Carbamoylphosphat-Synthetase |
| pyrB | ATCase | Aspartat-Transcarbamoylase |
| pyrC | DHOase | Dihydroorotase |
| pyrD | DHODH | Dihydroorotat-Dehydrogenase |
| pyrE | OPRTase | Orotat-Phosphoribosyltransferase |
| pyrF | ODCase | Orotidin-5′-Phosphat-Decarboxylase |
In Bakterien codieren sechs unterschiedliche Gene (pyrA-pyrF) für sechs unabhängige Enzyme.[3] Eine Besonderheit zeigt das Bakterium Escherichia coli. Seine dem Gen pyrA äquivalenten Gene carA und carB codieren für die beiden Untereinheiten des ersten Enzyms CPSase.
Niedere Eukaryoten
| Gen | Enzym | Name |
|---|---|---|
| ura2 | CPSase | Carbamoylphosphat-Synthetase |
| ura2 | ATCase | Aspartat-Transcarbamoylase |
| ura4 | DHOase | Dihydroorotase |
| ura1 | DHODH | Dihydroorotat-Dehydrogenase |
| ura5 ura10 | OPRTase | Orotat-Phosphoribosyltransferase |
| ura3 | ODCase | Orotidin-5′-Phosphat-Decarboxylase |
In Saccharomyces cerevisiae codiert das Gen ura2 für ein Enzym mit bifunktionaler CPSase-ATCase-Aktivität.[4] Dieses Enzym ließ sich außerdem aus Neurospora crassa[5] und Aspergillus[6] isolieren. Die folgenden vier enzymatischen Schritte werden codiert durch die eigenständigen Gene ura4 (DHOase), ura1 (DHODH), ura5;ura10 (OPRTase) und ura3 (ODCase).
Höhere Eukaryoten
| Gen | Enzym | Name |
|---|---|---|
| pyr1-3 cad | CPSase | Carbamoylphosphat-Synthetase II |
| pyr1-3 cad | ATCase | Aspartat-Transcarbamoylase |
| pyr1-3 cad | DHOase | Dihydroorotase |
| pyr4 | DHODH | Dihydroorotat-Dehydrogenase |
| pyr5-6 | OPRTase | Orotat-Phosphoribosyltransferase |
| pyr5-6 | ODCase | Orotidin-5′-Phosphat-Decarboxylase |
In höheren Eukaryoten, mit Ausnahme der Pflanzen, sind drei Strukturgene an der Pyrimidinsynthese beteiligt. Dabei codiert ein Clustergen pyr1-3 für ein multifunktionelles Polypeptid, welches für die ersten drei Syntheseschritte verantwortlich ist. In Säugern wird es cad genannt, abgeleitet von CPSase, ATCase und DHOase. Das zweite Gen pyr4 codiert für Dihydroorotat-Dehydrogenase (DHODH). Das dritte ist wiederum ein Clustergen pyr5-6 und codiert für ein Polypeptid, welches OPRTase- und ODCase-Aktivität besitzt.[7]
| Gen | Enzym | Name |
|---|---|---|
| carA/carB | CPSase | Carbamoylphosphat-Synthetase |
| pyrB | ATCase | Aspartat-Transcarbamoylase |
| pyrC | DHOase | Dihydroorotase |
| pyrD | DHODH | Dihydroorotat-Dehydrogenase |
| pyrE-F | OPRTase | Orotat-Phosphoribosyltransferase |
| pyrE-F | ODCase | Orotidin-5′-Phosphat-Decarboxylase |
Die Pyrimidinsynthese in Pflanzen zeigt auch eine Tendenz zur Bildung von Fusionsgenen, jedoch gibt es keinen Hinweis auf einen multifunktionellen Komplex, der mehr als einen der ersten vier enzymatischen Schritte katalysiert.[8] Die Gene werden bezeichnet als carA/carB (CPSase), pyrB (ATCase), pyrC (DHOase) und pyrD (DHODH). Die Umsetzung von Orotat zu UMP erfolgt in Pflanzen durch UMP-Synthase (pyrE-F), einem dimeren Polypeptid, dessen Monomere jeweils beide Enzymaktivitäten der OPRTase und ODCase haben.[9]
Bei Betrachtung des vollständig sequenzierten Genoms von Arabidopsis thaliana[10] fällt auf, dass für jedes Enzym in der de-novo-Synthese nur je ein Genlokus vorhanden ist.[11]
Einzelnachweise
- K. G. Wagner, A. I. Backer: Dynamics of nucleotides in plants studied on a cellular basis. In: Int. Rev. Cytol. 134, 1992, S. 184; doi:10.1016/S0074-7696(08)62027-6.
- M. Denis-Duphil: Pyrimidine biosynthesis in Saccharomyces cerevisiae: the ura2 cluster gene, its multifunctional enzyme product, and other structural or regulatory genes involved in de novo UMP synthesis. In: Biochem. Cell Biol. 67, Nr. 9, 1989, S. 612–631.
- G. A. O’Donovan, J. Neuhard: Pyrimidine metabolism in microorganisms. In: Bacteriol. Rev. 34, Nr. 3, 1970, 278–343.
- F. Lacroute: Regulation of pyrimidine biosynthesis in Saccharomyces cerevisiae. In: J. Bacteriol. 95, 1968 S. 824–832.
- L. Williams, S Bernhardt; Davis, R.: Copurification of pyrimidine-specific carbamyl phosphate synthetase and aspartate transcarbamylase of Neurospora crassa. In: Biochemistry. 9, Nr. 22, 1970, S. 4329–4335.
- L Palmer; Cove, D.: Pyrimidine biosynthesis in Aspergillus nidulans: isolation and preliminary characterisation of auxotrophic mutants. In: Mol Gen Genet. 138, Nr. 3, 1975, S. 243–255.
- Jones, 1980.
- M. Denis-Duphil: Pyrimidine biosynthesis in Saccharomyces cerevisiae: the ura2 cluster gene, its multifunctional enzyme product, and other structural or regulatory genes involved in de novo UMP synthesis. In: Biochem. Cell Biol. 67, Nr. 9, 1889, S. 612–631.
- Walther et al., 1984.
- Arabidopsis-Genome-Initiative, 2000.
- R. Boldt, R. Zrenner: Purine and pyrimidine biosynthesis in higher plants. In: Physiol Plant 117, Nr. 3, 2003, S. 297–304.