Pandoraviren
Die „Pandoraviren“ sind eine vorgeschlagene Gattung „Pandoravirus“ („PDV“)[3] beziehungsweise Familie „Pandoraviridae“ (von altgriechisch Πανδώρα Pandṓra, deutsch ‚Allgeberin‘, siehe auch Büchse der Pandora) von Riesenviren. Es handelt sich um DNA-Viren aus dem Phylum Nucleocytoviricota (veraltet Nucleocytoplasmic large DNA viruses, NCLDV). Das doppelsträngige DNA-Genom hat eine Größe von 1,9–2,5 Megabasenpaaren. Innerhalb dieser Gruppe scheinen die Pandoraviren nach Natalia Yutin, Eugene V. Koonin et al. stark abgewandelte Phycodnaviren zu sein, was möglicherweise eine Zuordnung zu dieser Virusfamilie (Phycodnaviridae) statt zu einer eigenen (per Vorschlag genannt „Pandoraviridae“)[4] favorisiert.[5][6] Pandoraviren besitzen unter den Viren das größte bekannte Genom.[7] Der Tropismus (das Wirtsspektrum) der Pandoraviren umfasst Amöben. Pandoraviren wurden in Deutschland jedoch bereits vor einigen Jahren gefunden, wobei allerdings erst 2013 molekularbiologisch bestätigt werden konnte, dass es sich tatsächlich um Pandoraviren handelt. Das Interessante hierbei ist, dass sie in Akanthamöben aus den Kontaktlinsenbehältern einer Keratitispatientin nachgewiesen wurden.[8]
| „Pandoraviren“ | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
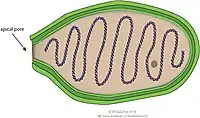
Schemazeichnung eines Virions von „Pandoravirus“ | ||||||||||||||||
| Systematik | ||||||||||||||||
| ||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||
| ||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||
| „Pandoravirus“ | ||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||
| „PDV“ | ||||||||||||||||
| Links | ||||||||||||||||
|
Struktur
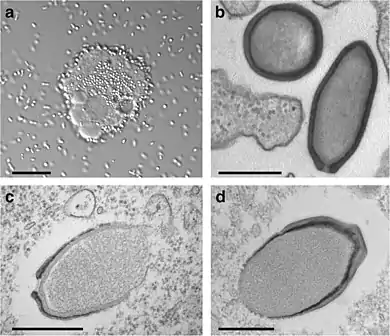
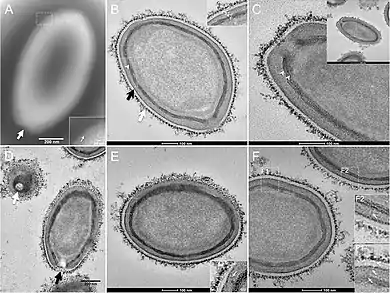
(a) Massenproduktion von „Pandoravirus macleodensis“-Nachkommen durch eine Amöbenzelle von Acanthamoeba castellanii vor der Zelllyse. Umweltbakterien aus der Probe sind im Kulturmedium zusammen mit „P. macleodensis“-Virionen zu sehen. (Maßstab 10 µm).
(b) TEM-Aufnahme eines Ultradünnschnitts einer A. castellanii-Zelle während der frühen Phase der Infektion durch „P. neocaledonia“. Die Pseudopodien (Pseudopodien) der Amöbe sind bereit, die umgebenden Virionen zu verschlingen. Zehn Minuten später ist dieser Vorgang abgeschlossen und die Virionen befinden sich in den Vakuolen (Maßstab 500 nm).
(c) TEM-Aufnahme eines Ultradünnschnitts einer A. castellanii-Zelle während des Zusammenbaus (Assemblierung) eines „P. salinus“-Virions (Maßstab 500 nm).
(d) TEM-Aufnahme eines Ultradünnschnitts eines naszierenden „P. quercus“-Virions. (Maßstab 500 nm).
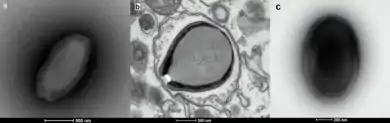
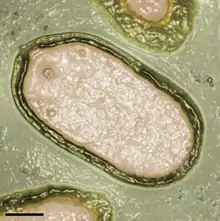
Das Virion (Virusteilchen) besitzt Ausmaße von etwa einem Mikrometer Länge und 0,7 Mikrometer Breite, wodurch sie zu den größten bekannten Viren gehören. Das Virion ist oval geformt und hat eine Öffnung an einem Ende. Pandoraviren sind etwa so groß wie ein kleineres Bakterium.
Genom
Das Genom von „Pandoravirus salinus“ hat eine Größe von 2.473.870 bp (Basenpaare) und kodiert vorhergesagt 1430 Proteine. Der GC-Gehalt liegt bei 62 %, bei „Pandoravirus dulcis“ ist die Größe 1.908.524 bp bei vorhergesagt 1070 kodierten Proteinen und einem GC-Gehalt von 64 %.[13] Das Erbgut von „Pandoravirus salinus“ besteht aus insgesamt 2.556 Genen, bei „Pandoravirus dulcis“ aus 1500 Genen. 93 Prozent der Gene dieser beiden Pandoraviren sind völlig fremdartig (ohne Homologie in Datenbanken). Es gibt Hinweise auf eine entfernte Verwandtschaft mit den Phycodnaviren: Pandoraviren haben wie das „Mollivirus“ offenbar einen gemeinsamen Vorfahren mit den Coccolithoviren innerhalb der Familie der Phycodnaviridae.[7][14]
Systematik
Äußere Systematik
Schulz et al. schlugen im November 2018 folgende Systematik vor:[14][15]
| Phycodnaviridae |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Das Kladogramm ist ergänzt nach Rolland et al. (2019).[17] Die basalen Vertreter der Gruppe sind noch in Diskussion, siehe Phycodnaviridae §Innere Systematik.
Innere Systematik
Weder die Gattung als solche, noch einzelne Spezies sind bisher vom International Committee on Taxonomy of Viruses (ICTV) offiziell anerkannt (Stand November 2018). Das NCBI kennt mit Stand Mai 2020 in der Gattung folgende Spezies:[18]
- Gattung „Pandoravirus“
- Spezies „P. braziliensis“
- Spezies „P. celtis“
- Spezies „P. dulcis“ (PVd, Erstbeschreibung/Typus)[19]
- Spezies „P. hades“
- Spezies „P. inopinatum“
- Spezies „P. kadiweu“
- Spezies „P. macleodensis“
- Spezies „P. massiliensis“
- Spezies „P. neocaledonia“
- Spezies „P. pampulha“[20] (siehe Pampulha-See)
- Spezies „P. persephone“
- Spezies „P. quercus“
- Spezies „P. salinus“ (PVs)[19]
- Spezies „P. tropicalis“ (siehe Pampulha-See)
Zu Details siehe auch Andrade (2019).[21]
Das folgende Kladogramm ist eine Konsensus der Vorschläge von Aherfi et al. (2018),[9] Legendre et al. (2018)[22] sowie CNRS (2018)[23] und (2019):[24]
| „Pandoravirus“ |
| |||||||||||||||||||||||||||||||||||||||
Literatur
- Nadège Philippe, Matthieu Legendre, Gabriel Doutre, Yohann Couté, Olivier Poirot, Magali Lescot, Defne Arslan, Virginie Seltzer, Lionel Bertaux, Christophe Bruley, Jérome Garin, Jean-Michel Claverie, Chantal Abergel: Pandoraviruses: Amoeba Viruses with Genomes Up to 2.5 Mb Reaching That of Parasitic Eukaryotes. In: Science, Band 341 (2013), Nr. 6143, S. 281–286, doi:10.1126/science.1239181.
- Dickson Kinyanyi, George Obiero, Peris W Amwayi, Stephen Mwaniki, Mark Wamalwa: In silico structural and functional prediction of African swine fever virus protein-B263R reveals features of a TATA-binding protein, in: PeerJ 6(4):e4396 (2018), doi:10.7717/peerj.4396, S. 13, insbes. Fig. 7.
- Weijia Zhang, Jinglie Zhou, Taigang Liu, Yongxin Yu, Yingjie Pan, Shuling Yan, Yongjie Wang: Four novel algal virus genomes discovered from Yellowstone Lake metagenomes, in: Scientific Reports volume 5, Artikel Nr. 15131, 2015, doi:10.1038/srep15131.
Weblinks
- Forscher entdecken rätselhafte Riesenviren. scinexx
- Forschung Aktuell. Deutschlandfunk
- Ana Cláudia dos S. P. Andrade, Thalita S. Arantes, Rodrigo A. L. Rodrigues, Talita B. Machado, Fábio P. Dornas, Melissa F. Landell, Cinthia Furst, Luiz G. A. Borges, Lara A. L. Dutra, Gabriel Almeida, Giliane de S. Trindade, Ivan Bergier, Walter Abrahão, Iara A. Borges, Juliana R. Cortines, Danilo B. de Oliveira, Erna G. Kroon, Jônatas S. Abrahão: Ubiquitous giants: a plethora of giant viruses found in Brazil and Antarctica, in: Virology Journal, Band 15, Nr. 22, 24. Januar 2018, doi:10.1186/s12985-018-0930-x
Anmerkungen
- Das Material wurde von dieser Quelle kopiert, die unter einer Creative Commons Attribution 4.0 International License verfügbar ist.
Einzelnachweise
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- Eugene V. Koonin, Natalya Yutin: Evolution of the Large Nucleocytoplasmatic DNA Viruses of Eukaryotes and Convergent Origins of Viral Gigantism. In: Advances in Virus Research, Band 103, AP 21. Januar 2019, S. 167–202, doi:10.1016/bs.aivir.2018.09.002.
-
Gabriel Augusto Pires de Souza, Victória Fulgêncio Queiroz, Maurício Teixeira Lima, Erik Vinicius de Sousa Reis, Luiz Felipe Leomil Coelho, Jônatas Santos Abrahão: Virus goes viral: an educational kit for virology classes, in: Virology Journal, Band 17, Nr. 13, 31. Januar 2020, doi:10.1186/s12985-020-1291-9

- Lucie Gallot-Lavallée, Guillaume Blanc, Jean-Michel Claverie; Grant McFadden (Hrsg.): Comparative Genomics of Chrysochromulina Ericina Virus and Other Microalga-Infecting Large DNA Viruses Highlights Their Intricate Evolutionary Relationship with the Established Mimiviridae Family. In: Journal of Virology, American Society for Microbiology (2017), doi:10.1128/JVI.00230-17.
- Natalya Yutin, Eugene V, Koonin: Pandoraviruses are highly derived phycodnaviruses. In: Biology Direct, 2013 8, S. 25, doi:10.1186/1745-6150-8-25
- Natalya Yutin, Yuri I. Wolf, Eugene V. Koonin: Origin of giant viruses from smaller DNA viruses not from a fourth domain of cellular life. In: Virology. Oktober 2014; S. 38–52, PMC 4325995 (freier Volltext), doi:10.1016/j.virol.2014.06.032, PMID 25042053, siehe Supplement 04
- Natalya Yutin, Eugene V. Koonin: Pandoraviruses are highly derived phycodnaviruses. In: Biology Direct. 8, Oktober 2013, S. 25. doi:10.1186/1745-6150-8-25. PMID 24148757. PMC 3924356 (freier Volltext).
- P. Scheid, B. Hauröder, R. Michel: Investigations of an extraordinary endocytobiont in Acanthamoeba sp.: development and replication. In: Parasitol Res. Band 106, Nr. 6, 2010, S. 1371–1377. doi:10.1007/s00436-010-1811-4.
- Sarah Aherfi, Julien Andreani, Emeline Baptiste, Amina Oumessoum, Fábio P. Dornas, Ana Claudia dos S. P. Andrade, Eric Chabriere, Jônatas Abrahão, Anthony Levasseur, Didier Raoult, Bernard La Scola, Philippe Colson: A Large Open Pangenome and a Small Core Genome for Giant Pandoraviruses. In: Front Microbiol., 9, 10. Juli 2018, S. 1486, doi:10.3389/fmicb.2018.01486, PMC 6048876 (freier Volltext), PMID 30042742.

-
Matthieu Legendre, Elisabeth Fabre, Olivier Poirot, Sandra Jeudy, Audrey Lartigue, Jean-Marie Alempic, Laure Beucher, Nadège Philippe, Lionel Bertaux, Karine Labadie, Yohann Couté, Chantal Abergel, Jean-Michel Claverie: Diversity and evolution of the emerging Pandoraviridae family, in: Nature communications, Band 9, Nr. 2285, 11. Juni 2018, doi:10.1038/s41467-018-04698-4.
 Preprint, auf: CSH Lab. bioRxiv, doi:10.1101/230904
Preprint, auf: CSH Lab. bioRxiv, doi:10.1101/230904
-
Vincent Racaniello: Pandoravirus, bigger and unlike anything seen before, auf: virology blog vom 1. August 2013.
 . Siehe auch:
. Siehe auch:
- Pandoravirus : découverte d’un chaînon manquant entre le monde viral et le monde cellulaire, INSERM (Salle de presse) Technologie pour la sante, Communiqués et dossiers de presse (Pressemitteilung) vom 19. Juli 2013 (französisch)
-
Djamal Brahim Belhaouari, Jean-Pierre Baudoin, Franck Gnankou, Fabrizio Di Pinto, Philippe Colson, Sarah Aherfi, Bernard La Scola: Evidence of a Cellulosic Layer in Pandoravirus massiliensis Tegument and the Mystery of the Genetic Support of Its Biosynthesis, in: Front. Microbiol. 10:2932, 20. Dezember 2019, doi:10.3389/fmicb.2019.02932.

- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Frederik Schulz, Lauren Alteio, Danielle Goudeau, Elizabeth M. Ryan, Feiqiao B. Yu, Rex R. Malmstrom, Jeffrey Blanchard, Tanja Woyke: Hidden diversity of soil giant viruses. In: Nature Communications, volume 9, 19. November 2018, Article number: 4881 (2018), doi:10.1038/s41467-018-07335-2.

- Die Gattungen Prymnesiovirus und Raphidovirus sind in dieser Arbeit nicht berücksichtigt, Phaeovirus ist als Phaevirus verschrieben. Mit YLPV scheinen „Yellowstone Phycodnavirus“ YSLPV 1 bis 3 gemeint zu sein, die Schreibweise „Yellow Lake Phycodnavirus“ (Ylpv-A, Ylpv-B) findet sich sonst nur bei Kinyanyi et al. (2018). Zur Klärung siehe Zhang et al. (2015)
- Korrigiert, siehe Phycodnaviridae §Systematik
- Clara Rolland, Julien Andreani, Amina Cherif Louazani, Sarah Aherfi, Rania Francis, Rodrigo Rodrigues, Ludmila Santos Silva, Dehia Sahmi, Said Mougari, Nisrine Chelkha, Meriem Bekliz, Lorena Silva, Felipe Assis, Fábio Dornas, Jacques Yaacoub Bou Khalil, Isabelle Pagnier, Christelle Desnues, Anthony Levasseur, Philippe Colson, Jônatas Abrahão, Bernard La Scola: Discovery and Further Studies on Giant Viruses at the IHU Mediterranee Infection That Modified the Perception of the Virosphere, in: Viruses 11(4), März/April 2019, pii: E312, doi:10.3390/v11040312, PMC 6520786 (freier Volltext), PMID 30935049, Fig. 2a
- NCBI: Pandoravirus (genus)
- Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea, in: eLife 7, 27. März 2018, e33014, doi:10.7554/eLife.33014. Hier insbes.: Fig. 6 Supplement 1
- NCBI: Pandoravirus pampulha (species)
- Ana Cláudia dos Santos Pereira Andrade, Paulo Victor de Miranda Boratto, Rodrigo Araújo Lima Rodrigues, Talita Machado Bastos, Bruna Luiza Azevedo, Fábio Pio Dornas, Danilo Bretas Oliveira, Betânia Paiva Drumond, Erna Geessien Kroon, Jônatas Santos Abrahão; Rozanne M. Sandri-Goldin (Hrsg.): New Isolates of Pandoraviruses: Contribution to the Study of Replication Cycle Steps. In: Journal of Virology. Band 93, Nr. 5, Februar 2019, e01942-18 doi:10.1128/JVI.01942-18.
- Matthieu Legendre, Elisabeth Fabre, Olivier Poirot, Sandra Jeudy, Audrey Lartigue, Jean-Marie Alempic, Laure Beucher, Nadège Philippe, Lionel Bertaux, Eugène Christo-Foroux, Karine Labadie, Yohann Couté, Chantal Abergel, Jean-Michel Claverie: Diversity and evolution of the emerging Pandoraviridae family. In: Nature Communications, Band 9, Nr. 1, 11. Juni 2018, doi:10.1038/s41467-018-04698-4
- List of the main “giant” viruses known as of today. (PDF; 334 kB) Université Aix Marseille, Centre national de la recherche scientifique, 18. April 2018.
- Centre national de la recherche scientifique: List of the main “giant” viruses known as of today (March 2019), Université Aix Marseille, März 2019.
- Pandoravirus dulcis. In: NCBI Taxonomy Browser. 1349409.
- Pandoravirus salinus. In: NCBI Taxonomy Browser. 1349410.