Formylglycin-generierendes Enzym
Formylglycin-generierendes Enzym (FGE) ist ein Enzym aus der Gruppe der Oxygenasen. Es verändert posttranslational ein Cystein in Sulfatasen in ein Formylglycin. FGE kann sowohl in Pro- als auch in Eukaryoten vorkommen.
| Formylglycin-generierendes Enzym | ||
|---|---|---|
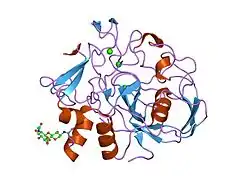 | ||
| C336S-Mutante | ||
| Andere Namen |
FGE, Sulfatase modifying Factor 1, UNQ3037, AAPA3037 | |
| Masse/Länge Primärstruktur | 374 Aminosäuren, 40.556 Da | |
| Bezeichner | ||
| Externe IDs | ||
| Enzymklassifikation | ||
| EC, Kategorie | 1.8.3.7 | |
| Orthologe (Mensch) | ||
| Entrez | 285362 | |
| Ensembl | ENSG00000144455 | |
| UniProt | Q8NBK3 | |
| Refseq (mRNA) | NM_001164674.1 | |
| Refseq (Protein) | NP_001158146.1 | |
| Genlocus | Chr 3: 4.03 – 4.47 Mb | |
| PubMed-Suche | 285362 | |
Struktur
Das aktive Enzym ist ein Monomer mit zwei Proteindomänen. Die Sekundärstruktur besteht aus α-Helices und β-Faltblättern, welche durch Calciumionen stabilisiert werden. Es hat drei Disulfidbrücken.[1] Die Aktivierung erfolgt durch die Abspaltung eines Signalpeptids nach der Beförderung ins endoplasmatische Retikulum.
Funktionsweise
Das FGE wird durch das Gen SUMF1 kodiert. Zu dem Zeitpunkt der Reaktion ist die Sulfatase noch nicht gefaltet. FGE erkennt die kurze Sequenz CxPxR im aktiven Zentrum von Sulfatasen und xxxLTGR als Hilfssequenz. Das Cystein in dieser Sequenz wird posttranslational in Formylglycin umgewandelt. Dadurch werden Sulfatasen aktiviert. FGE ist eine Oxygenase, welche Kupfer als Kofaktor besitzt. An der Reaktion von Cystein zu Formylglycin ist ein Cystein an der Position 341 im FGE beteiligt, welches sich an das zu verändernde Cystein bindet.[2] Aus dem Cystein wird durch Oxidation mit molekularem Sauerstoff und einem nicht identifizierten Reduktionsmittel die Reaktion zum 3-Oxoalanin (auch Formylglycin (fGly) genannt) an Zielproteinen katalysiert.[3][4]
Die 3-Oxoalanin-Modifikation, tritt bei der posttranslationalen Modifikation von Arylsulfatasen und einigen alkalischen Phosphatasen auf, die die hydratisierte Form von 3-Oxoalanin als katalytisches Nukleophil verwenden.[5][6] Substrate für FGE sind unter anderem N-Acetylgalactosamin-6-sulfat-Sulfatase (GALNS), Arylsulfatase A (ARSA), Steroid-Sulfatase (STS) und Arylsulfatase E (ARSE).
Vorkommen
FGE kommt im endoplasmatischen Retikulum von Eukaryoten vor. Auch Prokaryoten weisen FGE auf, wobei es bei diesen das zusätzliche System AtsB gibt.[7]
Defekt
Bei Mutationen im SUMF1-Gen, kommt es zur Inaktivierung des FGE. Dadurch können Sulfatasen, welche Sulfat-Ester hydrolysieren, nicht mehr aktiviert werden. Es kommt zur multiplen Sulfatasendefizienz.[8]
Einzelnachweise
- Thomas Dierks, Achim Dickmanns, Andrea Preusser-Kunze, Bernhard Schmidt, Malaiyalam Mariappan: Molecular Basis for Multiple Sulfatase Deficiency and Mechanism for Formylglycine Generation of the Human Formylglycine-Generating Enzyme. In: Cell. Band 121, Nr. 4, 20. Mai 2005, ISSN 0092-8674, S. 541–552, doi:10.1016/j.cell.2005.03.001, PMID 15907468.
- P. G. Holder, L. C. Jones, P. M. Drake, R. M. Barfield, S. Bañas, G. W. de Hart, J. Baker, D. Rabuka: Reconstitution of Formylglycine-generating Enzyme with Copper(II) for Aldehyde Tag Conversion. In: Journal of Biological Chemistry. Band 290, Nummer 25, Juni 2015, S. 15730–15745, doi:10.1074/jbc.M115.652669, PMID 25931126, PMC 4505483 (freier Volltext).
- M. P. Cosma, S. Pepe, I. Annunziata, R. F. Newbold, M. Grompe, G. Parenti, A. Ballabio: The multiple sulfatase deficiency gene encodes an essential and limiting factor for the activity of sulfatases. In: Cell. Band 113, Nummer 4, Mai 2003, S. 445–456, doi:10.1016/s0092-8674(03)00348-9, PMID 12757706.
- A. Preusser-Kunze, M. Mariappan, B. Schmidt, S. L. Gande, K. Mutenda, D. Wenzel, K. von Figura, T. Dierks: Molecular characterization of the human Calpha-formylglycine-generating enzyme. In: Journal of Biological Chemistry. Band 280, Nummer 15, April 2005, S. 14900–14910, doi:10.1074/jbc.M413383200, PMID 15657036.
- M. P. Cosma, S. Pepe, I. Annunziata, R. F. Newbold, M. Grompe, G. Parenti, A. Ballabio: The multiple sulfatase deficiency gene encodes an essential and limiting factor for the activity of sulfatases. In: Cell. Band 113, Nummer 4, Mai 2003, S. 445–456, doi:10.1016/s0092-8674(03)00348-9, PMID 12757706.
- A. Preusser-Kunze, M. Mariappan, B. Schmidt, S. L. Gande, K. Mutenda, D. Wenzel, K. von Figura, T. Dierks: Molecular characterization of the human Calpha-formylglycine-generating enzyme. In: Journal of Biological Chemistry. Band 280, Nummer 15, April 2005, S. 14900–14910, doi:10.1074/jbc.M413383200, PMID 15657036.
- Qinghua Fang, Jianhe Peng, Thomas Dierks: Post-translational Formylglycine Modification of Bacterial Sulfatases by the Radical S-Adenosylmethionine Protein AtsB. In: Journal of Biological Chemistry. Band 279, Nr. 15, 9. April 2004, ISSN 0021-9258, S. 14570–14578, doi:10.1074/jbc.M313855200, PMID 14749327 (jbc.org [abgerufen am 19. November 2019]).
- Maria Pia Cosma, Stefano Pepe, Giancarlo Parenti, Carmine Settembre, Ida Annunziata: Molecular and functional analysis of SUMF1 mutations in multiple sulfatase deficiency. In: Human Mutation. Band 23, Nr. 6, 2004, ISSN 1098-1004, S. 576–581, doi:10.1002/humu.20040.