Evolutionäre Entwicklungsbiologie
Die evolutionäre Entwicklungsbiologie oder kurz Evo-Devo (abgeleitet vom englischen Begriff evolutionary developmental biology) ist eine Forschungsrichtung der Biologie, die untersucht, wie sich die Steuerung der Individualentwicklung der Lebewesen (Ontogenese) in der Evolutionsgeschichte entwickelt hat.
Obwohl die evolutionäre Entwicklungsbiologie bereits in der Theoriebildung des 19. Jahrhunderts eine bedeutende Rolle spielte, entstand eine nennenswerte experimentelle Basis für eine begründete Weiterentwicklung der Theorie erst ab den 1980er Jahren mit der zunehmenden Aufklärung der Embryonalentwicklung durch die Entdeckung von Steuer-Genen und den Wirkmechanismen ihrer Produkte.
Seither wird mit entwicklungsbiologischen und molekularbiologischen Labor-Methoden versucht zu ermitteln, welche Faktoren und Steuerungsmechanismen für die Ausbildung von Geweben und Organen verantwortlich sind. Sachlich damit verknüpft ist die Frage, wie diese Steuerung als Ergebnis des Verlaufs der Stammesgeschichte der Organismen rekonstruiert werden kann. Auf der theoretischen, wie auch der experimentellen Ebene findet daher zwangsläufig eine Integration von Entwicklungsbiologie und Evolutionsbiologie statt.
Eine wesentliche Erkenntnisquelle ist dabei die Entschlüsselung der genetischen Basis für zahlreiche bis in die 1980er Jahre vollkommen rätselhafte Entwicklungsvorgänge, welche mit der Entdeckung der sog. Hox-Gene begonnen hatte.
Geschichte der Erforschung evolutionärer Veränderungen in der Ontogenese
Darwin und das 19. Jahrhundert
Zur Zeit von Charles Darwin war es weder möglich, die Entwicklungsprozesse genauer zu untersuchen, noch waren der genaue Mechanismus der Vererbung oder Gene und DNA bekannt.
Aus dem 19. Jahrhundert sind neben Darwin einige Embryologen zu nennen, die ebenfalls evolutionäre Gesichtspunkte behandelten:
- Karl Ernst von Baer stellte an Wirbeltieren fest, dass Embryonen verschiedener Arten umso schwieriger unterscheidbar sind, je früher in ihrer Entwicklung sie angetroffen werden (Baer-Regel)
- Fritz Müller kombinierte in seinem Buch Für Darwin (1864) natürliche Selektion und Embryologie und demonstrierte an Entwicklungsphasen von Krebstieren, dass ihre Stammesgeschichte ohne Darwins Theorie nicht erklärt werden könne.
- Ernst Haeckel. Auf ihn geht die heute nicht mehr gebräuchliche biogenetische Grundregel zurück, die in Schärfung eines damals weit verbreiteten Rekapitulationsgedankens angibt, dass die beobachteten Parallelen zwischen Ontogenese und Phylogenese der Organismen auf der embryonalen Wiederholung von Merkmalen beruht, die in der Stammesgeschichte der Arten schon im Erwachsenen-Stadium ausgebildet waren.
- Wilhelm Roux war Schüler von Haeckel und Begründer der Entwicklungsmechanik. Er war bereits der Ansicht, dass kein fertiger Bauplan vererbt wird (Präformationstheorie), sondern dass „den einzelnen Zellen ein gewisser Spielraum bleibt, innerhalb dessen sich das Geschehen gegenseitig selbst reguliert“ (1881). Aus diesem Gedanken wurden epigenetische Vorstellungen bestärkt, wie sie zuvor schon von Caspar Friedrich Wolff (1734–1794) angenommen wurden.
Synthetische Evolutionstheorie seit 1930
In den letzten Jahrzehnten des 19. und den ersten des 20. Jahrhunderts wurden Darwins Erkenntnisse in die neue Fachrichtung der vergleichenden Embryologie aufgenommen. Entdeckungen wie diejenige der Keimblätter (Endo-, Meso-, Ektoderm) waren wesentlich für die Entschlüsselung der Homologie der Körperbaupläne. Nach den großen Entdeckungen der Anfangszeit erlahmte der Elan aber dadurch, dass zwischen renommierten Forschern Meinungsverschiedenheiten über die Bedeutung zahlreicher Einzelheiten ausbrachen, die mit den damaligen Methoden nicht entscheidbar waren. Das Hauptinteresse der Forschung wandte sich daraufhin neuen Disziplinen wie der Entwicklungsmechanik und der Genetik zu.[1][2]
Auch während des Entstehens der Synthetischen Evolutionstheorie in den 1930er und 1940er Jahren gab es vereinzelt Wissenschaftler, die sich um eine stärkere Thematisierung der Entwicklung bemühten (z. B. Richard Goldschmidt, Conrad Hal Waddington, Iwan Iwanowitsch Schmalhausen). Die Synthetische Evolutionstheorie war jedoch mit dem dominierenden Fundament der Populationsgenetik (Ronald Aylmer Fisher, Sewall Wright, J. B. S. Haldane) unter der Mithilfe anderer Disziplinen (u. a. Zoologie, Systematik: Ernst Mayr) stark auf statistisch-deskriptives Denken ausgerichtet, so dass Prinzipien der Individualentwicklung keine Aufnahme in den Kanon fanden. Thomas Hunt Morgan, einer der frühen Vertreter der Synthetischen Evolutionstheorie, selbst auch Embryologe, stellte 1932 die Behauptung auf, die Genetik sei der einzige wissenschaftlich gültige Ansatz für das Studium der Evolution.[3] Vorstellungen, die sich mit der direkten Wirkung von Umwelteinflüssen auf den entstehenden Organismus befassten (Epigenetik), wurden nicht weiter verfolgt, weil diese dem neodarwinistischen Dogma widersprachen, wonach kein Informationsfluss möglich ist, der von außen auf die DNA wirkt und sie vererbbar verändert (Weismann-Barriere). Vor diesem Hintergrund kann verstanden werden, dass ein Forscher wie Conrad Hal Waddington, der 1942 eine Umweltinduzierung von Entwicklungsveränderungen und die Kanalisierung von Entwicklungsprozessen theoretisch beschrieb und deswegen als ein wichtiger Vorläufer der Evolutionären Entwicklungsbiologie gilt, seitens der Synthetischen Evolutionstheorie nicht beachtet wurde. Waddingtons Thesen gelangten erst in den vergangenen zwei Jahrzehnten zu einer Renaissance.[4][5]
Die Synthetische Evolutionstheorie, das Standardmodell der heutigen, auf Darwin zurückgehenden Evolutionstheorie, sieht die Abfolge von zufälligen und systematischen (bei sexueller Reproduktion: Rekombination) Variationen, natürlicher Selektion und resultierender Adaption von Populationen als ausreichend an, um die Entstehung organismischer Vielfalt zu erklären.[6] Ihre Vertreter vorrangig der 1930er bis 1950er Jahre nahmen dabei im Vergleich zu Darwin zum Teil restriktive Einschränkungen vor, die sich aus der gerade neu entdeckten Genetik ergaben. Es gibt folgende Grundannahmen der Synthetischen Theorie:
- Gradualismus ist die Annahme, dass sich evolutionäre Entwicklungen stets in kontinuierlichen, kleinen Veränderungen vollziehen, die sich zu größeren summieren.[7] (das Konzept des Gradualismus geht dabei ursprünglich auf Charles Darwin selbst zurück).
- Die Weismann-Barriere. Demnach besteht keine Möglichkeit einer vererbbaren Beeinflussung des Genoms bzw. der Keimzellen durch individuelle Erfahrung der Organismen.
Constraints und Heterochronie
Seit den achtziger Jahren mehrten sich die Stimmen, die für eine stärkere Beachtung der Entwicklung für die Evolution plädierten (Stephen Jay Gould u. a.): Man untersuchte verstärkt die entwicklungsbiologischen Beschränkungen, die das Spektrum der evolutionären Variation begrenzen (Constraints). Andere Forscher beschäftigten sich mit den zeitlichen Verschiebungen der modularen Komponenten im Entwicklungsablauf (Heterochronie), wodurch z. B. Verschiebungen in den Größenverhältnissen zwischen verschiedenen Organen erklärbar wären. Gavin de Beer hatte bereits 1954 die These aufgestellt, dass Änderungen im Timing von Entwicklungsereignissen die Variation von Merkmalen verursachen können, etwa längere oder kürzere Beinen oder die Ausbildung oder Nichtausbildung eines Schwanzes auslösen können.[8]
Master-Kontrollgene und Genregulation
Nach Scott F. Gilbert kann 1977 als Konzeptionsjahr der neuen Forschungsrichtung Evo-Devo angesehen werden, begründet durch das Erscheinen dreier bedeutender Publikationen in diesem Jahr:[9] Stephen J. Goulds Ontogeny and Phylogeny, Francois Jacobs Evolution by Thinkering sowie eine technische Arbeit von A. Maxam und Walter Gilbert zu DNA-Sequenzierung. 1982/83 entdeckte man wichtige Master-Kontrollgene,[10] die an der Regulierung grundlegender Körperbaupläne beteiligt sind, darunter die Hox-Gene, die für die Spezifikation der Körperlängsachse (in entwicklungsbiologischer Terminologie: der Anterior-Posterior-Körperachse) hauptverantwortlich sind, später die Pax-Gene mit grundlegender Bedeutung für die Augenentwicklung sowie die Mkx-Gene, die an der Herz-Formation beteiligt sind. Es stellte sich heraus, dass die Gruppe der Hox-Gene „in abgewandelter Form in bisher allen untersuchten vielzelligen Tieren vorkommt“,[11] sie sind homolog und müssen daher über einen sehr großen Zeitraum in der Evolution konserviert sein; mindestens seit der „kambrischen Explosion“ vor 530 Millionen Jahren, Paul Layer spricht sogar von rund einer Milliarde Jahre.[12] Die Entdeckung der Hox-Gene und ihrer Homologie für die Tierstämme zählt zu den herausragenden Entdeckungen der modernen Biologie der letzten Jahrzehnte. (siehe hierzu auch Homöobox).
In der Folge ermöglichten eine immer einfachere, schnellere und kostengünstigere Sequenzierung von Genomen und die vergleichende Genetik einen verbesserten Einblick in die Genregulationsprozesse während der Entwicklung. Dies hatte zur Folge, dass sich jene Thematik zu einem der stärksten Forschungsfelder von Evo-Devo entwickelte.[13]
Theorie der erleichterten Variation
Die Entschlüsselung der genetischen Basis der Entwicklungsvorgänge beim Wachstum hat gezeigt, dass die wesentlichen Prozesse in Entwicklungs-Modulen organisiert sind. Dutzende bis hunderte genetisch codierte Strukturen und Struktureinheiten werden über gemeinsame regulatorische Einheiten synchron gesteuert. „Master-Kontrollgene“ an Schlüsselstellen können die Entwicklung ganzer Organe induzieren, z. B. kann das pax6-Gen überall die Entwicklung funktionsfähiger Augen induzieren. Die auslösenden regulatorischen Einheiten, meist zelluläre Signalwege und (über Transkriptionsfaktoren gesteuerte) cis-regulatorische Abschnitte im Genom abseits der proteincodierenden Sequenzen, steuern dabei die Entwicklung keinesfalls bis ins Detail, sondern bilden gleichsam Schalter, die in sich koordinierte Entwicklungspfade ein- oder ausschalten können. Die genetische Basis des Steuerungswegs ist also unterschiedlich zu derjenigen der damit gesteuerten Struktur selbst. Dies bedeutet, dass sie unabhängig davon variieren und selektiert werden kann. Sean Carroll prägte dafür das Bild des „genetischen Werkzeugkastens“.
Andere Prozesse werden gar nicht bis ins letzte Detail genetisch festgelegt. Das Entwicklungsprogramm stellt hier lediglich eine noch weitgehend gestaltlose Grundstruktur bereit, die dann erst durch Einflüsse der Umwelt im Detail gestaltet wird: beispielsweise die Reifung des Zentralnervensystems, bei dem von den zahllosen angelegten synaptischen Verbindungen zwischen den Nervenzellen die benötigten verstärkt werden und die nicht genutzten zugrunde gehen. Dadurch braucht die Detailarchitektur nicht genetisch vorgegeben zu werden.
Die Autoren Kirschner und Gerhart fassen die Auswirkungen dieser Erkenntnisse auf die Evolutionstheorie zusammen, sie sprechen von erleichterter Variation (facilitated variation).
Konservierte Kernprozesse
Die grundlegenden Strukturen der Zellorganisation und zahlreiche der dem Körperbauplan und seinen Organen zugrunde liegenden Strukturen werden demnach als konservierte Kernprozesse betrachtet. Sie dienen der Feinsteuerung durch die Entwicklungsmodule danach gleichsam als Rohmaterial. Die Einzelprozesse ändern sich dabei nicht. Zellverhaltensweisen können also evolutionär neu kombiniert werden oder in neuem Ausmaß eingesetzt werden. Wichtige Beispiele für solche konservierten Kernprozesse sind nach Kirschner und Gerhart:
- der einheitliche genetische Code aller Lebewesen
- die selektiv durchlässige Zellmembran zur Kommunikation zwischen Zellen sowie
- die identische Funktion der Hox-Gene.
Die stabilen Kernprozesse erlauben aus Sicht der evolutionären Entwicklungsbiologie Ausprägungsformen oder Eigenschaften, die eine erleichterte phänotypische Variation ermöglichen. Das sind nach Kirschner/Gerhart:
- explorative Prozesse,
- schwache regulatorische Kopplungen und
- Kompartimentbildung beim Embryo.
Exploratives Verhalten
Die differenzierte Ausbildung etwa von Sehnen, Muskeln, Nerven und Blutgefäßen während der Entwicklung wird nicht im Detail vom Genom vorgegeben. Ihre Entstehungsweise kann als explorativ bezeichnet werden. Dabei zeigen Zellen je nach ihrer zellularen Umgebung alternative Reaktionen. So können Zellen „verzweigte Strukturen“[14] schaffen, die den gewünschten Geweberaum optimal ausfüllen[15] (z. B. Blutgefäß, Nervensystem) (Abb. 2). Explorative Strukturen sind im Verlauf der Entwicklung hochgradig anpassungsfähig.
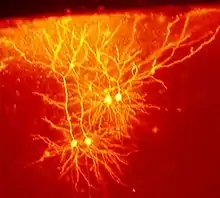
Schwache regulatorische Kopplungen zwischen Zellen
Die für evolutionäre Variation notwendigen Neukombinationen der Kernprozesse sind durch Zellkommunikation vermittelt. Die Autoren sprechen hier von schwachen regulatorischen Kopplungen. „Schwach“, weil das Zellsignal die Entwicklung nicht im Detail steuert, sondern nur einschaltet, also eine nur schwache Beziehung zu den Spezifika des Outputs im Zielbereich hat. In der Regel bestimmt der Signalstoff an der Zieladresse das „An“ oder „Aus“ für die Expression eines dort vorhandenen Gens, dieser kann in derselben Zelle oder in einer anderen Zelle sein. Was dann jedoch genau geschieht, ist durch die eigene Regulation im Zielbereich festgelegt. Der zelluläre Mechanismus im Zielbereich ist schon früher entwickelt worden und braucht für die spezifische Reaktion nur aktiviert werden.[16]
Kompartimentierung
Erst im Verlauf der Entwicklung kommt es zur Ausbildung differenzierter Zellen für spezifische Gewebetypen (Haut, Muskel, Nerven, Organe etc.). Es gibt also Regionen des Embryos, in denen in einer ganz bestimmten Phase der Entwicklung ein oder mehrere ganz bestimmte Gene der Zellen exprimiert (aktiviert) und bestimmte Signalproteine produziert werden.[17] Die Fähigkeit, unterschiedlich konservierte Kernprozesse an unterschiedlichen Orten im Organismus zu aktivieren und diese Reaktionsräume eigentlich erst zu schaffen, nennen sie Kompartimentierung. Ein Insektenembryo bildet in der mittleren Phase der Entwicklung ca. 200 Kompartimente aus. Kompartimentkarten dienen als Gerüst für Anordnung und Bau komplexer anatomischer Strukturen von Lebewesen. Jeder Tierstamm hat seine typische Karte.[17] Die Ausprägung dieser Kompartimente ist die eigentliche Aufgabe der hox-Gene.
Die Organismen, d. h. der Phänotyp, spielen demnach eine Hauptrolle bei der Festlegung von Natur und Maß der Variation.[18] Phänotypische Variation kann nicht beliebig sein. Vielmehr bedingt erleichterte Variation einen beeinflussten „vorsortierten“ Output phänotypischer Variation durch einen Organismus.[19] Variation wird vorwiegend deshalb erleichtert, weil vielfältige Entwicklungsmöglichkeiten in dem verfügbar sind, was Organismen bereits besitzen.[20]
Methoden der empirischen Forschung
Die evolutionäre Entwicklungsbiologie bedient sich in der empirischen Forschung entwicklungsbiologischer und molekularbiologischer Labormethoden, um Faktoren und Steuerungsmechanismen für die Ausbildung und den evolutionären Wandel von Geweben, Organen und morphologischen Strukturen zu erkennen. Das Auftreten solcher Veränderungen wird im Verlauf der Stammesgeschichte der Organismen rekonstruiert.
Zunächst standen experimentelle Transplantationsversuchen an Embryonen im Mittelpunkt. Grafts wurden beispielsweise an den Wirbeltier-Extremitäten entfernt und an anderen Stellen wieder eingepflanzt. In jüngerer Zeit wird molekularbiologisch mit In-situ-Hybridisierung und vor allem mit Gen-Knockout operiert. Durch das Abschalten von Genen kann man auf deren Funktion bei der Entwicklung schließen. Man spricht auch von gain of function bzw. loss of function Experimenten. Einen wichtigen Beitrag für Evo-Devo haben bildgebende microCT-Verfahren (Abb. 7), Computertomografie im Mikro- und Nanometerbereich.[21] Mit Kontrastmitteln lassen sich Genaktivitäten sichtbar machen, so dass der Beitrag eines Gens oder mehrerer beim raum-zeitlichen Entwicklungsvorgang beobachtbar wird.[22] Benötigt wird neben der Genexpressionsebene die „kalibrierte, dreidimensionale Darstellung anatomischer Strukturen in deren natürlichem Aussehen und räumlichen Beziehungen, so nahe am natürlichen Zustand wie für präparierte Specimen nur irgend möglich“ (Metscher).[23]
Einen umfassenden Anspruch zur Kartierung des kompletten Embryonalverlaufs von Drosophila mit bildgebenden Verfahren hat das Projekt BDTNP (Berkeley Drosophila Transcription Network Project). Ziel ist dabei, vollständige Genexpressionsatlanten zu erstellen. Erzeugt wird ein Datensatz von 75.000 Bildern je Embryo mit sichtbar gemachten Aktivitäten von ca. 50 % der Gene. Das „repräsentiert eine solide Beobachtungsgrundlage für die Analyse der Beziehung zwischen Gensequenz, gewebespezifischer Genexpression und Entwicklung in der Tierwelt“ (Tomancak).[22] Der komplette Atlas enthält die Daten aller Transkriptionsprodukte des Drosophila-Genoms in allen Phasen der Entwicklung. Das führt in Zukunft zur „automatisierten Erstellung und Speicherung der Expressionsmuster lebender Arten in vier Dimensionen“.[22] Das Projekt BDTNP zeigt beim heutigen Stand im Internet mit Videostreams die computergestützte statistische Auswertung spezifischer Genexpressionen hunderter von Embryonalvergleichen von Drosophila (virtuelle Embryonen). Der Vergleich der Prozesse dient der Erzeugung statistischer Wahrscheinlichkeiten für das Entstehen phänotypischer Bandbreiten bestimmter Gewebe. Mit Stressoren (Hitze, Kälte, Ernährung etc.) lassen sich zukünftig Expressionsmuster verändern, statistisch auswerten und auf diese Weise mögliche evolutionäre Entwicklungspfade aufspüren.
Ausgewählte empirische Forschungsergebnisse
Belege bei der Taufliege

Erkenntnisse, dass Umweltfaktoren auf Vererbung und Evolution wirken, hatte bereits der Brite Conrad Hal Waddington (1942). Er konnte seine Theorie später (1953) anhand von Veränderungen an den Adern der Fruchtfliege (Drosophila melanogaster) empirisch belegen, indem er die Fliegeneier einige Generationen lang jeweils kurzen Hitzeschocks aussetzte. Dabei blieben nach einigen Generationen die Querverstrebungen an den Flügeln aus. Wurden die Hitzeschocks in Folgegenerationen als externer Stressor aufgehoben, blieben die durch sie induzierten Variationen am Flügeladersystem aber weiterhin erhalten, das heißt, die Querverstrebungen erschienen nicht wieder. Die Hitzeschocks waren ein ausreichend starker Anstoß, dass bisherige Entwicklungspfade verlassen wurden. Bereits angelegte, aber bis dahin nicht genutzte, maskierte Pfade traten durch den äußeren Einfluss zu Tage. Die Entwicklung wurde mit Waddingtons Worten in einem neuen Pfad kanalisiert. Der Umweltfaktor war nur so lange erforderlich, bis der neu kanalisierte Entwicklungsverlauf, wie Waddington es ausdrückte, nachträglich auch genetisch assimiliert ist.[24] Hier bleibt er wieder so lange kanalisiert bzw. stabil, und zwar auch bei neuen auftretenden Mutationen, bis entweder eine Mutation oder aber neue Umwelteinflüsse stark genug sind, dass die Kanalisierung an ihre Grenzen stößt. Gegebenenfalls führt das dann unter Einwirkung von Schwellenwerteffekten, wie oben erläutert, zu einer neuen Variation des Phänotyps.
Was Waddington mit den Laborversuchen nicht zeigen konnte, ist, wie ein adaptiver Weg entsteht, dass auf einen Umweltfaktor (hier: Hitze) geeignet reagiert wird. Die Variation der Flügeladern ist kein adaptives Merkmal auf Hitzeeinwirkung. „Es ist keineswegs sicher, dass er mit nennenswerter Häufigkeit auf irgendeine besondere adaptive Morphologie gestoßen wäre“.[25]
Veränderung der Schnäbel bei Darwin-Finkenarten
Bei Darwin-Finkenarten auf den Galápagos-Inseln gibt es Indizien dafür, dass es in nur wenigen Generationen auf Grund von verändertem Nahrungsangebot (Initiator) zur Umbildung der Schnäbel kommen kann.[26]
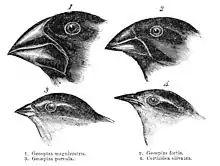
Man konnte einen Transkriptionsfaktor identifizieren, der an der Schnabelbildung im Embryo maßgeblich beteiligt ist. Außerdem wurden etwa 15 Genomabschnitte gefunden, die große Unterschiede zwischen Gruppen von Finken mit stumpfen oder spitzen Schnäbeln aufwiesen.[27]
Das in Neuralleistenzellen produzierte knochenmorphogenetische Protein BMP4, experimentell in die Neuralleiste eines Hühnchens eingepflanzt, führte dort ebenfalls zur Veränderung der Schnabelform. Das Hühnchen entwickelte breitere und größere Schnäbel als normal. Andere Wachstumsfaktoren haben nicht diese Wirkung.[28] Obgleich also der experimentell manipulierte Schnabel seine Größe bzw. Form ändert, wird er dennoch in die Anatomie des Vogelkopfes integriert. „Es kommt nicht zu einer monströsen Fehlentwicklung“ (Kirschner/Gerhart).
Die Schnabelbildung ist ein komplexer Entwicklungsprozess, an dem fünf Nester von Neuralzellen beteiligt sind. Die Nester empfangen Signale von Gesichtszellen an den fünf Orten und reagieren auf sie. Daher beeinflussen Merkmale, die die Neuralleistenzellen beeinflussen, das Schnabelwachstum in koordinierter Weise.[29] Eventuell könntre so besser als nach der klassischen synthetischen Evolutionstheorie erklärt werden, wie in nur wenigen Generationen allein durch das Wechselspiel von zufälliger Mutation und Selektion eine derartig umfangreiche, koordinierte phänotypische Variation entstehen kann, die eines wechselseitigen Zusammenspiels vieler separater Entwicklungsparameter bedarf.
Evo-Devo zeigt an diesem Beispiel die erklärte Wirkungsweise: Kleine Ursache (wenige Veränderungen regulatorischer Proteine) führen zu großer Wirkung (integrierte Veränderung der Schnabelform), gesteuert durch epigenetische Prozesse der Entwicklung, insbesondere durch ein breites adaptives Zellverhalten der Neuralleistenzellen des Schnabels und des Gesichtsumfelds.[30] Aus der gut erforschten Kenntnis der Entwicklung des Schnabels und seiner Modifikationen kann geschlossen werden, dass sich „recht umfangreiche Veränderungen der Schnabelgröße und Schnabelform mit ein paar regulatorischen Mutationen eher erreichen lassen als mit einer Summierung von langen Folgen kleiner Veränderungen“.[30] Nicht final erforscht ist in diesem Beispiel, wodurch die Veränderungen des Bmp4-Spiegels in der Entwicklung ausgelöst werden. Eine Möglichkeit sind genetische Zufallsmutationen, wahrscheinlicher sind Reaktionswege der Entwicklung auf den Stress der Tiere, der durch die anhaltende Veränderung des Nahrungsangebots entsteht. Diese Veränderung wurde ja von den Grants im Zusammenhang mit der Variation der Schnäbel dokumentiert.[31][32]
Evolution des Auges
Durch Analyse spontan entstandener Mutationen der Taufliege Drosophila, denen die Augen fehlen, ist es Genetikern gelungen, ein Schlüsselgen aus der Regulationskaskade der Augenentwicklung zu identifizieren.[33] Dieses Gen erwies sich als ein Transkriptionsfaktor, das heißt, es codiert ein Protein, welches an die DNA bindet und dadurch die Transkription anderer Gene verstärkt bzw. verhindert. Das pax6 genannte Gen gehört zu einer ganzen Familie regulatorischer Gene, die alle Entwicklungsvorgänge steuern. In einem aufsehenerregenden Versuch ist es den Forschern gelungen, durch künstlich induzierte Expression des Gens auch in anderen Körperteilen (funktionsfähige!) Augen zu erzeugen: An den Antennen, an der Flügelbasis, am Thorax usw. Durch heute fast routinemäßige Vergleiche mit dem Genom anderer Organismen erwies sich: Gene ähnlicher Sequenz, die aller Wahrscheinlichkeit nach homolog sind, wurden bei Tierarten aus nahezu allen daraufhin untersuchten Tierstämmen gefunden: z. B. bei Wirbeltieren (Maus, Mensch), Weichtieren (Muscheln, Tintenfische), Fadenwürmern u. v. a., und in allen Fällen war es (neben einigen anderen Aufgaben) an der Entwicklung von Augen beteiligt.[34] Sogar die primitiven Augenflecken des Plattwurms Dugesia und die Linsenaugen am Schirmrand der Würfelqualle Tripedalia cystophora wurden von dem gleichen bzw. einem homologen Gen gesteuert.
Dies war deswegen unerwartet, weil sich diese Tiere in der Evolution mindestens seit dem Kambrium vor über 540 Millionen Jahren auseinanderentwickelt haben. Trotzdem war es möglich, mit dem Gen der Maus bei der Taufliege Augen zu induzieren. Die Augenentwicklung erfordert das fein abgestimmte Zusammenspiel einiger hundert Effektorgene.
Dies lässt sich am ehesten dadurch erklären, dass diese Gene, die im Jargon der Genetiker „stromabwärts“ (engl.: downstream) von pax6 liegen, Bindungsstellen (sog. cis-regulatorischen Elemente) für das Pax6-Protein enthalten. Pax6 ist dabei nur ein einziger Faktor in einem fein austarierten Netzwerk von Signalketten und Steuerungswegen, welches im Detail noch kaum bekannt ist. Für solche in der Evolution über hunderte von Millionen Jahren konservierte Entwicklungswege wurde 1997 das Konzept Deep Homology (tiefe Homologie) eingeführt.[35]
Bei Betrachtung der Augen im Detail zeigt sich allerdings, dass es nicht unbedingt wahrscheinlich ist, dass eine einfache Weiterentwicklung eines einmal entstandenen Auges bereits die ganze Geschichte ausmacht. Zwar haben alle Augen aller Tiere dasselbe lichtempfindliche Molekül, eine Variante des Sehpigments Rhodopsin (welches bereits bei Einzellern und auch bei Prokaryonten vorkommt). Neben dem Sehpigment gehört bereits zu den einfachsten Augen ein lichtabschirmendes Pigment (zum Richtungssehen), außer bei den allereinfachsten Konstruktionen auch ein lichtdurchlässiger „Glaskörper“, der aus einem durchsichtigen Protein („Crystallin“ genannt) besteht. Beim Vergleich verschiedener Augentypen zeigt es sich, dass unterschiedliche Organismen unterschiedliche Pigmente (Melanin, Pterin, Ommochrom) und vor allem völlig unterschiedliche Crystalline verwenden. Beinahe alle Crystalline sind darüber hinaus Enzyme oder Abkömmlinge von solchen, die andernorts im Körper noch völlig andere, essenzielle Aufgaben zu erfüllen haben.[36] Außerdem gibt es die Rezeptorzellen in zwei Ausfertigungen, als „rhabdomerische“ und „ciliare“ Rezeptoren mit völlig unterschiedlichem Zellbauplan. Die rhabdomerische Ausfertigung findet sich bei den Arthropoden, die ciliare bei den Wirbeltieren, aber auch bei den Würfelquallen. Die wesentlichen Bestandteile des Auges sind also zwar unter in der Evolution hochgradig konservierter Entwicklungskontrolle, darunter aber beinahe zufällig „durcheinandergewürfelt“. Dies erscheint am ehesten dadurch erklärbar, dass beim Aufbau des immer komplexer werdenden Auges immer mehr ursprünglich unabhängig und für einen anderen Zweck entstandene Strukturen, aber auch Entwicklungspfade und Signalwege neben ihrer ursprünglichen Funktion auch bei der Augenentwicklung verwendet wurden. Dies ist am besten dadurch erklärbar, dass sie cis-regulatorische Sequenzen evolutiv neu erworben haben, die durch Kontrollgene der Augenentwicklung wie z. B. pax6 steuerbar sind[37] (die Gene sind ja in allen Körperzellen identisch vorhanden!). Der Entwicklungsweg insgesamt ist also homolog, die weiteren Strukturen wie Pigmente, Glaskörper, Linse etc. sind aber vermutlich konvergente Bildungen. Für ihre Entstehung sind aber weder neue Proteinfamilien oder auch nur neue Gene erfunden worden, sondern bereits bestehende wurden umfunktioniert („rekrutiert“ oder auch „ko-optiert“).[38]
Veränderung des Skeletts bei der Entstehung des Schildkrötenpanzers

Der Panzer der Schildkröten stellt eine Besonderheit unter den Wirbeltieren dar, deren evolutionäre Entwicklung erst durch genetische Methoden weitgehend aufgeklärt werden konnte. Hierbei entstand seit Ende der 1990er Jahre die vorherrsche Auffassung, dass Schildkröten entgegen den bis dahin herrschenden Annahmen eine Schwestergruppe zu den Krokodilen und Vögeln darstellen, also mit diesen zusammen eine Gruppe (Klade) bilden. Die Abspaltung der neuen Gruppe der Schildkröten erfolgte etwa vor 260 Millionen Jahren, und es wird vermutet, dass die Entwicklung ihrer Besonderheiten - wie die des Panzers - schrittweise in den nachfolgen 100 Millionen Jahren erfolgte.[39][40]
In umfangreichen Genomanalysen verschiedener embryonaler Entwicklungsstadien einer Schildkrötenart konnten inzwischen mehrere Steuergene und Signalketten identifiziert werden, die an der Sonderentwicklung des Panzers beteiligt sind. Dazu gehören u. a. das Gen für Wnt5a aus der Familie der Wnt-Signalwege.[41]
Flösselhechte: Experiment Landgang

In einem achtmonatigen Versuch mit juvenilen Flösselhechten der Gattung Polypterus aus dem tropischen Afrika (Polypterus senegalus) wurde 2014 erstmals eruiert, wie gut sich Flösselhechte an die Bedingungen an Land anpassen, wenn man ihnen die aquatische Lebensweise vollständig entzieht. Dabei zeigte sich, dass sich die Tiere überraschend schnell an die neuen Bedingungen anpassen konnten. Die Versuchstiere überlebten nicht nur, sondern blühten in der neuen Umgebung sogar auf. Ihre Anpassungen umfassten sowohl Änderungen der Muskulatur also auch der Knochenstruktur. Die Versuchsindividuen konnten signifikant besser auf dem Trockenen laufen als die aquatischen Kontrolltiere. Für Evolutionsbiologen der evolutionären Entwicklungsbiologie erlaubt diese unerwartet hohe Entwicklungsplastizität, Rückschlüsse darauf zu ziehen, wie die ersten Meeresbewohner, etwa der Tiktaalik, vor 400 Millionen Jahren an Land gingen und mit dem Übergang von Flossen zu Extremitäten allmählich Tetrapoden entstehen konnten. Dieser Versuch mit Flösselhechten bestätigte für einen evolutionär äußerst wichtigen Systemübergang des Landgangs, aus dem schließlich alle Landwirbeltiere hervorgingen, die Hypothese, dass Tiere in evolutionär kurzer Zeit sowohl ihre Anatomie als auch ihr Verhalten als Reaktion auf Umweltänderungen plastisch anpassen können.[42][43] Genetische Mutationen könnten langfristig die durch die neue Umweltsituation geschaffenen Bedingungen unterstützen und für geeignete Vererbung sorgen. Die evolutionäre Abfolge wäre demnach nicht genetische Mutation, natürliche Selektion, Adaptation in der Population, sondern umgekehrt: Veränderung der Umweltbedingungen, dauerhafte, noch nicht genetisch vererbbare phänotypische Adaptation, unterstützende genetische Mutationen.
Literatur
Konzeptionelle Grundlagen
- Ron Amundson: The Changing Role of the Embryo in Evolutionary Thought. 2005, ISBN 0-521-80699-2.
- Wallace Arthur: Understanding Evo-Devo Cambridge University Press, 2021, ISBN 9781108836937.
- Ingo Brigandt: Jenseits des Neodarwinismus? Neuere Entwicklungen in der Evolutionsbiologie. In: Philipp Sarasin, Marianne Sommer: Evolution – Ein interdisziplinäres Handbuch. J. B. Metzler, 2010, S. 115–126.
- Sean B. Carroll: EvoDevo – Das neue Bild der Evolution. Berlin 2008, ISBN 978-3-940432-15-5. (Orig.: Endless Forms Most Beautiful, USA 2006)
- Scott F. Gilbert: The morphogenesis of evolutionary development biology. 2003.
- Brian Keith Hall, Wendy M. Olson (Hrsg.): Keywords and Concepts in Evolutionary Developmental Biology, Harvard University Press, 2003, ISBN 9780674022409.
- Mark C. Kirschner, John C. Gerhart: Die Lösung von Darwins Dilemma – Wie Evolution komplexes Leben schafft. Rowohlt, 2007, ISBN 978-3-499-62237-3. (Orig.: The Plausibility of Life (2005))
- Manfred D Laubichler: Evolutionäre Entwicklungsbiologie, Frankfurt am Main, Fischer, S, 2007, ISBN 9783596155705.
- Alessandro Minelli, Giuseppe Fusco (Hrsg.): Evolving pathways - key themes in evolutionary developmental biology. Cambridge University Press, Cambridge/ New York 2008.
- Alessandro Minelli: Forms of Becoming - The Evolutionary Biology of Development. Princeton University Press, 2009, ISBN 978-0-691-13568-7.
- Gerd B. Müller, Stuart A. Newman: Origination of Organismal Form – Beyond the Gene in Development and Evolutionary Biology. MIT-Press, 2003, ISBN 0-262-13419-5.
- Christiane Nüsslein-Volhard: Das Werden des Lebens – Wie Gene die Entwicklung steuern. dtv, 2006, ISBN 3-423-34320-6.
- Mary Jane West-Eberhard: Development Plasticity and Evolution. Oxford University Press, 2003.
Weiterführende Literatur und Internetartikel
- Scott F. Gilbert, David Epel: Ecological Development Biology. Integrating Epigenetics, Medicine and Evolution. Sinauer Ass. USA, 2009.
- Eva Jablonka, Marion J. Lamb: Evolution in four Dimensions. Genetic, Epigenetic, Behavioral and Symbolic Variation in the History of Lfe. MIT Press, 2005. (PDF; 6,2 MB)
- M. Neukamm: Evolutionäre Entwicklungsbiologie: Neues Paradigma. In: Laborjournal. 15(11), 2009, S. 24–27. (pdf)
- Massimo Pigliucci: What, if anything, Is an Evolutionary Novelty? In: Philosophy of Science. 75, 12/2008, S. 887–898.
Einzelnachweise
- Brian K. Hall: Balfour, Garstang and de Beer: The First Century of Evolutionary Embryology. In: American Zoologist. 40(5), 2000, S. 718–728.
- Alan C. Love, Rudolf A. Raff: Knowing your ancestors: themes in the history of evo-devo. In: Evolution and Development. 5(4), 2003, S. 327–330.
- Scott F. Gilbert: The morphogenesis of evolutionary development biology. 2003, S. 471.
- Eva Jablonka, Marion J. Lamb: Evolution in four Dimensions. Genetic, Epigenetic, Behavioral and Symbolic Variation in the History of Life. MIT Press, 2005, S. 261–266.
- Scott F. Gilbert: The morphogenesis of evolutionary development biology. 2003, S. 474.
- M. Pigliucci, G. Müller: Evolution. The Extended Synthesis. 2010, Kap. 1: Elements of an Extended Evolutionary Synthesis.
- M. Pigliucci, G. Müller: Evolution. The Extended Synthesis. 2010, Kap. 1: Elements of an Extended Evolutionary Synthesis.
- Scott F. Gilbert: The morphogenesis of evolutionary development biology. 2003, S. 470.
- Scott F. Gilbert: The morphogenesis of evolutionary development biology. 2003, S. 473.
- Master-Kontrollgene oder Masterregulatorgene sind Gene, „die fortlaufend einen oder mehrere Transkriptionsfaktoren in gewissen sich differenzierenden Zellen exprimieren“ (Kirschner und Gerhard 2005, S. 384)
- Christiane Nüsslein-Volhard: Das Werden des Lebens – Wie Gene die Entwicklung steuern. 2006, S. 93.
- Paul G Layer: Evo-Devo: Die molekulare Entwicklungsbiologie als Schlüssel zum Verständnis der Evolutionstheorie. In: Zeitschrift Für Pädagogik Und Theologie. 61 (4), 2009, S. 328.
- Gerd B. Müller, Stuart A. Newman: The Innovation Triad. An Evo-Devo Agenda. In: Journal of Experimental Zoology. Band 304B, 2005, S. 387–503.
- Hans Meinhardt: Die Simulation der Embryonalentwicklung. In: Spektrum der Wissenschaft. 03/2010.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 264.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 302.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 382.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 328.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 333.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 369.
- Anm.: Die besten Geräte haben nach Metscher (Metscher 2009) heute eine Auflösung von 60 Nanometer. 1 nm = 1 Milliardstel m bzw. 1 Millionstel mm (z.Vgl.: Eine menschliche Zelle hat im Durchschnitt eine Größe von 10 bis 20 Mikrometer, das ist um die Größenordnung 1000 größer.)
- Pavel Tomancak u. a.: Patterns of gene expression in animal development. (Kurzfassung des Artikels: Global analysis of patterns of gene expression during Drosophila embryogenesis. In: Genome Biol. 8, 2007, no 145.1–145.34.
- Brian Metscher: MicroCT for comparative morphology: simple staining methods allow high contrast 3D imaging of diverse non-mineralized animal tissues. 2009. (online)
- Conrad Hal Waddington: Genetic Assimilation of an Acquired Charakter. In: Evolution. Vol. 7, No. 2, 1953, S. 118–126.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 316.
- Mark C. Kirschner, John C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 321f.
- S. Lamichhaney, J. Berglund u. a.: Evolution of Darwin's finches and their beaks revealed by genome sequencing. In: Nature. Band 518, Nummer 7539, Februar 2015, S. 371–375, doi:10.1038/nature14181, PMID 25686609, PDF.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 3121 mit Bezug auf Studien von Clifford Tabin an Darwinfinken
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 318.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 319.
- M. C. Kirschner, J. C. Gerhart: Die Lösung von Darwins Dilemma. 2007, S. 320ff.
- Die Darwinfinken – Evolution im Zeitraffer. In: Neue Zürcher Zeitung. 12. Juli 2006; P. u. R. Grant sehen Rückkreuzungen (Introgression) als eine adaptive Ursache für die Variation der Schnäbel S. 3.
- Georg Halder, Pstrick Callaerts, Walter J. Gehring: Induction of ectopic eyes by targetes expression of the eyless gene in Drosophila. In: Science. 267, 1995, S. 1788–1792.
- Walter J. Gehring, Kazuho Ikeo: Pax6: mastering eye morphogenesis and eye evolution. In: Trends in Genetics. 15(9), 1999, S. 371–377.
- N. Shubin, C. Tabin, S. Carroll: Fossils, genes and the evolution of animal limbs. In: Nature. Band 388, Nummer 6643, August 1997, S. 639–648, doi:10.1038/41710, PMID 9262397 (Review) (freier Volltext).
- Joram Piatigorsky, G.Wistow: The recruitment of crystallins: new functions precede gene duplication. In: Science. 252, 1991, S. 1078–1079.
- Pavel Vopalensky, Zbynek Kozmik: Eye evolution: common use and independent recruitment of genetic components. In: Philosophical Transactions of the Royal Society. B 364, 2009, S. 2819–2832.
- Joram Piatigorsky: A Genetic Perspective on Eye Evolution: Gene Sharing, Convergence and Parallelism. In: Evolution: Education and Outreach. 1(4), 2008, S. 403–414.
- Z. Wang, J. Pascual-Anaya u. a.: The draft genomes of soft-shell turtle and green sea turtle yield insights into the development and evolution of the turtle-specific body plan. In: Nature genetics. Band 45, Nummer 6, Juni 2013, S. 701–706, doi:10.1038/ng.2615, PMID 23624526, PMC 4000948 (freier Volltext).
- Tyler R. Lyson, Gabriel S. Bever: Origin and Evolution of the Turtle Body Plan. In: Annual Review of Ecology, Evolution, and Systematics. 51, 2020, S. 143, doi:10.1146/annurev-ecolsys-110218-024746, PDF.
- J. Zhang, P. Yu u. a.: Global Analysis of Transcriptome and Translatome Revealed That Coordinated WNT and FGF Regulate the Carapacial Ridge Development of Chinese Soft-Shell Turtle. In: International Journal of Molecular Sciences. Band 22, Nummer 22, November 2021, S. , doi:10.3390/ijms222212441, PMID 34830331, PMC 8621500 (freier Volltext).
- Emily M. Standen, Trina Y. Du, Hans C. E. Larsson: Developmental plasticity and the origin of tetrapods. In: Nature. 513, 4. September 2014, S. 54–58.
- How fish can learn to walk. (Video)