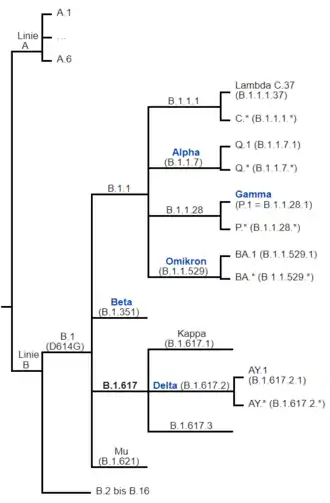
Bezeichnung der SARS-CoV-2-Varianten
Für die Bezeichnung der SARS-CoV-2-Varianten wurde von der Weltgesundheitsorganisation (WHO) am 31. Mai 2021 ein eigenes Bezeichnungssystem unter Verwendung ausgeschriebener Namen von Buchstaben des griechischen Alphabets eingeführt, für alle Varianten, die als besorgniserregend (englisch Variant of Concern, VOC) oder als von näherem Interesse klassifiziert sind (englisch Variant of Interest, VOI). Weiterhin gibt es parallel dazu die wissenschaftlichen Bezeichnungen der SARS-CoV-2-Varianten[2] nach Pango, GISAID und Nextstrain.
_(cropped).jpg.webp)
Das Bezeichnungssystem der WHO soll in der COVID-19-Pandemie eine weltweit einheitliche und allgemeinverständliche Kommunikation über Virusvarianten von praktischer epidemiologischer Bedeutung in Politik, Gesellschaft und öffentlicher Gesundheitsvorsorge gewährleisten. Ein weiteres Ziel stellt die Abkehr von den an Ländernamen orientierten Ad-hoc-Bezeichnungen dar, die zuvor in Politik und Medien verwendet worden waren. Dies soll eine Stigmatisierung oder Diskriminierung der betreffenden Regionen und der dort lebenden oder von dort stammenden Menschen vermeiden.[2] Die Richtlinien der WHO sehen seit 2015 vor, dass sich Bezeichnungen für neue Erreger oder Krankheiten nicht auf geografische Regionen, Tiere oder bestimmte Menschengruppen beziehen sollen.[3]
Liste der Varianten mit WHO-Bezeichnungen
Ende 2020 ordnete die WHO nach dem Auftreten erster Varianten von SARS-CoV-2 diejenigen, die ein (möglicherweise) erhöhtes Risiko für die globale öffentliche Gesundheit darstellten, in folgende Kategorien ein, um Prioritäten für die globale Überwachung und Forschung setzen:[4]
- Variant of Concern (VOC – ‚Besorgniserregende Variante‘) und
- Variant of Interest (VOI – ‚Variante von Interesse‘)
- Variant Under Monitoring (VUM – ‚Variante unter Überwachung‘)[4]
Seit 31. Mai 2021 werden die Variants of Interest und Variants of Concern von der WHO mit Buchstaben des griechischen Alphabets bezeichnet. Die anderen wissenschaftlichen Bezeichnungen der Virusvarianten bleiben parallel bestehen.[2]
| SARS-CoV-2-Varianten gemäß WHO[4] | |||||||
|---|---|---|---|---|---|---|---|
| WHO-Bezeichnung(a) | PANGO-Linie[5] | Erstnachweis | WHO-Klassifizierung und Datum | ||||
| Region | Datum | VOI | VUM | VOC | Deklassifizierung | ||
| Alpha | B.1.1.7 | September 2020 | … | … | 18.12.2020 | … | |
| Beta | B.1.351 B.1.351.* | Mai 2020 | … | … | 18.12.2020 | … | |
| Gamma | P.1 (B.1.1.28.1) P.1.* | November 2020 | … | … | 11.01.2021 | … | |
| Delta | B.1.617.2 AY.* (B.1.617.2.*) | Oktober 2020 | 04.04.2021 | … | 11.05.2021 | … | |
| Epsilon | B.1.427 B.1.429 | März 2020 | 05.03.2021 | 06.07.2021 | … | 09.11.2021 | |
| Zeta | P.2 (B.1.1.28.2) | April 2020 | 17.03.2021 | 06.07.2021 | … | 17.08.2021 | |
| Eta | B.1.525 | mehrere Länder | Dezember 2020 | 17.03.2021 | 20.09.2021[6] | … | 22.12.2021 |
| Theta | P.3 (B.1.1.28.3) | Januar 2021 | 24.03.2021 | 06.07.2021 | … | 17.08.2021 | |
| Iota | B.1.526 | November 2020 | 24.03.2021 | 20.09.2021[6] | … | 22.12.2021 | |
| Kappa | B.1.617.1 | Oktober 2020 | 04.04.2021 | 20.09.2021[6] | … | 29.12.2021 | |
| Lambda | C.37 (B.1.1.1.37) | Dezember 2020 | 14.06.2021 | … | … | … | |
| My (Mu)(b) | B.1.621 | Januar 2021 | 30.08.2021 | … | … | … | |
| übersprungen(c)(d) | |||||||
| übersprungen(c) | |||||||
| Omikron (Omicron)(b) | B.1.1.529 | November 2021 | … | 24.11.2021[7] | 26.11.2021 | … | |
Wissenschaftliche Bezeichnungen
Die Zuordnung zu bekannten oder neuen Viruslinien kann zweifelsfrei nur über eine Sequenzierung des vollständigen Virusgenoms erfolgen. Von den drei parallel verwendeten Nomenklatursystemen hat jedes bestimmte Stärken.
Pango-Nomenklatur (z. B. B.1.1.529)
Die Pango-Nomenklatur wurde 2020 zur detaillierten Nachverfolgung der SARS-CoV-2-Varianten aufgebaut. Ausgangsbezeichnung sind die beiden ersten Linien der Varianten A (z. B. Wuhan/WH04/2020) und B (z. B. Wuhan-Hu-1). Die davon abstammenden Varianten werden als phylogenetischer Baum aufgebaut, indem die direkten Untervarianten jeweils durch Anhängen einer Zahl bezeichnet werden, wie B.2 für die zweite Untervariante der Variante B. Auch von dieser Untervariante können weitere Untervarianten abstammen, wie B.2.4 als vierte Untervariante der vorherigen Variante. Dies kann beliebig weiter fortgeführt werden. Damit die Bezeichnungen nicht zu lang und unübersichtlich werden, werden die Zahlenangaben auf maximal drei begrenzt und dieser Bezeichnung bei der nächsten Untervariante der nächste Buchstabe (bzw. die nächste Buchstabenkombination) zugewiesen. So wird die erste Untervariante von B.1.1.529 nicht mehr mit B.1.1.529.1, sondern kurz mit BA.1, deren erste Untervariante mit BA.1.1 bezeichnet. Auch dieses Verfahren wird beliebig weiter fortgeführt.[12]
Die Pango-Nomenklatur stellt die Beziehungen der Varianten in einem feineren Maßstab dar als GISAID und Nextstrain. Sie hat das Potenzial, frühzeitig vor sich verbreitenden Varianten zu warnen, zum Beispiel vor regionalen Ausbrüchen oder möglicher Dominanz durch selektive Vorteile wie Immunevasion oder erhöhte Übertragbarkeit.[13]
GISAID-Nomenklatur (z. B. GRA)
GISAID (Global Initiative on Sharing All Influenza Data) ist weltweite Wissenschaftsinitiative, die freien Zugang zu Genomdaten von Influenza- und SARS-CoV-2-Viren fördert, sie ging 2008 in Betrieb.[14]
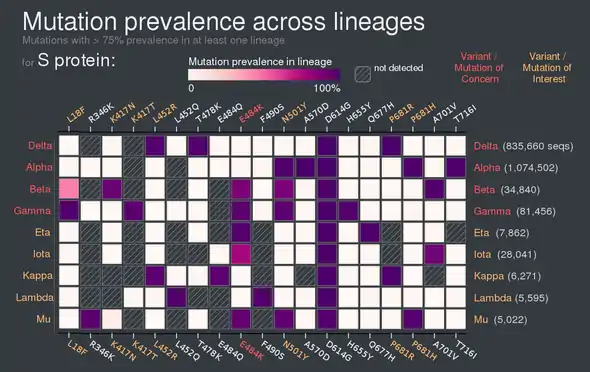
GISAID fasst SARS-CoV-2-Untervarianten in Kladen zusammen, recht ähnlich wie bei Nextstrain.[13] Diese Kladen werden nach dem Einbuchstabencode der relevantesten Aminosäure-Veränderung bezeichnet. Wird eine neue Klade mit zusätzlicher relevanter Aminosäure-Veränderung gebildet, wird diese jeweils hinten angehängt. So wird die SARS-CoV-2-Variante Alpha zusammen mit ihren Untervarianten in GISAID als „GRY“ bezeichnet, die drei Einbuchstabencodes G, R und Y stehen hier der Reihe nach für die Aminosäure-Veränderungen D614G, G204R und N501Y des Spike-Proteins.[15]
Nextstrain-Nomenklatur (z. B. 21K)
Nextstrain ist eine Zusammenarbeit zwischen Forschern in Seattle (USA) und Basel (Schweiz), die eine Sammlung von Open-Source-Tools zur Visualisierung der Genetik hinter der Ausbreitung von Virusausbrüchen bereitstellt. Mit Nextstrain lässt sich seit 2015 in Echtzeit verfolgen, wie sich ein Virus verändert, das heißt, welche Mutationen es während der Ausbreitung ansammelt.[16]
Nextstrain fasst SARS-CoV-2-Untervarianten in Kladen zusammen, recht ähnlich wie bei GISAID.[13] Eine neue Klade wird mit den beiden Endziffern der Jahreszahl sowie Ergänzung durch jährlich fortlaufenden Buchstaben bezeichnet, zum Beispiel „20I“ für die 2020 entdeckte SARS-CoV-2-Variante Alpha zusammen mit ihren Untervarianten.
Bezeichnung der Mutationen (z. B. D614G)
Eine Aminosäure-Veränderung im Protein, die durch nicht-synonyme Mutationen im Gen verursacht wird, wird mit
- dem Einbuchstabencode der ursprünglichen Aminosäure,
- deren Position in der Aminosäuresequenz als Zahl sowie
- dem Einbuchstabencode der neuen, veränderten Aminosäure bezeichnet.
Eine der ersten durchsetzungsfähigen Mutationen im Gen des Virus, die zur Veränderung D614G im Spike-Protein führte, bedeutet somit den Austausch von D (Asparaginsäure) durch G (Glycin) an der Position 614 der Aminosäuresequenz des Spike-Proteins.[17]
Weitere Bezeichnungen (z. B. VUI 202012/01)
Die SARS-CoV-2-Variante Alpha, in Pango mit B.1.1.7 bezeichnet, wurde in Großbritannien Ende 2020 noch mit „VUI 202012/01“ (englisch Variant Under Investigation ‚Variante unter Beobachtung‘) bezeichnet. Viele Medien sprachen bis zur Einführung der WHO-Bezeichnungen im Mai 2021 oftmals von der „britischen Variante“ oder „Kent-Variante“[18] – entgegen der Empfehlung der WHO, regionale Namen für Krankheitserreger zu vermeiden.[3]
WHO-Bezeichnungen
Die Bezeichnung der als besorgniserregend oder beobachtungsbedürftig eingestuften SARS-CoV-2-Varianten erfolgt zum Zeitpunkt ihrer Erstklassifizierung durch die WHO. Dabei werden die Bezeichnungen in Reihenfolge der Buchstaben des griechischen Alphabets vergeben. Nur bei Einführung des Bezeichnungssystems im Mai 2021 wurde abweichend verfahren: Die zehn zuvor von der WHO klassifizierten Virusvarianten wurden vorrangig nach ihrer damaligen Klassifizierung (Alpha bis Delta: VOC, Epsilon bis Kappa: VOI) und nachrangig in der Reihenfolge ihrer Erstklassifizierung bezeichnet.
Hintergrund der Einführung
Die wissenschaftlichen Bezeichnungen hätten ihre Vorteile, doch seien sie durch ihre Zahlenlastigkeit schwer auszusprechen sowie zu behalten und daher anfällig für eine falsche Wiedergabe, erklärte die WHO in Genf.[2] Mit der Namensänderung will die Weltgesundheitsorganisation vermeiden, dass Länder oder Regionen mit dort erstentdeckten Virusvarianten verknüpft werden und Menschen, die dort leben oder von dort kommen, diskriminiert werden.[2] „Kein Land soll für die Entdeckung und die Meldung von Varianten stigmatisiert werden“, erklärte die WHO-Epidemiologin für Infektionskrankheiten und technische Leiterin des COVID-19-Bereichs, Maria Van Kerkhove.[19] Mit der neuen Nomenklatur verbinde sich auch die Hoffnung, dass Staaten bereitwilliger über die Entdeckung neuer Varianten berichten könnten, wenn sie wüssten, dass die Bezeichnung der neuen Linie dann Rho oder Sigma laute und nicht mit ihrem Namen in Verbindung gebracht würde.[20]
Nach monatelangen Überlegungen unter Beteiligung des Bakteriologen Mark Pallen, Experte für mikrobielle Genomik am Quadram Institute und der University of East Anglia, hat man sich für das griechische Alphabet entschieden, wie der daran Beteiligte berichtete. Dabei hätten die Experten zunächst auch griechische Götter oder pseudo-klassische Wortschöpfungen in Betracht gezogen. Doch diese Ideen seien verworfen worden, weil viele der Namen bereits für Marken, Unternehmen oder Ortsnamen genutzt würden. Auch die Namensgebung von Fantasie- oder Familiennamen sei verworfen worden.[21] Die WHO-Namensbezeichnung wurde in Treffen von mehr als 50 Wissenschaftlern, Angehörigen der Gesundheitsberufe und Experten für Nomenklatur und Taxonomie entwickelt.[22] Virologe Frank Konings begleitete die Treffen; er ist Leiter der Virus Evolution Working Group der WHO.[23] Es sei überraschend schwierig gewesen, ein akzeptables System für die Neubenennung zu finden, so Maria van Kerkhove.[24] Pallen selbst habe den Vorschlag gestoppt, die Varianten als VOC1, VOC2 und so weiter zu bezeichnen. Ausgesprochen ähnelten sie doch zu sehr einem englischen Schimpfwort.[25] Florian Krammer, Virologe der Icahn School of Medicine at Mount Sinai in New York, kritisierte die leichte Verwechselbarkeit von Zeta, Eta und Theta.[26]
Nilu Ahmed, Psychologin an der Universität Bristol, erklärte, die Benennung von Viren nach Orten diene der Distanzierung der Menschen davon: „Das überträgt Schuld auf die anderen und erlaubt denen, die das Narrativ nutzen, sich als Opfer zu inszenieren.“ So sei die „Spanische“ Grippe dort nur zuerst öffentlich gemacht worden, doch nicht dort entsprungen.[27]
Vermeidung von Stigmatisierungen
Sowohl in den Medien als auch im Alltag verwendeten Menschen bis ins Frühjahr 2021 statt der wissenschaftlichen Bezeichnung für SARS-CoV-2-Varianten, wie etwa B.1.617.2 für die erstmals in Indien aufgetretene Variante, oft den Ort ihrer Entdeckung. So sprachen sie zum Beispiel von der „indischen Variante“, die nun neutral als Variante „Delta“ bezeichnet wird.[28] In einer offiziellen Erklärung forderte die indische Regierung insbesondere Social-Media-Plattformen auf, sämtliche Inhalte mit der Bezeichnung „indische Variante“ zu löschen.[28]
US-Präsident Donald Trump benutzte die Bezeichnung „China-Virus“ für das ursprüngliche im chinesischen Wuhan entdeckte Coronavirus. Was seine Worte tatsächlich befeuerten, waren Diskussionen über die Verantwortung und womöglich eine Schuld Chinas. In der Folge stiegen Angriffe auf asiatischstämmige US-Amerikaner an, weshalb sein Nachfolger Joe Biden ein Gesetz gegen Hassverbrechen an Bürgern asiatischer Herkunft erließ.[29]
Bundeskanzlerin Angela Merkel sagte am 12. Januar 2021: „Wir müssen das britische Virus in den Griff bekommen.“ Falls das nicht gelinge, seien weitere harte Maßnahmen unvermeidbar, womöglich auch ein Lockdown bis Ostern. Die Redaktion der britischen Boulevardzeitung Daily Mail zog bereits am selben Tag einen direkten Vergleich zwischen Merkels Wortwahl „britisches Virus“ und den Aussagen von Donald Trump.[30]
Siehe auch
Weblinks
- Tracking SARS-CoV-2 variants. Weltgesundheitsorganisation (WHO) (englisch).
- SARS-CoV-2 (hCoV-19) Mutation Reports – Lineage – Mutation Tracker. outbreak.info (englisch, Gesamt-Überblick aller relevanten Varianten mit Bewertung durch CDC, ECDC, PHE, WHO, mit Mutationen).
Pango-Nomenklatur
- Rules for the Designation and Naming of Pango Lineages. In: pango.network. (englisch, Details der Pango-Nomenklatur).
GISAID-Nomenklatur
- Clade and lineage nomenclature. GISAID, 21. März 2021 (englisch, Details der GISAID-Nomenklatur).
Nextstrain-Nomenklatur
- Nextstrain SARS-CoV-2 resources. In: nextstrain.org. (englisch, „Latest global SARS-CoV-2 analysis“ führt direkt zur Analyse).
- Emma Hodcroft: CoVariants. In: covariants.org. (englisch, Überblick Nextstrain-Kladen).
Einzelnachweise
- Aktualisierte Fassung: SARS-CoV-2 (hCoV-19) Mutation Reports – Lineage Comparison. In: outbreak.info. Abgerufen am 16. Januar 2022.
- WHO announces simple, easy-to-say labels for SARS-CoV-2 Variants of Interest and Concern. Weltgesundheitsorganisation (WHO), 31. Mai 2021, abgerufen am 1. Juni 2021 (englisch).
- WHO issues best practices for naming new human infectious diseases. WHO, 8. Mai 2015, abgerufen am 16. Januar 2022 (englisch): „The use of names such as ‘swine flu’ and ‘Middle East Respiratory Syndrome’ has had unintended negative impacts by stigmatizing certain communities or economic sectors […] We’ve seen certain disease names provoke a backlash against members of particular religious or ethnic communities, create unjustified barriers to travel, commerce and trade, and trigger needless slaughtering of food animals. […] Once disease names are established in common usage through the Internet and social media, they are difficult to change, even if an inappropriate name is being used. Therefore, it is important that whoever first reports on a newly identified human disease uses an appropriate name that is scientifically sound and socially acceptable.“
- WHO: Tracking SARS-CoV-2 variants; hier: Variants Under Monitoring (VUM). (Nicht mehr online verfügbar.) In: Activities. who.int, 26. November 2021, archiviert vom Original am 26. November 2021; abgerufen am 26. November 2021 (englisch).
- Pango-Network
- WHO: Weekly epidemiological update on COVID-19 – 21. September 2021 – Edition 58. (PDF; 2 MB) In: Publications / Overview / Emergency Situational Updates. who.int, 21. September 2021, S. 5–7, abgerufen am 23. September 2021 (englisch).
- WHO: Tracking SARS-CoV-2 variants; hier: Variants Under Monitoring (VUM). In: Activities. who.int, 24. November 2021, archiviert vom Original am 26. November 2021; abgerufen am 25. November 2021 (englisch).
- Warum die neue Corona-Variante nicht Xi heißt. In: spiegel.de. 27. November 2021, abgerufen am 27. November 2021.
- Marco Latzer, Johannes Hillig: Grosse Besorgnis wegen neuer Corona-Mutation: Was wir über Ny bisher wissen und was nicht. In: msn.ch. Blick, 26. November 2021, abgerufen am 27. November 2021.
- Johannes Hillig, Sven Ziegler, Sarah Frattaroli: Erste Länder stoppen Flüge – Swiss wartet ab: Super-Variante Ny breitet sich aus. In: msn.ch. Blick, 26. November 2021, abgerufen am 30. November 2021.
- Lineage List. cov-lineages.org, abgerufen am 14. Dezember 2021 (Relevanteste Varianten WHO und Deutschland, Stand Dezember 2021).
- Andrew Rambaut, Edward C. Holmes, Áine O’Toole et al.: A dynamic nomenclature proposal for SARS-CoV-2 lineages to assist genomic epidemiology. In: Nature Microbiology. Band 5. Springer Nature, 15. Juli 2020, S. 1403–1407; hier: S. 1404, doi:10.1038/s41564-020-0770-5, PMID 32669681, PMC 7610519 (freier Volltext) – (englisch, nature.com [PDF; 1,7 MB; abgerufen am 16. Januar 2022] Pango Nomenklatur): “We propose that major lineage labels begin with a letter. At the root of the phylogeny of SARS-CoV-2 are two lineages that we simply denote as lineages A and B. The earliest lineage A viruses, such as Wuhan/WH04/2020 (EPI_ISL_406801) […] Different nucleotides are present at those sites in viruses assigned to lineage B, of which Wuhan-Hu-1 (GenBank accession no. MN908947) sampled on 26 December 2019 is an early representative. […] To add further lineage designations, we […] estimated a maximum likelihood tree for these data […]. We defined further SARS-CoV-2 lineages, each descending from either lineage A or B, and assigned a numerical value (for example, lineage A.1 or lineage B.2). Lineage designations were made using the following set of conditions: (1) each descendant lineage should show phylogenetic evidence of emergence from an ancestral lineage into another geographically distinct population, implying substantial onward transmission in that population. […] (2) the lineages identified in step 1 can themselves act as ancestors for virus lineages that then emerge in other geographical areas or at later times, provided they satisfy criteria a–d. This results in a new lineage designation (for example, A.1.1); (3) the iterative procedure in step 2 can proceed for a maximum of three sublevels (for example, A.1.1.1) after which new descendant lineages are given a letter (in English alphabetical sequence from C) so A.1.1.1.1 would become C.1 and A.1.1.1.2 would become C.2.”, s. a. The Pango Nomenclature System – Other Documentation. (PDF) In: pango.network. Archiviert vom Original am 7. Januar 2022; abgerufen am 7. Januar 2022 (englisch, Querverweis der WHO von who.int/en/activities/tracking-SARS-CoV-2-variants auf pango.network).
- Erik Alm, Eeva K. Broberg, Thomas Connor et al.: Geographical and temporal distribution of SARS-CoV-2 clades in the WHO European Region, January to June 2020. In: Eurosurveillance. Band 25, Nr. 32. ECDC, 13. August 2020, ISSN 1560-7917, 2001410, doi:10.2807/1560-7917.ES.2020.25.32.2001410, PMID 32794443, PMC 7427299 (freier Volltext) – (englisch, eurosurveillance.org [PDF; 968 kB; abgerufen am 16. Januar 2022] cov-lineages.org stellt die Pango-Varianten dar): “Conclusions – Overall, the GISAID and Nextstrain nomenclatures provide similar pictures of the situation and may provide useful systems for genomic situation reporting globally. The cov-lineages.org nomenclature provides information at a finer scale and has the potential to provide early warning of expanding lineages that may represent regional outbreaks or later become dominant because of some selective advantage such as vaccine escape or increased transmissibility.”
- Questionnaire for databases – GISAID EpiFlu Database. (PDF; 370 KB) WHO, August 2018, S. 1, abgerufen am 16. Januar 2022 (englisch): „The launch of GISAID’s EpiFlu database on the occasion of the 61 st World Health Assembly (2008) marked the culmination of the establishment of the Global Initiative on Sharing All Influenza Data, a collaboration involving representatives of Member States, scientists of the Global Influenza Surveillance and Response System (GISRS) and Global Influenza Programme (GIP) of the WHO, and experts in licensing intellectual property.“
- Clade and lineage nomenclature. GISAID, 21. März 2021, abgerufen am 16. Januar 2022 (englisch).
- Corona-Pandemie: Mit Nextstrain dem Virus auf der Spur. In: unibas.ch. Universität Basel, 20. März 2021, abgerufen am 4. September 2021: „Dass Richard Neher heute so gefragt ist, liegt auch daran, dass er und sein Kollege Trevor Bedford vom Fred-Hutchinson-Krebsforschungszentrum in Seattle schon vor Jahren eine innovative Idee hatten. Sie wollten live mitverfolgen können, wie sich Krankheitserreger ausbreiten und haben eigens dafür die Webapplikation Nextstrain entwickelt. Seit 2015 ist die Webseite online.“
- Mutation D614G Coronavirus-Variante ist leichter übertragbar. In: pharmazeutische-zeitung.de. 13. November 2020, abgerufen am 6. Juli 2021.
- Ewen Callaway: Neue Namen für das Virus. Spektrum, abgerufen am 3. Juni 2021.
- Alpha, Beta, Gamma – WHO benennt Coronavirus-Varianten um. In: dw.com. Abgerufen am 3. Juni 2021.
- Alpha, Beta, Gamma: Neue Namen für Coronavirus-Varianten. In: aponet.de. 1. Juni 2021, abgerufen am 5. Juni 2021.
- Nach rassistischen Übergriffen: WHO stoppt Benennung von Covid-Mutationen nach Ländern. Focus Online, 1. Juni 2021, abgerufen am 3. Juni 2021.
- Frank Konings, Mark D. Perkins, Jens H. Kuhn et al.: SARS-CoV-2 Variants of Interest and Concern naming scheme conducive for global discourse. In: Nature microbiology. Band 6. Springer Nature, 9. Juni 2021, S. 821–823, doi:10.1038/s41564-021-00932-w, PMID 34108654 (englisch, nature.com [PDF; 785 kB; abgerufen am 20. Januar 2022] 47 Autoren): “A group convened and led by the Virus Evolution Working Group of the World Health Organization reports on its deliberations and announces a naming scheme that will enable clear communication about SARS-CoV-2 variants of interest and concern.”
- JV Chamary: WHO Has Renamed Coronavirus Variants Using Greek Letters. Forbes.com, 31. Mai 2021, abgerufen am 20. Januar 2022 (englisch): „Pallen was part of WHO’s discussions to find better names, which was coordinated by virologist Frank Konings, who leads WHO’s Virus Evolution Working Group.“
- Helen Branswell: The name game for coronavirus variants just got a little easier. In: statnews.com. The Boston Globe, 31. Mai 2021, abgerufen am 20. Januar 2022 (englisch): „But it was surprisingly tricky to come up with an acceptable system, Van Kerkhove said.“
- Alpha, Beta, Gamma – Virusvarianten werden umbenannt. Der Spiegel, 1. Juni 2021, abgerufen am 3. Juni 2021.
- Berit Uhlmann: B.1.1.7 soll nun Alpha heißen. Süddeutsche Zeitung, 1. Juni 2021, abgerufen am 3. Juni 2021.
- Christiane Oelrich: Corona-Varianten bekommen neutrale Namen. In: aerztezeitung.de. 1. Juni 2021, abgerufen am 20. Januar 2022: „Indem Phänomene wie Viren nach Orten benannt werden, distanzierten sich Menschen davon, sagte Dr. Nilu Ahmed, Psychologin an der Universität Bristol. „Das überträgt Schuld auf die anderen und erlaubt denen, die das Narrativ nutzen, sich als Opfer zu inszenieren.“ So etwas könne auch falsche Annahmen fördern: So sei die Spanische Grippe […] erst in Spanien ausführlich dokumentiert worden.“
- APA/AFP: Indische Regierung verbietet Begriff „indische Variante“. In: DiePresse. 22. Mai 2021, abgerufen am 23. Mai 2021.
- B.1.1.7 ist jetzt „Alpha“ WHO benennt Virusvarianten um. 1. Juni 2021, abgerufen am 5. Juni 2021.
- Peter Littger: Sprachlich unkluge Bezeichnung „Das britische Virus“ existiert nicht. 13. Februar 2021, abgerufen am 5. Juni 2021.