Porenbildendes Toxin
Ein porenbildendes Toxin (englisch pore-forming toxin, PFT) ist ein Protein, das eine Pore in bestimmten Biomembranen bildet und als Toxin wirkt.
Eigenschaften
Porenbildende Toxine sind Exotoxine, die meistens von pathogenen Bakterien hergestellt werden, z. B. Clostridium septicum und Staphylococcus aureus. Die toxische Wirkung wird durch die zytotoxische Wirkung auf Säugerzellen vermittelt, da die Zellmembranen von Zellen durch die Poren perforiert werden, woraufhin das Zytosol ausläuft, die Homöostase der Zelle nicht mehr aufrechterhalten werden kann und die Nekrose und Apoptose erfolgt. Durch den entstandenen Schaden für den Wirt werden die meisten porenbildenden Toxine zu den Virulenzfaktoren gezählt. Porenbildende Toxine dienen Bakterien zur Erhöhung ihres Nahrungsangebots, zur Veränderung der Umgebungsbedingungen und teilweise zur Vermeidung einer Immunreaktion durch Zerstörung von Immunzellen. Teilweise werden porenbildende Toxine von Bakterien zur Bekämpfung anderer Bakterienarten hergestellt, z. B. die kanalbildenden Subtypen der Colicine und möglicherweise auch Nisin.[1] Im Gegensatz zu porenbildenden Toxinen sind Bakteriocine oder die nichtribosomalen Peptide wie z. B. Polymyxine oder Iturine zwar destabilisierend für Biomembranen, aber meistens zu klein zur Ausbildung eines Ionenkanals.[2]
Neben den bakteriellen porenbildenden Toxinen werden porenbildende Proteine auch von Eukaryoten zur Abwehr bakterieller Infektionen gebildet, z. B. Defensin und Sarcotoxin. Andere dienen in giftigen Eukaryoten als Toxin, z. B. Melittin im Bienengift. Eukaryotische MACPF-ähnliche Proteine kommen beim Perforin und dem C9 des Komplementsystems als Teil der Immunabwehr vor.[3] Die porenbildenden Toxine mit antimikrobieller Wirkung gehören zu den antimikrobiellen Peptiden.
Typen
Bakterielle porenbildende Toxine werden in verschiedene Gruppen eingeteilt, die sich hinsichtlich des Aufbaus und des Wirkmechanismus unterscheiden:[4]
- α-porenbildende Toxine, z. B. Cytolysin A
- β-porenbildende Toxine, z. B. α-Hämolysin, PV-Leukozidin.
- Cholesterolabhängige Zytolysine, z. B. Pneumolysin
- Kleine porenbildende Toxine, z. B. Gramicidin A.
Weitere membranschädigende Toxine ohne Porenbildung sind zugleich AB-Toxine (z. B. Anthraxtoxin).
α-porenbildende Toxine
α-PFT wie das Cytolysin A (synonym Hämolysin E, silent hemolysin locus A) kommen in manchen Stämmen von Escherichia coli (v. a. uropathogene Stämme),[5] Salmonella enterica und Shigella flexneri vor.[6]
β-porenbildende Toxine
_and_soluble-form_PVL_(pale_green-green)_toxins_-_PDB_7AHL_and_1T5R.png.webp)
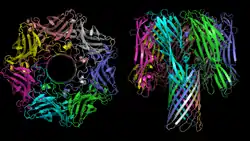
β-PFT sind in ihrer Proteinstruktur aus β-Faltblättern aufgebaut. α-Hämolysin[7] und Leukozidin S[8] sind strukturell verwandt, ebenso Aerolysin[9] und Clostridium ε-Toxin.[10]
β-PFT sind Proteine, die als lösliche Monomere oder porenbildende Proteinkomplexe vorliegen. Der Kopf des heptameren α-Hämolysins ragt aus der Membran heraus, während der Stamm in der Lipiddoppelschicht der Membran liegt. Der Stamm besteht aus einem vierzehnsträngigen β-barrel, mit zwei Strängen aus jedem Monomer. Das Vibrio cholerae Cytolysin ist ebenfalls heptamer.[11] Das Staphylococcus aureus γ-Hämolysin bildet eine oktamere Pore aus sechzehn Strängen.[12] Das Panton-Valentine-Leukozidin S besitzt als Monomer eine ähnliche Form.[13]
Die Bindung der Monomere aneinander und die Einfügung in die Membran erfolgt ähnlich wie bei den Zytolysinen durch Zusammenlagerung peripher an der Membran, gefolgt von einer Änderung der Proteinfaltung und der Einfügung des Stamms in die Membran anhand hydrophober Aminosäuren am Ende des Stammes.[4]
Manche β-PFT wie das Clostridium ε-Toxin und das Clostridium perfringens Enterotoxin (CPE) binden an Rezeptoren, vermutlich Claudin bei CPE,[14] sowie vermutlich GPI-Anker oder andere Glykosylierungen beim ε-Toxin. Durch die rezeptorvermittelte Akkumulation der Monomere wird die Zusammenlagerung zur multimeren Pore begünstigt. CPE dienen den Bakterien zur Abwehr von Makrophagen,[15] zur Veränderung der Umgebungsbedingungen[15] und zur Verbesserung des Nahrungsangebots.
Cholesterol-abhängige Zytolysine
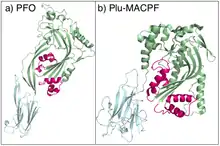
Cholesterol-abhängige Zytolysine (auch Thiol-aktivierte Zytolysine, englisch cholesterol-dependent cytolysin, CDC) sind z. B. Pneumolysin aus Streptococcus pneumoniae, Perfringolysin O aus C. perfringens, Listeriolysin O aus Listeria monocytogenes, Ivanolysin aus Listeria ivanovii, Seeligeriolysin aus Listeria seeligeri, Hämolysin aus Bacillus cereus,[16] Alveolysin aus Bacillus alvei, Streptolysin O aus Streptococcus pyogenes oder Tetanolysin aus Clostridium tetani. Sie bilden Poren von bis zu 260 Å (26 nm) Durchmesser, bestehend aus zwischen 30 und 44 Monomeren.[17] Die Multimerisierung erfolgt peripher an der Zellmembran, woraufhin sich eine α-Helix in eine amphipathische β-Schleife umlagert. CDC sind verwandt mit der MACPF-Familie porenbildender Proteine in Eukaryoten, vermutlich verwenden beide Familien den gleichen Mechanismus.[18]
Kleine porenbildende Toxine
Gramicidine sind nicht-ribosomal erzeugte kurze Proteine von etwa zehn bis fünfzehn Aminosäuren (D- und L-Aminosäuren), die ebenfalls Poren in Biomembranen bilden können.
Literatur
- F. Gisou van der Goot, Pore-forming toxins, Springer, 2001, ISBN 3-540-41386-3.
- A deadly toxin with a romantic name: Panton-Valentine Leukocidin complex. PDBe Quips
Weblinks
Einzelnachweise
- E. Breukink, B. de Kruijff: The lantibiotic nisin, a special case or not? In: Biochimica et Biophysica Acta. Band 1462, Nummer 1–2, Dezember 1999, ISSN 0006-3002, S. 223–234, PMID 10590310.
- H. Lee, H. Y. Kim: Lantibiotics, class I bacteriocins from the genus Bacillus. In: Journal of microbiology and biotechnology. Band 21, Nummer 3, März 2011, ISSN 1738-8872, S. 229–235, PMID 21464591.
- Tschopp J, Masson D, Stanley KK: Structural/functional similarity between proteins involved in complement- and cytotoxic T-lymphocyte-mediated cytolysis. In: Nature. 322, Nr. 6082, 1986, S. 831–4. doi:10.1038/322831a0. PMID 2427956.
- Marcus Mueller, Ulla Grauschopf, Timm Maier, Rudi Glockshuber, Nenad Ban: The structure of a cytolytic alpha-helical toxin pore reveals its assembly mechanism. In: Nature. 459, Nr. 7247, 4. Juni 2009, S. 726–730. doi:10.1038/nature08026. PMID 19421192.
- T. J. Wiles, M. A. Mulvey: The RTX pore-forming toxin α-hemolysin of uropathogenic Escherichia coli: progress and perspectives. In: Future microbiology. Band 8, Nummer 1, Januar 2013, S. 73–84, ISSN 1746-0921. doi:10.2217/fmb.12.131. PMID 23252494. PMC 3570152 (freier Volltext).
- S. Hunt, J. Green, P. J. Artymiuk: Hemolysin E (HlyE, ClyA, SheA) and related toxins. In: Advances in Experimental Medicine and Biology. Band 677, 2010, S. 116–126, ISSN 0065-2598. PMID 20687485.
- Song L, Hobaugh MR, Shustak C, Cheley S, Bayley H, Gouaux JE: Structure of staphylococcal α-hemolysin, a heptameric transmembrane pore. In: Science. 274, Nr. 5294, Dezember 1996, S. 1859–66. doi:10.1126/science.274.5294.1859. PMID 8943190.
- Guillet V, Roblin P, Werner S, et al.: Crystal structure of leucotoxin S component: new insight into the Staphylococcal β-barrel pore-forming toxins. In: J. Biol. Chem.. 279, Nr. 39, September 2004, S. 41028–37. doi:10.1074/jbc.M406904200. PMID 15262988.
- Parker MW, Buckley JT, Postma JP, et al.: Structure of the Aeromonas toxin proaerolysin in its water-soluble and membrane-channel states. In: Nature. 367, Nr. 6460, Januar 1994, S. 292–5. doi:10.1038/367292a0. PMID 7510043.
- Cole AR, Gibert M, Popoff M, Moss DS, Titball RW, Basak AK: Clostridium perfringens ε-toxin shows structural similarity to the pore-forming toxin aerolysin. In: Nat. Struct. Mol. Biol.. 11, Nr. 8, August 2004, S. 797–8. doi:10.1038/nsmb804. PMID 15258571.
- S. De, R. Olson: Crystal structure of the Vibrio cholerae cytolysin heptamer reveals common features among disparate pore-forming toxins. In: Proceedings of the National Academy of Sciences. Band 108, Nummer 18, Mai 2011, S. 7385–7390, ISSN 1091-6490. doi:10.1073/pnas.1017442108. PMID 21502531. PMC 3088620 (freier Volltext).
- K. Yamashita, Y. Kawai, Y. Tanaka, N. Hirano, J. Kaneko, N. Tomita, M. Ohta, Y. Kamio, M. Yao, I. Tanaka: Crystal structure of the octameric pore of staphylococcal γ-hemolysin reveals the β-barrel pore formation mechanism by two components. In: Proceedings of the National Academy of Sciences. Band 108, Nummer 42, Oktober 2011, S. 17314–17319, ISSN 1091-6490. doi:10.1073/pnas.1110402108. PMID 21969538. PMC 3198349 (freier Volltext).
- V. Guillet, P. Roblin, S. Werner, M. Coraiola, G. Menestrina, H. Monteil, G. Prévost, L. Mourey: Crystal structure of leucotoxin S component: new insight into the Staphylococcal beta-barrel pore-forming toxins. In: The Journal of biological chemistry. Band 279, Nummer 39, September 2004, S. 41028–41037, ISSN 0021-9258. doi:10.1074/jbc.M406904200. PMID 15262988.
- Fujita K, Katahira J, Horiguchi Y, Sonoda N, Furuse M, Tsukita S: Clostridium perfringens enterotoxin binds to the second extracellular loop of claudin-3, a tight junction integral membrane protein. In: FEBS Lett.. 476, Nr. 3, Juli 2000, S. 258–61. doi:10.1016/S0014-5793(00)01744-0. PMID 10913624.
- Bruce Alberts, Alexander Johnson, Peter Walter, Julian Lewis, Martin Raff, Keith Roberts: Molecular Biology of the Cell, 4. Auflage, Taylor & Francis 2002, ISBN 978-0-8153-3218-3.
- N. Ramarao, V. Sanchis: The pore-forming haemolysins of bacillus cereus: a review. In: Toxins. Band 5, Nummer 6, Juni 2013, S. 1119–1139, ISSN 2072-6651. doi:10.3390/toxins5061119. PMID 23748204. PMC 3717773 (freier Volltext).
- Tilley SJ, Orlova EV, Gilbert RJ, Andrew PW, Saibil HR: Structural basis of pore formation by the bacterial toxin pneumolysin. In: Cell. 121, Nr. 2, April 2005, S. 247–56. doi:10.1016/j.cell.2005.02.033. PMID 15851031.
- Carlos J. Rosado, Ashley M. Buckle, Ruby H. P. Law, Rebecca E. Butcher, Wan-Ting Kan, Catherina H. Bird, Kheng Ung, Kylie A. Browne, Katherine Baran, Tanya A. Bashtannyk-Puhalovich, Noel G. Faux, Wilson Wong, Corrine J. Porter, Robert N. Pike, Andrew M. Ellisdon, Mary C. Pearce, Stephen P. Bottomley, Jonas Emsley, A. Ian Smith, Jamie Rossjohn, Elizabeth L. Hartland, Ilia Voskoboinik, Joseph A. Trapani, Phillip I. Bird, Michelle A. Dunstone, and James C. Whisstock: A Common Fold Mediates Vertebrate Defense and Bacterial Attack. In: Science. 317, Nr. 5844, 2007, S. 1548–51. doi:10.1126/science.1144706. PMID 17717151.
- Rossjohn J, Feil SC, McKinstry WJ, Tweten RK, Parker MW: Structure of a cholesterol-binding, thiol-activated cytolysin and a model of its membrane form. In: Cell. 89, Nr. 5, 1997, S. 685–92. doi:10.1016/S0092-8674(00)80251-2. PMID 9182756.