Adenylylcyclasen
Adenylylcyclasen (AC), früher Adenylatcyclasen, zählen zur Klasse der Lyasen, d. h. molekülspaltender Enzyme. Bei höheren Organismen sind sie wichtige Vermittler zwischen Hormonen oder anderen Botenstoffen, die an der Außenseite der Zellmembran binden, und zellinternen Botenstoffen (Second Messenger, „sekundärer Botenstoff“), die innerhalb der Zelle die Wirkung dieser Hormone weiterleiten.[1]
| Adenylylcyclasen | ||
|---|---|---|
| ||
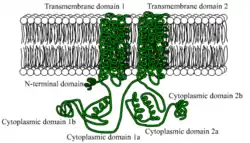
| Schematische Darstellung der Adenylylcyclase, eingebettet in die Zellmembran | ||
| Bezeichner | ||
| Gen-Name(n) | ADCY1, ADCY2, ADCY3, ADCY4, ADCY5, ADCY6, ADCY7, ADCY8, ADCY9, ADCY10 | |
| Enzymklassifikation | ||
| EC, Kategorie | 4.6.1.1, Lyase | |
| Substrat | ATP | |
| Produkte | cAMP + Diphosphat | |
| Vorkommen | ||
| Übergeordnetes Taxon | Lebewesen | |
Beim Vorgang der Signaltransduktion bindet zunächst das Hormon an einen passenden G-Protein-gekoppelten Rezeptor. Dieser setzt das entsprechende G-Protein frei, welches seinerseits die Adenylylcyclase aktiviert. Diese bildet nun im Innern der Zelle den sekundären Botenstoff cyclisches Adenosinmonophosphat (cAMP) aus Adenosintriphosphat (ATP).
Adenylylcyclasen kommen in nahezu allen Lebewesen vor. Beim Menschen sind von ihr derzeit zehn, überwiegend an die Zellmembran gebundene Isoenzyme bekannt.
Vorkommen
Adenylylcyclasen sind in der belebten Natur weit verbreitet. Bereits einfache einzellige eukaryotische Lebewesen wie Paramecium, Euglena und Dictyostelium sowie einige Prokaryoten nutzen Adenylylcyclasen zur Umwandlung von ATP in cAMP. Darüber hinaus nutzen einige prokaryotische Krankheitserreger die Funktion der Adenylylcyclasen oder des cAMPs höherer Lebewesen, indem sie selbst Adenylylcyclasen oder Aktivatoren von Adenylylcyclasen bilden und freisetzen.[2] So wird beispielsweise die Invasivität des Cholera-Erregers Vibrio cholerae auf das indirekt Adenylylcyclasen aktivierende Choleratoxin zurückgeführt. Die freigesetzten Adenylylcyclasen von Bordetella pertussis und Bacillus anthracis, welche erst im Körper des Wirtsorganismus aktiviert werden, sind Bestandteil von deren toxischem Prinzip.
Einteilung
Die Adenylylcyclasen gehören zur Gruppe der Lyasen. Klassisch werden die Adenylylcyclasen in drei Hauptklassen unterteilt. Die Klasse I umfasst die Adenylylcyclasen gramnegativer Bakterien. Adenylylcyclasen der Klasse II spielen als toxischen Proteine prokaryotischer Krankheitserreger eine Rolle sind vom Protein Calmodulin des Wirtsorganismus abhängig. Zahlenmäßig am größten sind die Adenylylcyclasen der Klasse III. Sie umfasst alle eukaryotischen Adenylylcyclasen sowie zahlreiche prokaryotische Enzyme. Seltener werden weitere drei Klassen (IV–VI) von Adenylylcyclasen erwähnt.[3]
Die Adenylylcyclasen des Menschen umfassen 10 verschiedene Isoenzyme und werden als AC1 bis AC10 (oder AC I bis AC X) bezeichnet. Die Isoenzyme AC1 bis AC9 sind membranständige Proteine. Das auch als lösliche Adenylylcyclase (sAC, von soluble adenylyl cyclase) bezeichnete Isoenzym AC10 ist ein cytosolisches Protein, das überwiegend in speziellen Zellkompartimenten lokalisiert vorkommt.[4] Darüber hinaus unterscheiden sich die Isoenzyme der Adenylylcyclasen in ihren Regulationsmechanismen.
| AC1 | AC2 | AC3 | AC4 | AC5 | AC6 | AC7 | AC8 | AC9 | AC10 | |
|---|---|---|---|---|---|---|---|---|---|---|
| Synonym | sAC | |||||||||
| Vorkommen | Gehirn, Nebennieren | Gehirn, Lunge, Skelettmuskulatur | Riechepithel, Gehirn, Nebennieren, Fettgewebe, Pankreas | ubiquitär | Herz, Gehirn | Herz, Nieren, Gehirn, Leber | Gehirn, Blutplättchen, Herz, Milz, Lunge | Gehirn, Lunge | Skelettmuskulatur, Herz, Gehirn, Pankreas | Keimzellen |
| Genetik | ||||||||||
| Gen-Name | ADCY1 | ADCY2 | ADCY3 | ADCY4 | ADCY5 | ADCY6 | ADCY7 | ADCY8 | ADCY9 | ADCY10 |
| Genlocus | 7p13-p12 | 5p15.3 | 2p24-p22 | 14q11.2 | 3q13.2-q21 | 12q12-q13 | 16q12.1 | 8q24 | 16p13.3 | 1q24 |
| Protein | ||||||||||
| UniProt-Bez. | Q08828 | Q08462 | O60266 | Q8NFM4 | O95622 | O43306 | P51828 | P40145 | O60503 | Q96PN6 |
| Länge (Aminosäuren) | 1119 | 1091 | 1144 | 1077 | 1261 | 1168 | 1080 | 1251 | 1353 | 1610 |
| Regulationa | ||||||||||
| Gαs | + | + | + | + | + | + | + | + | + | 0 |
| Gαi/o | − | − | − | − | − | 0 | ||||
| Gβγ | − | + | 0 | + | + | − | 0 | |||
| Ca2+/(Calmodulin)b | + | 0 | + | 0 | − | − | 0 | + | + | |
| Adenosin | − | − | − | − | − | − | − | − | − | − |
| Forskolin | + | + | + | + | + | + | + | + | 0 | 0 |
Struktur
.png.webp)
.png.webp)
Die in der belebten Natur vorkommenden Adenylylcyclasen folgen zum Teil höchst unterschiedlichen Bauplänen. Allen ist gemein, dass sie mindestens eine für die Funktion essenzielle Cyclase-Homologie-Domäne tragen. Die membranständigen Adenylylcyclasen mehrzelliger Tiere (Metazoa) bestehen aus je zwei Transmembrandomänen mit je sechs α-Helices (M1 und M2) und zwei cytosolischen Cyclase-Homologie-Domänen (C1 und C2). Die beiden etwa 40 kDa großen Cyclase-Homologie-Domänen lassen sich weiter in katalytische (C1a und C2a) und regulatorische Subdomänen unterteilen (C1b und C2b). Die katalytischen Subdomänen weisen ein hohes Maß an Homologie auf.[6]
Die dreidimensionale Struktur der katalytischen Subdomänen verschiedener tierischer Adenylylcyclasen konnte mit Hilfe der Röntgenstrukturanalyse aufgeklärt werden. Mit Hilfe dieser Technik konnte für die katalytischen Subdomänen eine aus einem βαββαβ-Motiv bestehende Struktur, welche beispielsweise auch bei einigen DNA-Polymerasen vorkommt, bestätigt werden. Beide katalytische Domänen interagieren miteinander unter Bildung einer funktionellen Einheit. An der Kontaktseite beider Untereinheiten besitzt die gebildete C1a-C2a-Einheit eine Furche mit einer Bindungsstelle für das Substrat ATP. Ebenfalls in dieser Furche ist eine weitere Bindungsstelle für den Aktivator Forskolin, einem Diterpen aus dem Harfenstrauch Plectranthus barbatus, enthalten. Die Bindungsstelle für die stimulierenden G-Proteine Gs und Golf umfasst einen kleinen Anteil des N-Terminus der C1a-Subdomäne und einen größeren Anteil der C2a-Subdomäne. Darüber hinaus wird eine weitere Bindungsstelle an der regulatorischen C1b-Subdomäne vermutet. Die postulierte Bindungsstelle für inhibitorische G-Proteine (Gi/o-Familie) an der C1a-Subdomäne ist von der Bindungsstelle stimulierender G-Proteine verschieden.[5]
Funktion
Katalyse der cAMP-Produktion
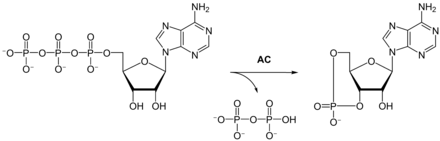
Die Adenylylcyclase katalysiert die Bildung von cAMP aus ATP unter Abspaltung von Pyrophosphat. Je nach Isoenzym werden von einem Molekül Adenylylcyclase zwischen einem und 100 Substratmolekülen pro Sekunde umgesetzt. Die jeweiligen Michaeliskonstanten Km, welche der Substratkonzentration bei halbmaximaler Umsatzgeschwindigkeit entsprechen, betragen zwischen 30 und 400 µM.[5]
Bei Vertebraten und Invertebraten (z. B. Aplysia) hat cAMP die Funktion eines Second Messengers.[7] Das Enzym ist also ein Bestandteil der Signalkaskade zur Weiterleitung von Signalen innerhalb von Zellen. Es ist auch oft verbunden mit einer Stimulierung der Transmitterfreisetzung und der Signalweiterleitung, mit einer Steigerung der Herzfrequenz und einer Relaxation glatter Muskulatur. Die Adenylylcyclase ist somit für die Weiterleitung von Reizen, sowie für die Wirkung zahlreicher Hormone (beispielsweise Glucagon und Adrenalin) und Neurotransmitter (beispielsweise Serotonin) verantwortlich.
Regulation
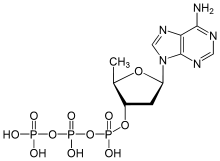
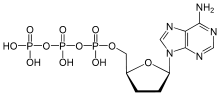
Im menschlichen Organismus wird die Aktivität der Adenylylcyclasen physiologisch insbesondere von den bei der Signaltransduktion beteiligten G-Proteinen reguliert. Stimulierende G-Proteine der Familie Gs/olf begünstigen eine geschlossene Konformation der dimeren katalytischen Einheit der Adenylylcyclase und aktivieren somit deren Enzymfunktion. Inhibitorische Gi/o-Proteine führen über eine Stabilisierung der offenen Konformation zu einer Hemmung der Adenylylcyclasefunktion. Einige Isoenzyme der Adenylylcyclase (AC1, AC3 und AC8) werden zusätzlich durch Calcium/Calmodulin reguliert.
Darüber hinaus können die Adenylylcyclasen pharmakologisch reguliert werden. Das Diterpen Forskolin ist ein nichtselektiver Aktivator der Adenylylcyclasen, der alle menschlichen Adenylylcyclasen mit Ausnahme der AC9 aktiviert. Die Entdeckung, dass Adenosin nichtselektiv alle Adenylylcyclasen hemmt, führte zu Entwicklung zahlreicher Nukleosid- und Nukleotid-Analoga. Sie führen zumeist als sogenannte P-Site-Inhibitoren der Adenylylcyclasen zu einer nicht- oder unkompetitiven Hemmung, die auf verschiedenen Mechanismen beruht. Für ihre inhibitorische Wirkung benötigen die Nukleosid-Analoga, wie beispielsweise 2′-Desoxyadenosin, zelleigenes Pyrophosphat als Co-Inhibitor. Ihre inhibitorische Potenz wird von den Pyrophosphat-unabhängigen Nukleotid-Analoga, wie beispielsweise 2′,5′-Didesoxyadenosin-3′-tetraphosphat, 2′,5′-Didesoxyadenosin-3′-triphosphat, übertroffen.[8] Darüber hinaus können die Adenylylcyclasen kompetitiv von nukleotidischen Substratanaloga, wie beispielsweise β-L-2′,3′-Didesoxy-5′-Adenosintriphosphat, durch Verdrängung des Substrats ATP gehemmt werden.
Einzelnachweise
- Eintrag zu Adenylat-Cyclase. In: Römpp Online. Georg Thieme Verlag, abgerufen am 13. Juni 2011.
- D. M. Cooper: Regulation and organization of adenylyl cyclases and cAMP. In: Biochem. J. Band 375, Pt 3, November 2003, S. 517–529, doi:10.1042/BJ20031061, PMID 12940771, PMC 1223734 (freier Volltext).
- Roger A. Johnson: Encyclopedia of molecular pharmacology. Hrsg.: Walter Rosenthal, Stefan Offermanns. Springer, Berlin 2008, ISBN 3-540-38916-4, Adenylate cyclases, S. 28–37.
- J. H. Zippin, Y. Chen, P. Nahirney u. a.: Compartmentalization of bicarbonate-sensitive adenylyl cyclase in distinct signaling microdomains. In: FASEB J. Band 17, Nr. 1, Januar 2003, S. 82–84, doi:10.1096/fj.02-0598fje, PMID 12475901.
- W. J. Tang, J. H. Hurley: Catalytic mechanism and regulation of mammalian adenylyl cyclases. In: Mol. Pharmacol. Band 54, Nr. 2, August 1998, S. 231–240, PMID 9687563.
- J. H. Hurley: Structure, mechanism, and regulation of mammalian adenylyl cyclase. In: J. Biol. Chem. Band 274, Nr. 12, März 1999, S. 7599–7602, PMID 10075642.
- Eric Kandel: Principles of Neural Science, Fourth Edition. McGraw-Hill Companies,Incorporated, 2000, ISBN 0-8385-7701-6 (eingeschränkte Vorschau in der Google-Buchsuche).
- C. W. Dessauer, J. J. Tesmer, S. R. Sprang, A. G. Gilman: The interactions of adenylate cyclases with P-site inhibitors. In: Trends Pharmacol. Sci. Band 20, Nr. 5, Mai 1999, S. 205–210, PMID 10354616.