Zytogenetik
Die Zytogenetik (auch Cytogenetik, Zellgenetik) ist das Teilgebiet der Genetik, das die Chromosomen vorwiegend mit dem Lichtmikroskop analysiert. Untersucht werden Anzahl, Gestalt, Struktur und Funktion der Chromosomen, denn die DNA eines Chromosomensatzes im Zellkern enthält den größten Teil der Erbinformation (des Genoms) eines Lebewesens. Anomalien der Chromosomen versucht man mit deren phänotypischer Auswirkung zu verbinden.
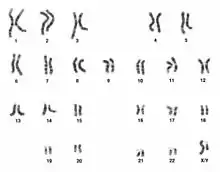
Chromosomen erfassen
Sowohl während der Meiose in Prophase 1 (Zygotän, Pachytän, Diplotän, Diakinese), Metaphase 1 und Metaphase 2, als auch während der mitotischen (Pro-)Metaphase lassen sich Chromosomen individuell erkennen und der Karyotyp erstellen. Nach Endoreplikation sind vergrößerte Chromosomen in Endometaphasen und während des ganzen Zellzyklus als Polytänchromosomen sichtbar. Der DNA-Gehalt eines Chromosomensatzes erschließt die Genomgröße eines Organismus, zeigt das Ausmaß von eventueller Endoreplikation und lässt auch selektive Endoreplikation ermessen.
Die Länge eines stabil vererbbaren Chromosomen-Armes ist durch die Ausdehnung der Spindelachse in der Anaphase begrenzt.[1] Deswegen sind die Elemente eines Karyotyps als Kompartimente eines großen Genoms zu verstehen. Frühe Versuche, ihre Anzahl beim Menschen zu bestimmen, ergaben 46 bis 48 Chromosomen; lediglich über das XX/XY-System der Geschlechtsbestimmung war man sich einig.[2][3] Reproduzierbare Analysen in den 1950er-Jahren verschafften der normalen menschlichen Karyotyp-Formel 2n = 46 allgemeine Anerkennung.[4][5]
Forschungsgeschichte
Theodor Boveri und Walter Sutton entwickelten unabhängig voneinander die Chromosomentheorie der Vererbung. Sie besagt kurzgefasst: Individuelle Chromosomen enthalten unterschiedliche Erbfaktoren (Gene).[6] Die polare Auftrennung der homologen Chromosomen eines Bivalentes erfolgt in der Meiose zufällig und unabhängig von der anderer Bivalente. Entsprechend zufällig werden die Gene der Bivalente verteilt. Dies entspricht den Regeln von Gregor Mendel. Innerhalb der einzelnen Chromosomen sind die Gene jedoch „gekoppelt“.[7]
Methoden
Der Erfolg der Zytogenetik hing von der Einsicht ab, dass man Gewebe nicht mit dem Mikrotom zerschneiden durfte, sondern deren Zellen in hypotonischer Lösung mit feinen Nadeln möglichst voneinander trennen sollte. Sanftes Quetschen drückt die Chromosomen in eine Ebene. Farbstoffe wie Karmin, Orcein oder das Giemsa-Gemisch machen Chromosomen zu Farbkörpern, um sie vom Zytoplasma abzuheben. Besonders deutlich gelingt dies mit der Feulgenreaktion (Feulgen-Prozedur), weil sie spezifisch die chromosomale DNA (Kern-DNA) anfärbt.
In der pathologischen Routine werden Schnittpräparate menschlicher Gewebe begutachtet. Um für zytogenetische Probleme eine Fraktion unverletzter mitotischer Zellkerne zu erhalten, ist das Mikrotom auf 15 μm Schnittdicke einzustellen.[8]
Induzierte Bänderung
Die klassischen Methoden erlaubten jedoch nicht, die menschlichen (Metaphase-)Chromosomen zweifelsfrei zu unterscheiden. Dies gelang erst durch besondere Bänderungs-Techniken an Nicht-Interphasekernen. So ruft der fluoreszierender Farbstoff (Fluorochrom) Quinacrin ein Bandenmuster hervor, das die einzelnen Chromosomenpaare identifiziert.[9] Charakteristische Bandenmuster entstehen auch, wenn die DNA in den Chromosomen etwas denaturiert wird.[10][11] Ein genormtes Kürzelsystem bezeichnet jeden Abschnitt der technisch gebänderten Chromosomen.[12] Das p steht für petit (franz. klein) und bezeichnet den kurzen Arm eines Chromosoms. Für den langen Arm wählte man den nachfolgenden Buchstaben q. So können nicht nur anomale Chromosomenzahlen (wie Trisomie 21) festgestellt, sondern auch strukturelle Chromosomenänderungen (Deletionen, Duplikationen, Translokationen) erfasst werden.
Genaktivität und Replikation
Um die Interphase im Zellzyklus zu studieren, bot man lebenden Zellen radioaktiv markierte Moleküle an. Einbau von 3H-Uridin, spezifisch für RNA, wies die Genexpression aktiver Chromosomenorte nach.[13] Einbau von 3H-Thymidin, spezifisch für DNA, zeigte die chromosomale DNA-Synthese in der S-Phase.[14] Als Betastrahlung hinterließen Elektronen jeweils ihre Spuren als Autoradiografie in einem den Zellen aufgelegten Film.
- In späteren Replikationsanalysen ersetzte Bromdesoxyuridin das radioaktiv markierte Thymidin.
- Fortgeschrittene Gentechnik machte auch bei Fragen zur Genaktivität radioaktive Substanzen überflüssig. Dabei verlagert sich der Nachweis von der Transkriptionsebene (RNA-Synthese) auf die vollendete Translation: Ort und Zeit der Aktivierung eines genspezifischen Promotors werden durch das vom Reportergen hervorgerufene Protein angezeigt. Als Voraussetzung muss die DNA-Sequenz des Reportergens hinter die Promotorsequenz des zu untersuchenden Gens gehängt werden. Mit diesem DNA-Konstrukt wird ein Organismus transformiert. Beliebt ist das Reportergen, welches das Grün fluoreszierende Protein (GFP) kodiert.[15] Mit diesem System kann man Genexpression in Zellen, Geweben und Organen verfolgen.
Restriktionsenzyme
Die Isolierung zahlreicher DNA-Restriktasen bedeutete für die Zytogenetik und für die Genetik allgemein einen revolutionären, nachhaltigen Durchbruch. Diese meist aus Bakterien gewonnenen Enzyme sind molekulare Scheren: Sie zerschneiden DNA an kurzen, definierten Basensequenzen.[16][17][18][19] Die Restriktasen und die Polymerase-Kettenreaktion (PCR) sind die beiden Pfeiler der modernen molekularen Genetik und Gentechnik.[20] Sie ermöglichen, ganze Genome zu sequenzieren oder verschiedene Arten der Fluoreszenz-in-situ-Hybridisierung (FISH) durchzuführen. Für letztere werden DNA- oder RNA-Proben mit Fluorochromen markiert und auf komplementäre DNA-Sequenzen der angepeilten Chromosomen hybridisiert. Zur Diagnose ist ein Fluoreszenzmikroskop erforderlich.
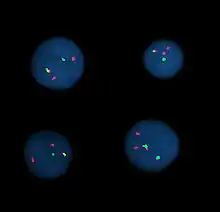
Mit FISH bemalte Chromosomen
Für das „Bemalen“ werden gleichzeitig mehrere Sonden (englisch probes), mit unterschiedlichen Fluorochromen markiert, auf Metaphasen hybridisiert. So erhält jedes Chromosomenpaar im Karyotyp durch das Chromosome painting einen eigenen Farbton.[21] Zu diesem Zweck gibt es zwei Verfahren.
- Mehrfarben (multiplex) Fluoreszenz-in-situ-Hybridisierung (M-FISH). Von einer mit verschiedenen Fluorochromen dekorierten Metaphase werden entsprechend viele Digitalbilder aufgenommen. Ein Computerprogramm analysiert die Originalaufnahmen und vereinigt sie in einem einzigen Bild, das die Chromosomenpaare in gut unterscheidbaren Falschfarben darstellt.[22] Die Methode ist auch bestens geeignet, Chromosomenmutationen in Interphase-Zellkernen darzustellen (Bild: Interphasen-Zytogenetik).
- Spectral karyotyping (SKY). Diese Art des Chromosome painting erfordert leistungsstarke Rechner. Denn von jedem Pixel des Digitalbildes werden zwei Teilstrahlen hergestellt, welche mittels Interferometer mit Gangunterschied zur Interferenz gebracht werden. Eine Fourier-Transformation liefert aus dem Interferogramm genaue Daten über das ursprüngliche Fluoreszentspektrum. Von diesem erzeugt ein nachgeschaltetes Computerprogramm wiederum gut unterscheidbare Falschfarben für die einzelnen Chromosomenpaare im Karyotyp.[23] Als Fourier-Spektroskopie bietet SKY gegenüber M-FISH den Vorteil größerer Abstände zwischen chromosomalen Signalen und Rauschen.[24]
Duale Hybridisierung
Die Chromogene in-situ Hybridisierung (CISH, auch: Duale ISH) ist eine günstige Alternative für FISH. Sie wird angewendet bei der Suche nach einer bestimmten Mutation auf einem bestimmten Chromosom. Gegenüber FISH bietet CISH mehrere Vorteile: Verwendung von zwei stabilen, absorbierenden Farben, die nicht bleichen; Diagnose am Hellfeld-Mikroskop, damit schneller und billiger. Fragestellung bei Brustkrebs und anderen soliden Tumoren: Ist das Gen HER2 auf Chromosom 17q12 für den Rezeptor 2 des humanen epidermalen Wachstumsfaktors amplifiziert? Dazu wird die HER2-DNA-Probe mit der ersten Farbe hybridisiert, und zwar auf einen Zellkern in Interphase. Mit zweiter Farbe wird gleichzeitig CEN-17 als Referenz-DNA für das Zentromer in Chromosom 17 hybridisiert. Das Verhältnis 2 oder größer bedeutet HER2-Amplifikation, verbunden mit schlechter medizinischer Prognose.[25][26]
Genome im Vergleich
Comparative Genomic Hybridisation (CGH) entdeckt quantitative Sequenzänderungen in einem Genom empfindlicher als FISH. Für die CGH wird die DNA aus Zellen, die untersucht werden sollen, mit einem ersten Fluorochrom markiert. Ebenso markiert man DNA aus normalen Zellen mit einem zweiten Fluorochrom unterschiedlicher Emissionswellenlänge. In gleichen Mengen werden Proben-DNA und Referenz-DNA gleichzeitig auf gespreitete, normale Metaphasen hybridisiert. „Normal“ bedeutet einen, soweit bekannt, gesunden Organismus als Spender. Bei der CGH „streiten“ (kompetitieren) die beiden DNAs um dieselben Bindungsstellen in den Ziel-Chromosomen. Entlang der Metaphase-Chromosomen wird die komplexe Fluoreszenz gemessen. Stimmen beide DNA-Proben überein, ist ihr Verhältnis 1,0. Das erste Fluorochrom (an die Proben-DNA gebunden) ergibt bei Duplikationen in einem Chromosom den Wert 1,5. Sind beide homologen Chromosomen an derselben Stelle verdoppelt, ist der den Wert 2,0. Bei Deletionen in einem Chromosom (Verlust der Heterozygotie) ist der Wert 0,5. Sind beide homologen Chromosomen deletiert, ist der Wert 0,0 (kein Signal vom ersten Fluorochrom).[27][28][29] Die molekulare Zytogenetik, auch die CGH, kann einen einzelnen Zellkern analysieren[30] oder die DNA-Unterschiede der beiden geschlechtsgekoppelten Chromosomen offenbaren.[31]
Zellkerne und Zahlen
Simples Zählen
Die Anzahl mitotischer oder meiotischer Chromosomen ist für eine Art und ihre Individuen konstant: „Die Geschlechtszellen (Eier oder Spermatozoën) eines Organismus enthalten halb so viele Chromosomen als die erste Embryonalzelle, aus welcher dieser Organismus entstanden ist.“[32] Diese Zahlenkonstanz ist ein Artmerkmal.[33] Die einfache (haploide) Anzahl n wird in der Regel von 2n Chromosomen einer mitotischen Metaphase abgeleitet. Denn in den tatsächlich haploiden Kernen der Geschlechtszellen sind die Chromosomen nicht unmittelbar zu zählen. Allerdings zeigt Meiose I, wenn die Bivalente durch Chiasmata paaren, 1n Chromosomen.
Längen messen
„Die Chromosomen sind grundsätzlich bilaterale symmetrische oder asymmetrische Gebilde.“[34] Dieser Satz inspirierte zu vielen Messungen, um die Länge von Chromosomen sowie das Verhältnis ihrer kurzen zu ihren langen Armen zu bestimmen. So klassifiziert die Lage des Kinetochors ein symmetrisches Chromosom als metazentrisch und ein asymmetrisches als submetazentrisch, subtelozentrisch oder akrozentrisch. Ebenso ist die Lage einer sekundären Einschnürung als Nukleolusorganisatorregion numerisch zu definieren. Solche Längenmessungen erlauben, die Identität eines Chromosoms in einem Karyotyp mit relativ wenigen Elementen zu diagnostizieren.
Zellkerne wiegen
- Mikrofotometrie (auch Fotomikrometrie, Mikroskopfotometrie oder Zytometrie). Diese Technik wird vorwiegend dazu verwendet, den DNA-Gehalt von ganzen Zellkernen und auch ihrer Chromosomen zu bestimmen. Am praktikabelsten erwies sich die Arbeit im sichtbaren Spektrum. Nachdem die Kern-DNA mit der Feulgen-Prozedur gefärbt ist, wird im Absorptionsmaximum (560 nm) gemessen. Um die resultierenden Absorptionseinheiten in Pikogramm (pg) oder Megabasenpaaren (Mbp) umzurechnen, sind nebenher Referenzkerne mit bekanntem DNA-Gehalt zu registrieren.[35] Das Verfahren ist zeitaufwändig. Es erfordert sorgfältige Präparation auf gläsernen Objektträgern (engl. slide based microphotometry), bietet aber den Vorteil, Messungen wiederholen und zusammenhängende Gewebeteile und Zelltypen beurteilen zu können. Eine Pionierarbeit definierte den Wert 1 C als DNA-Gehalt des (haploiden) Genoms einer Art.[36]
- Durchflusszytometrie (auch Impulszytofotometrie). Für dieses Verfahren wird der Zellverband eines Gewebes gänzlich aufgelöst, die Zellen oder ihre Kerne meist mit Fluorochromen gefärbt. Es bietet den Vorteil, in kurzer Zeit große Stichproben zu analysieren.[37]
Literatur
- Peter S. Harper: First Years of Human Chromosomes: The Beginnings of Human Cytogenetics. Cold Spring Harbor Laboratory Press, Cold Spring Harbor 2006. ISBN 1904842240.
- Walter Nagl: Chromosomen: Organisation, Funktion und Evolution des Chromatins. Parey, Berlin 2.1980. ISBN 3-489-60234-X.
- M. Schmid, Indrajit Nanda (Hrsg.): Chromosomes today, volume 14. Springer, Berlin 2004. ISBN 978-94-017-1033-6 (eBook); ISBN 978-1-4020-0091-1 (Hardcover); ISBN 978-90-481-5855-3 (Softcover).
- Jürgen Schulz-Schaeffer: Cytogenetics: Plants, Animals, Humans. Reprint der Erstauflage 1980; Springer, New York 2011. ISBN 978-1-4612-6062-2.
- Adrian T. Sumner: Chromosomes: Organization and function. Blackwell Science, Oxford 2003. ISBN 0-632-05407-7.
- Eeva Therman, Millard Susman: Human chromosomes: Structure, behavior, and effects. Springer, Berlin, New York 3.1993. ISBN 3-540-97871-2, ISBN 0-387-97871-2.
- Michael Theile, Siegfried Scherneck: Zellgenetik. Akademie-Verlag, Berlin 1978. ISBN 3-528-06836-1.
- Walther Traut: Chromosomen: Klassische und molekulare Cytogenetik. Springer, Berlin 1991. ISBN 3-540-53319-2.
- Michael James Denham White: The Chromosomes. Chapman & Hall, London, 6.1973. ISBN 0-412-11930-7.
Zeitschriften
- Chromosoma. Biology of the Nucleus. ISSN 0009-5915 (Druck), ISSN 1432-0886 (Online).
- Chromosome Research. High quality papers on all aspects of chromosome and nuclear biology. ISSN 0967-3849 (Druck), ISSN 1573-6849 (Online).
- Cytogenetic and Genome Research. ISSN 1424-8581 (Druck), ISSN 1424-859X (Online).
- Human Genetics. ISSN 0340-6717 (Druck), ISSN 1432-1203 (Online).
Weblinks
- Links zum Chromosome painting: feedforbiotech.blogspot.de, mun.ca
- Durchflusszytometrie, Beispiel Kiel
Einzelnachweise
- Ingo Schubert, J. L. Oud: There is an upper limit of chromosome size for normal development of an organism. In: Cell. Band 88, 1997, S. 515–520.
- Hans von Winiwarter: Études sur la spermatogenese humaine. In: Archives de Biologie (Liege) Band 27, Nr. 93, 1912, S. 147–149.
- Theophilus S. Painter: Studies in mammalian spermatogenesis, II. The spermatogenesis of man. In: Journal of Experimental Zoology Band 37, 1923, S. 291–336, 1923.
- T. C. Hsu: Mammalian chromosomes in vitro, I. The karyotype in man. (PDF) In: Journal of Heredity Band 43, 1952, S. 167–172.
- Joe Hin Tjio, Albert Levan: The chromosome number of man. In: Hereditas Band 42, 1956, S. 1–6.
- Theodor Boveri: Über mehrpolige Mitosen als Mittel zur Analyse des Zellkerns. In: Verh Phys-Med Ges Würzburg Band NF 35, 1902, S. 67–90, S. 81.
- Walter Sutton Stanborough: The chromosomes in heredity. In: Biol Bull Marine Biol Labor Woods Hole (Mass.) Band 4, 1902 (oder 1903), S. 231–248.
- Rüdiger G. Steinbeck, Gert U. Auer, Anders D. Zetterberg: Reliability and significance of DNA measurements in interphase nuclei and division figures in histological sections. In: European Journal of Cancer Band 35, Nr. 5, 1999, S. 87–795.
- Torbjörn Caspersson, Lore Zech, C. Johansson: Differential binding of alkylating fluorochromses in human chromosomes. In: Experimental Cell Research Band 60, 1970, S. 315–319.
- Wolfgang Schnedl: Analysis of the human karyotype using a reassociation technique. In: Chromosoma Band 34, 19971, S. 448–454.
- J. J. Yunis: High resolution of human chromosomes. In: Science. Band 191, Nummer 4233, März 1976, S. 1268–1270, PMID 1257746. doi:10.2307/1741162 (zurzeit nicht erreichbar).
- L. G. Shaffer, J. McGowan-Jordan, M. Schmid (Hrsg.): ISCN 2013. An international system for human cytogenetic nomenclature (2013): Recommendations of the international standing committee on human cytogenetic nomenclature, published in collaboration with "Cytogenetic and Genome Research" plus fold-out: "The Normal Human Karyotype G- and R-bands". Karger, Basel 2012. ISBN 978-3-318-02253-7.
- Claus Pelling: Chromosomal synthesis of ribonucleic acid as shown by incorporation of Uridine labelled with tritium. (PDF) In: Nature Band 184, 4686, 1959, S. 655–656, 1959.
- James Herbert Taylor, Philip S. Woods, Walter L. Hughes: The organization and duplication ofNr. chromosomes as revealed by autoradiographic studies using tritium-labeled thymidine. In: PNAS Band 43, 1957, S. 122–128. PMC 528395 (freier Volltext)
- S. R. Kain, M. Adams, A. Kondepudi, T. T. Yang, W. W. Ward, P. Kitts: Green fluorescent protein as a reporter of gene expression and protein localization. In: BioTechniques Band 19, Nr. 4, 1995, S. 650–655.
- Werner Arber, S. Linn: DNA modification and restriction. In: Annual Review of Biochemistry Band 38, 1969, S. 467–500. ISSN 0066-4154 (Print), ISSN 1545-4509 (Electronic).
- Hamilton O. Smith, K. W. Wilcox: A restriction enzyme from Hemophilus influenzae, I. Purification and general properties. In: Journal of Molecular Biology Band 51, Nr. 2, 1970, S. 379–391. ISSN 0022-2836 (Print), ISSN 1089-8638 (Electronic).
- K. Danna, Daniel Nathans: Specific cleavage of simian virus 40 DNA by restriction endonuclease of Hemophilus influenzae. In: PNAS Band 68, Nr. 12, 1971, S. 2913–2917. PMC 389558 (freier Volltext)
- Werner Arber: Restriction endonucleases. In: Angewandte Chemie (Internationale Ausgabe in Englisch) Band 17, Nr. 2, 1978, S. 73–79. ISSN 1433-7851 (Print), ISSN 1521-3773 (Electronic).
- Richard J. Roberts: How restriction enzymes became the workhorses of molecular biology. In: PNAS Band 102, Nr. 17, 2005, S. 5905–5908. PMC 1087929 (freier Volltext)
- Thomas Ried, Evelin Schröck, Yi Ning, Johannes Wienberg: Chromosome painting: a useful art. In: Human Molecular Genetics Band 7, Nr. 10, 1998, S. 1619–1626.
- Michael R. Speicher, David C. Ward: The coloring of cytogenetics. In: Nature Medicine Band 2, Nr. 9, 1996, S. 1046–1048, 1996. doi:10.1038/nm0996-1046.
- Evelin Schröck, S. du Manoir, T. Veldman, B. Schoell, J. Wienberg, M. A. Ferguson-Smith, Y. Ning, D. H. Ledbetter, I. Bar-Am, D. Soenksen, Y. Garini, Thomas Ried: Multicolor spectral karyotyping of human chromosomes. In: Science Band 273, Nr. 5274, 1996, S. 494–497. doi:10.1126/science.273.5274.494.
- H. Parsche, K. Luchner: Moderne Methoden der Spektralanalyse. In: Physik in unserer Zeit Band 6, 1975, S. 151–161. Online ISSN 1521-3943.
- Lim Sung-Jig, Alegria Cantillep, Philip M. Carpenter: Validation and workflow optimization of human epidermal growth factor receptor 2 testing using INFORM HER2 dual-color in situ hybridization. In: Human Pathology Band 44, Nr. 11, 2013, S. 2590–2596.
- J. Mollerup, U. Henriksen, S. Müller, A. Schønau: Dual color chromogenic in situ hybridization for determination of HER2 status in breast cancer: A large comparative study to current state of the art fluorescence in situ hybridization. In: BioMed Central Clinical Pathology Band 12, 2012, S. 3.
- Anne Kallioniemi, Olli-P. Kallioniemi, Damir Sudar, Denis Rutovitz, Joe W. Gray, Fred Waldman, Dan Pinkel: Comparative genomic hybridization for molecular cytogenetic analysis of solid tumors. In: Science Band 258, Nr. 5083, 1992, S. 818–821.
- Olli-P. Kallioniemi, Anne Kallioniemi, Damir Sudar, Denis Rutovitz, Joe W. Gray, Fred Waldman, Dan Pinkel: Comparative genomic hybridization: A rapid new method for detecting and mapping DNA amplification in tumors. In: Seminars in Cancer Biology Band 4, 1993, S. 41–46.
- Evelin Schröck et al.: Comparative genomic hybridization of human malignant gliomas reveals multiple amplification sites and nonrandom chromosomal gains and losses. In: The American Journal of Pathology Band 144, Nr. 6, 1994, S. 1203–1218. PMC 1887475 (freier Volltext)
- Christoph A. Klein, O. Schmidt-Kittler, J. A. Schardt, K. Pantel, M. R. Speicher, Gert Riethmüller: Comparative genomic hybridization, loss of heterozygosity, and DNA sequence analysis of single cells. In: Proceedings of the National Academy of Sciences USA Band 96, Nr. 8, 1999, S. 4494–4499. PMC 16360 (freier Volltext)
- Walther Traut, Ulrike Eickhoff, Jan-C. Schorch: Identification and analysis of sex chromosomes by comparative genomic hybridization (CGH). In: Methods in Cell Science Band 23, 2001, S. 155–161.
- Theodor Boveri: Zellenstudien (3): Über das Verhalten der chromatischen Kernsubstanz bei der Bildung der Richtungskörper und bei der Befruchtung. In: Jenaische Zeitschrift für Naturwissenschaft Band 24, 1890, S. 314–401. Dort S. 372f.
- Georg Tischler: Chromosomenzahl – Form und Individualität im Pflanzenreich. In: Fortschritte der Botanik Band 5, 1915, S. 172–284.
- Emil Heitz: Der bilaterale Bau der Geschlechtschromosomen und Autosomen bei Pellia fabbroniana, P. epiphylla und einigen anderen Jungermanniaceen. In: Planta Band 5, 1928, S. 725–768. Dort S. 764.
- Helmut Zacharias, Boris Anokhin, Konstantin Khalturin, Thomas C. G. Bosch: Genome sizes and chromosomes in the basal metazoan Hydra. In: Zoology Band 107, 2004, S. 219–227.
- Hewson Swift: The constancy of desoxyribose nucleic acid in plant nuclei. In: PNAS Band 36, Nr. 11, 1950, S. 643–654. PMC 1063260 (freier Volltext)
- J. Kusenda, M. Fajtova, A. Kovarikova: Monitoring of minimal residual disease in acute leukemia by multiparametric flow cytometry. In: Neoplasma Band 61, Nr. 2, 2014, S. 119–127. doi:10.4149/neo_2014_017. Siehe Menü: 2014.