2A-Peptide
2A-Peptide sind eine Klasse von viralen Strukturmotiven, welche in Proteinen vorkommen und diese während der Translation teilen.[1]
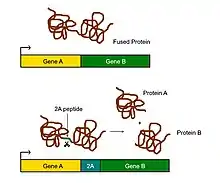
Eigenschaften
Die erste 2A-Peptid-Sequenz wurde im Maul-und-Klauenseuche-Virus (als Übersetzung von engl. Foot-and-mouth disease virus, abgekürzt FMDV) entdeckt. 2A-Peptide wurden in verschiedenen Säugetierviren (der Familie der Picornaviren) und vielen Insektenviren beschrieben.[2] Die Benennung erfolgte nach dem Namen des Gens einer Peptidase namens 2Apro des Virus, in dem es erstmals gefunden wurde.[3] So stammt das P2A-Peptid vom Porzinen Teschovirus 1 (als Übersetzung von engl. porcine teschovirus-1, abgekürzt PTV1). Hier führt die 2A-Sequenz zur cotranslationalen Teilung von zwei Proteinen innerhalb eines einzelnen offenen Leserahmens (als Übersetzung von engl. open reading frame, abgekürzt ORF). Es wird angenommen, dass die 2A-Sequenz die Ausbildung einer regulären Peptidbindung am Ribosom inhibiert und es folglich zur Trennung der Peptide während der Translation kommt.[1][2] Vermutlich wird während der Translation am Ribosom die Bildung einer Gly-Pro-Bindung C-terminal am 2A-Peptid übersprungen.[4][5] Der genaue Mechanismus ist noch unbekannt.[6] Die Spaltung erfolgt stets vor dem C-terminalen Prolin des 2A-Peptids. Daher erhält das zweite (C-terminale) Peptid ein Prolin als N-Terminus und das erste (N-terminale) Peptid die restliche 2A-Sequenz als C-Terminus.[2]
Anwendung
Gentechnisch werden 2A-Peptide beim Vektordesign eingesetzt, um ein Protein nach der Expression an gezielter Stelle in zwei Proteine zu teilen.[7] Dies wird als polycistronische Expression bezeichnet. Ihr Vorteil gegenüber den analog verwendeten IRES Sequenzen ist die äquimolare Expression beider Proteine von einem gemeinsamen Promotor,[1][8] während bei IRES das zweite Protein in geringerer Menge gebildet wird.[9] Es wurde eine Genexpression von bis zu fünf Proteinen mit vier dazwischenliegenden 2A-Peptiden aus einem Gen beschrieben.[9] Von verschiedenen untersuchten 2A-Peptiden war P2A das 2A-Peptid mit der effizientesten Spaltung, während F2A das mit der geringsten Effizienz war.[10] Bei F2A können bis zu 50 % der Fusionsproteine ungespalten bleiben, wodurch unbeabsichtigte neue Funktionen entstehen können.[11] 2A-Peptide können neben Säugetier- und Insektenzellen auch in Saccharomyces cerevisiae,[12] Kokzidien[13] und (mit Modifikationen) in Pflanzen verwendet werden.[14][15] Das C-terminale Protein sollte keine Myristoylierung aufweisen, da durch 2A-Peptide dann die zelluläre Lokalisation verändert werden kann.[16] Die Trennung der Proteine kann durch Transfektion eines Vektors, das für ein Protein-2A-Peptid-Protein codiert, in Zellkulturen mit nachfolgendem Western Blot überprüft werden.[17] Da nach der Spaltung der 2A-Peptide kurze Peptidsequenzen an den Termini der getrennten Proteine zurückbleiben, kann es zu funktionellen Störungen dieser Proteine kommen.[1]
Vertreter
Die nachfolgende Tabelle listet die gentechnologisch relevantesten Vertreter der 2A-Peptide auf. Ein „GSG“-Linker (Gly-Ser-Gly) N-terminal vor dem 2A-Peptid kann die Effektivität zudem verstärken.[1] 2A-Peptide besitzen die Konsensussequenz DxExNPGP.[18]
| Bezeichnung | Sequenz |
|---|---|
| T2A[8] | EGRGSLLTCGDVEENPG'P |
| P2A[8] | ATNFSLLKQAGDVEENPG'P |
| E2A[8] | QCTNYALLKLAGDVESNPG'P |
| F2A[1] | VKQTLNFDLLKLAGDVESNPG'P |
Einzelnachweise
- A. L. Szymczak-Workman, K. M. Vignali, D. A. A. Vignali: Design and Construction of 2A Peptide-Linked Multicistronic Vectors. In: Cold Spring Harbor Protocols. 2012, Nr. 2, 2012, ISSN 1559-6095, S. 199–204. doi:10.1101/pdb.ip067876. PMID 22301656.
- G. A. Luke, P. de Felipe, A. Lukashev, S. E. Kallioinen, E. A. Bruno, M. D. Ryan: Occurrence, function and evolutionary origins of '2A-like' sequences in virus genomes. In: The Journal of general virology. Band 89, Pt 4April 2008, S. 1036–1042, doi:10.1099/vir.0.83428-0, PMID 18343847, PMC 2885027 (freier Volltext).
- M. D. Ryan, A. M. King, G. P. Thomas: Cleavage of foot-and-mouth disease virus polyprotein is mediated by residues located within a 19 amino acid sequence. In: The Journal of general virology. Band 72 ( Pt 11), November 1991, S. 2727–2732, doi:10.1099/0022-1317-72-11-2727, PMID 1658199.
- M. L. Donnelly, G. Luke, A. Mehrotra, X. Li, L. E. Hughes, D. Gani, M. D. Ryan: Analysis of the aphthovirus 2A/2B polyprotein 'cleavage' mechanism indicates not a proteolytic reaction, but a novel translational effect: a putative ribosomal 'skip'. In: The Journal of general virology. Band 82, Pt 5Mai 2001, S. 1013–1025, doi:10.1099/0022-1317-82-5-1013, PMID 11297676.
- P. Sharma, F. Yan, V. A. Doronina, H. Escuin-Ordinas, M. D. Ryan, J. D. Brown: 2A peptides provide distinct solutions to driving stop-carry on translational recoding. In: Nucleic acids research. Band 40, Nummer 7, April 2012, S. 3143–3151, doi:10.1093/nar/gkr1176, PMID 22140113, PMC 3326317 (freier Volltext).
- Y. Wang, F. Wang, R. Wang, P. Zhao, Q. Xia: 2A self-cleaving peptide-based multi-gene expression system in the silkworm Bombyx mori. In: Scientific Reports. Band 5, November 2015, S. 16273, doi:10.1038/srep16273, PMID 26537835, PMC 4633692 (freier Volltext).
- A. L. Szymczak, D. A. Vignali: Development of 2A peptide-based strategies in the design of multicistronic vectors. In: Expert Opinion on Biological Therapy. Band 5, Nummer 5, Mai 2005, S. 627–638, doi:10.1517/14712598.5.5.627, PMID 15934839.
- Ziqing Liu, Olivia Chen, J. Blake Joseph Wall, Michael Zheng, Yang Zhou, Li Wang, Haley Ruth Vaseghi, Li Qian, Jiandong Liu: Systematic comparison of 2A peptides for cloning multi-genes in a polycistronic vector. In: Scientific Reports. 7, Nr. 1, 2017, ISSN 2045-2322, S. 2193. bibcode:2017NatSR...7.2193L. doi:10.1038/s41598-017-02460-2. PMID 28526819. PMC 5438344 (freier Volltext).
- G. A. Luke, M. D. Ryan: "Therapeutic applications of the 'NPGP' family of viral 2As". In: Reviews in Medical Virology. Band 28, Nummer 6, 11 2018, S. e2001, doi:10.1002/rmv.2001, PMID 30094875.
- J. H. Kim, S. R. Lee, L. H. Li, H. J. Park, J. H. Park, K. Y. Lee, M. K. Kim, B. A. Shin, S. Y. Choi: High cleavage efficiency of a 2A peptide derived from porcine teschovirus-1 in human cell lines, zebrafish and mice. In: PLOS ONE. Band 6, Nummer 4, 2011, S. e18556, doi:10.1371/journal.pone.0018556, PMID 21602908, PMC 3084703 (freier Volltext).
- S. Velychko, K. Kang, S. M. Kim, T. H. Kwak, K. P. Kim, C. Park, K. Hong, C. Chung, J. K. Hyun, C. M. MacCarthy, G. Wu, H. R. Schöler, D. W. Han: Fusion of Reprogramming Factors Alters the Trajectory of Somatic Lineage Conversion. In: Cell Reports. Band 27, Nummer 1, 04 2019, S. 30–39.e4, doi:10.1016/j.celrep.2019.03.023, PMID 30943410.
- T. M. Souza-Moreira, C. Navarrete, X. Chen, C. F. Zanelli, S. R. Valentini, M. Furlan, J. Nielsen, A. Krivoruchko: Screening of 2A peptides for polycistronic gene expression in yeast. In: FEMS yeast research. Band 18, Nummer 5, 08 2018, S. , doi:10.1093/femsyr/foy036, PMID 29617770.
- X. Tang, X. Liu, G. Tao, M. Qin, G. Yin, J. Suo, X. Suo: "Self-cleaving" 2A peptide from porcine teschovirus-1 mediates cleavage of dual fluorescent proteins in transgenic Eimeria tenella. In: Veterinary research. Band 47, Nummer 1, 06 2016, S. 68, doi:10.1186/s13567-016-0351-z, PMID 27352927, PMC 4924277 (freier Volltext).
- S. Burén, C. Ortega-Villasante, K. Otvös, G. Samuelsson, L. Bakó, A. Villarejo: Use of the foot-and-mouth disease virus 2A peptide co-expression system to study intracellular protein trafficking in Arabidopsis. In: PLOS ONE. Band 7, Nummer 12, 2012, S. e51973, doi:10.1371/journal.pone.0051973, PMID 23251667, PMC 3522588 (freier Volltext).
- B. Zhang, M. Rapolu, S. Kumar, M. Gupta, Z. Liang, Z. Han, P. Williams, W. W. Su: Coordinated protein co-expression in plants by harnessing the synergy between an intein and a viral 2A peptide. In: Plant biotechnology journal. Band 15, Nummer 6, Juni 2017, S. 718–728, doi:10.1111/pbi.12670, PMID 27879048, PMC 5425387 (freier Volltext).
- S. Hadpech, W. Jinathep, S. Saoin, W. Thongkum, K. Chupradit, U. Yasamut, S. Moonmuang, C. Tayapiwatana: Impairment of a membrane-targeting protein translated from a downstream gene of a "self-cleaving" T2A peptide conjunction. In: Protein expression and purification. Band 150, 10 2018, S. 17–25, doi:10.1016/j.pep.2018.05.002, PMID 29733907.
- A. L. Szymczak-Workman, K. M. Vignali, D. A. Vignali: Verification of 2A peptide cleavage. In: Cold Spring Harbor protocols. Band 2012, Nummer 2, Februar 2012, S. 255–257, doi:10.1101/pdb.prot067892, PMID 22301658.
- X. Yang, A. Cheng, M. Wang, R. Jia, K. Sun, K. Pan, Q. Yang, Y. Wu, D. Zhu, S. Chen, M. Liu, X. X. Zhao, X. Chen: Structures and Corresponding Functions of Five Types of Picornaviral 2A Proteins. In: Frontiers in Microbiology. Band 8, 2017, S. 1373, doi:10.3389/fmicb.2017.01373, PMID 28785248, PMC 5519566 (freier Volltext).