F420
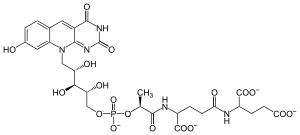
F420 ist ein Cofaktor, eine chemische Verbindung, die im Zytoplasma methanogener Archaeen, mancher Bakterien und einzelliger Eukaryoten vorkommt. Es handelt sich biochemisch um Elektronentransporter, und chemisch um Deazaflavine, ähnlich dem Riboflavin. Sie unterscheiden sich in der Länge der Polyglutamat-Kette, die bei den Mycobakterien fünf bis sieben Glu-Reste enthält. Ein Mitglied der Stoffgruppe wurde erstmals 1972 isoliert. Die chemische Struktur wurde 1978 aufgeklärt. Der Kofaktor verdankt seinen Namen seiner starken Lichtabsorption bei einer Wellenlänge λmax = 420 nm.[2][3][4]
| Strukturformel | ||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
 | ||||||||||||||||||||||
| Allgemeines | ||||||||||||||||||||||
| Name | F420 | |||||||||||||||||||||
| Andere Namen |
| |||||||||||||||||||||
| Summenformel | C29H32N5O18P4− | |||||||||||||||||||||
| Externe Identifikatoren/Datenbanken | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Eigenschaften | ||||||||||||||||||||||
| Molare Masse | 769,6 g·mol−1 | |||||||||||||||||||||
| Sicherheitshinweise | ||||||||||||||||||||||
| ||||||||||||||||||||||
| Soweit möglich und gebräuchlich, werden SI-Einheiten verwendet. Wenn nicht anders vermerkt, gelten die angegebenen Daten bei Standardbedingungen. | ||||||||||||||||||||||
Optische Eigenschaften
Oxidiertes F420 absorbiert bei 420 nm, nach Absorption wird Licht bei 520 nm emittiert.[3] Am isosbestischen Punkt bei 401 nm besitzt der Kofaktor einen Extinktionskoeffizient von 25,9 mM−1cm−1. Nach Reduktion (F420H2) verliert F420 sein Absorptionsmaximum bei 420 nm und dieses verschiebt sich auf 320 nm, jedoch mit einem geringeren Extinktionskoeffizient.[5]
Biologische Bedeutung
F420 besitzt zwar eine ähnliche Struktur wie Riboflavin bzw. FAD, chemisch gesehen ähnelt es aber eher den Nikotinamiden, wie z. B. NADP+. Das Redoxpotential von F420 liegt bei −340 mV und ähnelt demnach dem des es NAD(P)s (−320 mV). F420 überträgt ausschließlich ein Hydridion (zwei Elektronen und ein Proton) – wie auch NAD+ bzw. NADP+.[6]
Die Grundstruktur, das 7,8-Didemethyl-8-hydroxy-5-deazariboflavin-5'-phosphat, kommt in Archaeen, aber auch in Gram-positiven Eubakterien wie Streptomyces oder Mycobacteria vor.[7] Der Kofaktor wurde auch im Cyanobakterium Anacystis nidulans[8] und in der (eukaryotischen) Grünalge Scenedesmus acutus[9] entdeckt. Jedoch variiert die Grundstruktur in jenen Organismen.
F420 ist beteiligt an Prozessen der Methanogenese,[10] der Sulfitreduktion,[11] der Sauerstoffentgiftung[12] und dem Elektronentransport[13] in Archaea.
Es ist ein Kofaktor in der Antibiotikasynthese in Streptomyceten sowie für die Reduktion von Stickstoffdioxid und PA-824 in Mycobakterien, wo es durch die Glucose-6-phosphat-Dehydrogenase wieder zurückreduziert wird.[14][15][16] PA-824 ist ein experimentelles Medikament zur Behandlung von Tuberkulose.
F420-abhängige Enzyme
F420 wird häufig als Elektronenüberträger in der Methanogenese verwendet, er tritt aber auch bei anderen Prozessen hervor:
Tetrahydromethanopterin-abhängiges Enzym
Während der Methanogenese – ausgehend von CO2 – spielen Tetrahydromethanopterin-abhängige Enzyme eine zentrale Rolle. Die F420-abhängige N5,N10-Methylentetrahydromethanopterin-Dehydrogenase (EC 1.5.99.9) reduziert an Methanopterin gebundenes Methenyl zu Methylen.[17][18] Dabei wird (reduziertes) F420H2 verbraucht (vgl. Gleichung 1).
Die F420-abhängige N5,N10-Methylentetrahydromethanopterin-Reduktase (EC 1.5.99.11) reduziert unter Verbrauch von F420H2 das an Methanopterin gebundene Methylen weiter zu Methyl (vgl. Gleichung 2):
F420-reduzierende Hydrogenase
Für die Regenerierung von oxdierten F420 wird ein Enzym benötigt, was man als F420-reduzierende Hydrogenase (EC 1.12.98.1) bezeichnet.[19] Das Enzym ist entweder häufig membrangebunden oder vereinzelt auch im Cytoplasma lokalisiert.[20]
NADP/F420 Oxidoreduktase
Die Übertragung von 2 Reduktionsäquivalenten von F420H2 auf NADP+ wird durch eine Transhydrogenase katalysiert, einer NADP/F420 Oxidoreduktase.[21] NADPH selbst wird in methanogenen Bakterien für die Synthese gewisser zellulärer Metaboliten benötigt, daneben aber auch bei NADPH-abhängigen Alkoholdehydrogenasen.[22]
Formiatdehydrogenease
Manche methanogene Organismen können Reduktionsäquivalente durch Oxidation von Ameisensäure gewinnen. Da die Oxidation mit der Reduktion von F420 einhergeht, wird so F420 wieder regeneriert. Dieses Enzym wurde bei Methanobacterium formicicum bereits gereinigt und in E. coli exprimiert.[23]
Alkoholdehydrogenase
Isopropanol bzw. Ethanol werden von verschiedenen methanogenen Organismen als alternative Elektronenquelle für die Reduktion von CO2 verwendet. So wird in methanogenen Archaeen Isopropanol von einer F420-abhängigen sekundären Alkoholdehydrogenase unter Verbrauch reduzierten F420 zu Aceton oxidiert.[24]
Pharmakologische Bedeutung
Bei einem in silico-Screening nach F420-abhängigen Enzymen wurden in M. tuberculosis eine überraschend hohe Zahl von Kandidaten entdeckt. Wenngleich diese Enzyme biochemisch noch nicht charakterisiert sind, könnten sie ein pharmakologisches Target darstellen, da in der Darmflora fast keine Bakterien mit solchen Enzymen vorhanden sind, und Antibiotika gegen M. tuberculosis auf der Grundlage der Hemmung von F420-abhängigen Enzymen daher kaum Nebenwirkungen auf die Darmflora hätten.[25]
Siehe auch
Literatur
- RH. White: Biosynthesis of the methanogenic cofactors. In: Vitam Horm., 2001, 61, S. 299–337; PMID 11153270; doi:10.1016/S0083-6729(01)61010-0
- U. Deppenmeier: The unique biochemistry of methanogenesis. In: Prog Nucleic Acid Res Mol Biol., 2002, 71, S. 223–283; PMID 12102556; doi:10.1016/S0079-6603(02)71045-3
- U. Deppenmeier: Redox-driven proton translocation in methanogenic Archaea. In: Cell Mol Life Sci., 2002, 59(9), 2002, 1513–1533; PMID 12440773; doi:10.1007/s00018-002-8526-3
- Michael T. Madigan, John M. Martinko, Thomas D. Brock: Mikrobiologie. 11. überarb. Auflage. Pearson Studium, 2006, ISBN 3-8273-7187-2, S. 640–643
- Georg Fuchs (Hrsg.): Allgemeine Mikrobiologie. 8. völlig überarb. u. erw. Auflage. Thieme, 2007, ISBN 3-13-444608-1, S. 395–399
Einzelnachweise
- Dieser Stoff wurde in Bezug auf seine Gefährlichkeit entweder noch nicht eingestuft oder eine verlässliche und zitierfähige Quelle hierzu wurde noch nicht gefunden.
- P. Cheeseman et al.: Isolation and properties of a fluorescent compound, factor 420, from Methanobacterium strain M.o.H. In: J Bacteriol., 1972, 112(1), S. 527–531 (englisch); PMID 5079072; jb.asm.org (PDF)
- LD. Eirich et al.: Proposed structure for coenzyme F420 from Methanobacterium. In: Biochemistry, 1978, 17(22), S. 4583–4593; PMID 728375
- G. Bashiri, C. J. Squire, N. J. Moreland, E. N. Baker: Crystal structures of F420-dependent glucose-6-phosphate dehydrogenase FGD1 involved in the activation of the anti-tuberculosis drug candidate PA-824 reveal the basis of coenzyme and substrate binding. In: The Journal of biological chemistry. Band 283, Nummer 25, Juni 2008, S. 17531–17541. doi:10.1074/jbc.M801854200. PMID 18434308.
- AA. DiMarco et al.: Unusual coenzymes of methanogenesis. In: Annu Rev Biochem., 1990, 59, S. 355–394; PMID 2115763
- F. Jacobson, C. Walsh: Properties of 7,8-didemethyl-8-hydroxy-5-deazaflavins relevant to redox coenzyme function in methanogen metabolism. In: Biochemistry, 1984, 23, S. 979–988
- JRD. McCormick, George O. Morton: Identity of cosynthetic factor I of Streptomyces aureofaciens and fragment FO from coenzyme F420 of Methanobacterium species. In: J. Am. Chem. Soc., 1982, 104(14), S. 4014–4015; doi:10.1021/ja00378a044
- AP. Eker et al.: DNA photoreactivating enzyme from the cyanobacterium Anacystis nidulans. In: J Biol Chem., 1990, 265(14), S. 8009–8015 (englisch); PMID 2110564; jbc.org (PDF)
- AP. Eker et al.: Photo-reactivating enzyme from the green alga Scenedesmus acutus. Evidence for two different chromophores. In: Biochemistry, 1998, 27(5), S. 1758–1765; doi:10.1021/bi00405a056
- DE. Graham, RH. White: Elucidation of methanogenic coenzyme biosyntheses: from spectroscopy to genomics. In: Nat Prod Rep., 2002, 19(2), S. 133–147; PMID 12013276
- EF. Johnson, B. Mukhopadhyay: A new type of sulfite reductase, a novel coenzyme F420-dependent enzyme, from the methanarchaeon Methanocaldococcus jannaschii. In: J Biol Chem., 2005, 280(46), S. 38776–38786 (englisch); PMID 16048999; jbc.org (PDF)
- H. Seedorf et al.: F420H2 oxidase (FprA) from Methanobrevibacter arboriphilus, a coenzyme F420-dependent enzyme involved in O2 detoxification. In: Arch Microbiol., 2004, 182(2–3), S. 126–137; PMID 15340796
- U. Deppenmeier: The membrane-bound electron transport system of Methanosarcina species. In: J Bioenerg Biomembr., 2004, 36(1), S. 55–64; PMID 15168610
- Daniels, L, Bakhiet, N, Harmon, K: Widespread distribution of a 5-deazaflavin cofactor in Actinomyces and related bacteria. In: Syst. Appl. Microbiol. Vol. 6, no. 1, pp. 12–17. 1985.
- E. Purwantini, B. Mukhopadhyay: Conversion of NO2 to NO by reduced coenzyme F420 protects mycobacteria from nitrosative damage. In: Proceedings of the National Academy of Sciences Band 106, Nummer 15, April 2009, S. 6333–6338. doi:10.1073/pnas.0812883106. PMID 19325122. PMC 266939 (freier Volltext).
- U. H. Manjunatha, H. Boshoff u. a.: Identification of a nitroimidazo-oxazine-specific protein involved in PA-824 resistance in Mycobacterium tuberculosis. In: Proceedings of the National Academy of Sciences Band 103, Nummer 2, Januar 2006, S. 431–436. doi:10.1073/pnas.0508392103. PMID 16387854.
- BW. te Brömmelstroet et al.: Purification and properties of 5,10-methylenetetrahydromethanopterin dehydrogenase and 5,10-methylenetetrahydromethanopterin reductase, two coenzyme F420-dependent enzymes, from Methanosarcina barkeri. In: Biochim Biophys Acta., 1991, 1079 (3), S. 293–302; PMID 1911853
- CH. Hagemeier et al.: Coenzyme F420-dependent methylenetetrahydromethanopterin dehydrogenase (Mtd) from Methanopyrus kandleri: a methanogenic enzyme with an unusual quaternary structure. In: J Mol Biol., 2003, 332(5), S. 1047–1057; PMID 14499608
- JA. Fox et al.: 8-Hydroxy-5-deazaflavin-reducing hydrogenase from Methanobacterium thermoautotrophicum: 1. Purification and characterization. In: Biochemistry, 1987, 26(14), S. 4219–4227; PMID 3663585
- U. Deppenmeier: The Unique Biochemistry of Methanogenesis. In: PROG Nucleic Acid Res Mol Biol., 2002, 71, S. 223–283; PMID 12102556
- S. Yamazaki, L. Tsai: Purification and properties of 8-hydroxy-5-deazaflavin-dependent NADP+ reductase from Methanococcus vannielii. In: J Biol Chem., 1980, 255 (13), S. 6462–6465 (englisch); PMID 7391030; jbc.org (PDF)
- H. Berk, RK. Thauer: Function of coenzyme F420-dependent NADP reductase in methanogenic archaea containing an NADP-dependent alcohol dehydrogenase. In: Arch Microbiol., 1997, 168, 5, S. 396–402; PMID 9325428
- AP. Shuber et al.: Cloning, expression, and nucleotide sequence of the formate dehydrogenase genes from Methanobacterium formicicum. In: J Biol Chem., (1986, 261, 28), S. 12942–12947 (englisch); PMID 3531194; jbc.org (PDF)
- SW. Aufhammer et al.: Coenzyme binding in F420-dependent secondary alcohol dehydrogenase, a member of the bacterial luciferase family. In: Structure, 2004, 12 (3), S. 361–370; PMID 15016352
- J. D. Selengut, D. H. Haft: Unexpected abundance of coenzyme F(420)-dependent enzymes in Mycobacterium tuberculosis and other actinobacteria. In: Journal of bacteriology, Band 192, Nummer 21, November 2010, S. 5788–5798; doi:10.1128/JB.00425-10, PMID 20675471, PMC 2953692 (freier Volltext).