Dependoparvovirus
Dependoparvovirus (ehemals: Dependovirus, Adeno-associated virus group, AAV) ist ein Genus der Parvoviridae, einer Familie von DNA-Viren, Unterfamilie Parvovirinae. Bekannteste Vertreter der Dependoparvoviren sind das Adeno-assoziierte Virus (AAV) A und B. Die Replikation der Dependoparvoviren ist von einer Koinfektion mit einem Helfervirus abhängig, woher auch der Name stammt. Als Helferviren werden Adenoviren oder Herpesviren verwendet.[2][3]
| Dependoparvovirus | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
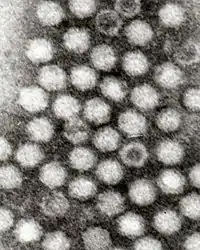
Adeno-assoziierte Viren | ||||||||||||||||||
| Systematik | ||||||||||||||||||
| ||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||
| ||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||
| Dependoparvovirus | ||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||
| AAV | ||||||||||||||||||
| Links | ||||||||||||||||||
|
Eigenschaften
Virion
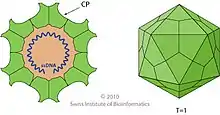
Die Virionen (Virusteilchen) der Dependoparvoviren besitzen ein unbehülltes ikosaedrisches Kapsid von etwa 22 nm Durchmesser.[4] Das Kapsid ist aus den drei Kapsidproteinen VP1–3 aufgebaut und besteht aus 60 Proteinen mit einer Triangulationszahl von eins. Jedes Kapsid besitzt fünf VP1-Proteine, fünf VP2-Proteine und 50 VP3-Proteine.[5] Darin befindet sich die einzelsträngige virale DNA.
Genom
Das DNA-Genom ist einzelsträngig von 4,7 Kilobasen (Kb) Länge mit unterschiedlicher Polarität und besitzt zwei offene Leseraster. Am 3'-Ende befindet sich das Gen cap für die Kapsidproteine, welches durch alternatives Spleißen die Proteine VP1 oder VP2 und VP3 ergibt. Das zweite Gen rep codiert für die replikationsrelevanten Proteine. Durch alternatives Spleißen entstehen entweder Rep78, Rep68, Rep52 oder Rep40, wobei die Nummerierung nach der elektrophoretischen Mobilität erfolgt.[4] Aus dem Genom werden sechs mRNA gebildet (4,2 Kb, 3,9 Kb, 3,6 Kb, 3,3 Kb, 2,6 Kb und 2,3 Kb Länge), die alle einen Poly-A-Schwanz tragen.[6]
Die an den Enden des Genoms befindlichen Inverted Repeats von etwa 145 Basen bilden aufgrund der palindromischen DNA-Sequenz der ersten 125 Basen eine T-förmige Sekundärstruktur.[6] Das inverted repeat dient sowohl als Ausgangspunkt der Replikation als auch für die Insertion der viralen DNA in die DNA des Wirtes auf Chromosom 19.[6] Die komplementären Bereiche des inverted repeats besitzen eine freie Hydroxygruppe am 3'-Ende für die Replikation. Das 3'-Ende wird als Primer für die Synthese des leading strand verwendet, anschließend werden doppelsträngige Übergangsformen gebildet.[4][5]
Dependoparvoviren sind für sich replikationsdefizient, d. h., sie benötigen ein Helfervirus.[7] Die zur Replikation notwendigen Proteine der Adenoviren wurden identifiziert und werden zur Herstellung von dependoviralen Vektoren verwendet.[8] Weiterhin kann eine Replikation der Dependoparvoviren auch durch UV-Strahlung, Cycloheximid, Aphidicolin, Topoisomeraseinhibitoren, Hydroxyurea und verschiedene chemische Karzinogene ausgelöst werden.[9][10][11][6]
Die Insertion der viralen DNA erfolgt auf Chromosom 19 bevorzugt in Troponin-Gene. Die enthaltenen Gene werden daher verstärkt in Muskelzellen abgelesen.[6] Durch die Insertion und die genetische Repression entsteht die Viruslatenz und die persistente Infektion. Deshalb werden Dependoparvoviren als virale Vektoren in der Gentherapie verwendet, wo eine längerfristige Genexpression der DNA des rekombinanten Proteins im Vektor ohne eine Produktion viraler Proteine erwünscht ist. Daneben können die Genome mancher AAV auch als Episom vorliegen.[6]
Proteom
Das Proteom der Dependoparvoviren umfasst unter anderem die drei im Virion vorkommenden strukturellen Proteine VP1–3 und die nicht strukturellen Proteine und Helicasen Rep78, Rep68, Rep52 oder Rep40.[6] Rep78 ist eine Helicase und ein Repressorprotein am rep bindig element (RBE) im p5-Promotor, wodurch die Herstellung von Rep78 und Rep68 in Abwesenheit eines Helfervirus weitestgehend eingestellt wird und stattdessen die Insertion erfolgt.[6] Rep52 ist ein Repressor am p19-Promotor, welcher die Genexpression von Rep52 und Rep40 kontrolliert.[6]
Replikationszyklus
Nach der Adsorption der Virionen an die Zellmembran erfolgt die Einstülpung per Endocytose. Während der Reifung des Endosoms zum Lysosom erfolgt die Penetration der Endosomenmembran und die Freisetzung des Genoms ins Zytosol. Das virale Genom wird in den Zellkern importiert, woraufhin die Genexpression der rep-Gene erfolgt und die Replikation des viralen Genoms über eine doppelsträngige DNA-Zwischenstufe eingeleitet wird. Durch Genexpression der cap-Gene werden die Komponenten des Virions vervollständigt. Danach erfolgt die Bindung der einzelsträngigen DNA positiver oder negativer Polarität an die Kapsidproteine, wodurch sich die Virionen zusammenfügen. Dependoparvoviren verlassen die Zelle nach deren Zerstörung durch die lytischen Helferviren.
Systematik
Zum Genus Dependoparvovirus gehören:
- Spezies Adeno-associated dependoparvovirus A (AAV-A)
- Adenoassoziiertes Virus 1 bis 4 (AAV-1 bis AAV-4)
- Spezies Adeno-associated dependoparvovirus B (AAV-B)
- Adenoassoziiertes Virus 5 (AAV-5)
- Spezies Anseriform dependoparvovirus 1
- Spezies Avian dependoparvovirus 1
- Spezies Chiropteran dependoparvovirus 1
- Spezies Pinniped dependoparvovirus 1
- Spezies Squamate dependoparvovirus 1
- Spezies Squamate dependoparvovirus 2
Wirtsspektrum
Dependoparvoviren kommen in verschiedenen Wirbeltieren vor. Sie sind dabei vor allem von der Anwesenheit eines Helfervirus abhängig. Es gibt keine mit Dependoparvoviren assoziierten Infektionskrankheiten, wobei Antikörper bei einer Infektion gebildet werden.[12] Mehr als 90 % der Erwachsenen sind AAV-seropositiv, d. h., sie waren oder sind mit AAV infiziert.[6]
Gentherapie
Verschiedene Dependoparvoviren integrieren ihre virale DNA in das Genom ihres Wirts, bevorzugt in Chromosom 19, was die Wahrscheinlichkeit verringert, in einen wichtigen Bereich einzudringen, der die normale Genfunktion stören oder das Krebsrisiko erhöhen könnte. Daher werden Dependoparvoviren wie das AAV in der Gentherapie als viraler Vektor eingesetzt,[13] Als virale Vektoren verwendete Dependoparvoviren besitzen eine relativ geringe DNA-Verpackungskapazität.[14] Die Adeno-assoziierten viralen Vektoren haben eine maximale DNA-Länge von acht Kilobasen (einzelsträngig), die noch in Virionen verpackt werden können.[15] Bei der Verwendung selbst-komplementärer DNA sinkt die maximale Länge eines Transgens auf fünf Kilobasen, bei Längen darüber sinkt der Virustiter.[16] Derzeit wird daran gearbeitet, die Menge an Informationen zu erhöhen, die dieser Vektor liefern kann. Dies kann durch die ITRs (englisch inverted terminal repeats) erreicht werden, die sich sowohl am 5'- als auch am 3'-Ende des Genoms befinden. Da die ITRs dieselbe Sequenz haben, bleiben nach ihrer Entfernung komplementäre Stränge frei. Die komplementären Stränge können rekombinieren und zwei 5-kb-insertierte Fragmente miteinander verbinden.[14]
Wie bei allen viralen Vektoren entstehen im Rahmen einer angeborenen und einer adaptiven Vektorimmunität Antikörper gegen die viralen Proteine (in diesem Fall gegen Kapsidproteine). Daher kann man zur Vermeidung eines vorzeitigen Abbaus des Vektors oder überschießender Immunreaktionen einen Serotyp pro Patienten nur einmal einsetzen,[17] so dass fortlaufend neue Serotypen entwickelt werden.[18][19][20] Jedoch wurden Insertionen von AAV-Vektoren auch in transkriptionsaktiven Bereichen des Genoms außerhalb der Troponingene gefunden, was zur Entstehung von Tumoren beitragen kann.[21][22]
Weblinks
- Dependoparvovirus. In: ViralZone. Abgerufen am 27. Dezember 2013.
- MeSH Dependoparvovirus
- Neil Savage: Gentherapie: Bessere Verpackung für DNA-Pakete. In: Spektrum. 07/2019, S. 20–23.
Einzelnachweise
- ICTV: ICTV Taxonomy history: Primate erythroparvovirus 1, EC 51, Berlin, Germany, July 2019; Email ratification March 2020 (MSL #35)
- M. W. Myers, C. A. Laughlin, F. T. Jay, B. J. Carter: Adenovirus helper function for growth of adeno-associated virus: effect of temperature-sensitive mutations in adenovirus early gene region 2. In: Journal of Virology. Band 35, Nr. 1, 1. Juli 1980, S. 65–75, PMID 6251278, PMC 288783 (freier Volltext).
- H. Handa, B. J. Carter: Adeno-associated virus DNA replication complexes in herpes simplex virus or adenovirus-infected cells. In: The Journal of Biological Chemistry. Band 254, Nr. 14, 25. Juli 1979, S. 6603–6610, PMID 221504.
- M. Gonçalves: Adeno-associated virus: from defective virus to effective vector. In: Virology Journal. Band 2, Nr. 1, 2005, S. 43–60, doi:10.1186/1743-422X-2-43, PMID 15877812, PMC 1131931 (freier Volltext) – (virologyj.com).
- ICTVdB Management: ICTVdB – The Universal Virus Database, version 4: Dependovirus. Hrsg.: C. Büchen-Osmond. Columbia University, New York 2006 (nih.gov [abgerufen am 4. Mai 2009]).
- D. M. Knipe, Peter M. Howley (Hrsg.): Fields Virology. 5. Auflage. Philadelphia 2007, ISBN 978-0-7817-6060-7.
- T. B. Lentz, S. J. Gray, R. J. Samulski: Viral vectors for gene delivery to the central nervous system. In: Neurobiology of disease. Band 48, Nummer 2, November 2012, S. 179–188, ISSN 1095-953X. doi:10.1016/j.nbd.2011.09.014. PMID 22001604. PMC 3293995 (freier Volltext).
- T. Matsushita, S. Elliger, C. Elliger u. a.: Adeno-associated virus vectors can be efficiently produced without helper virus. In: Gene Therapy. Band 5, Nr. 7, Juli 1998, S. 938–945, doi:10.1038/sj.gt.3300680, PMID 9813665.
- A. O. Yalkinoglu, R. Heilbronn, A. Bürkle, J. R. Schlehofer, H. zur Hausen: DNA amplification of adeno-associated virus as a response to cellular genotoxic stress. In: Cancer Research. Band 48, Nr. 11, 1. Juni 1988, S. 3123–3129, PMID 2835153.
- B. Yakobson, T. Koch, E. Winocour: Replication of adeno-associated virus in synchronized cells without the addition of a helper virus. In: Journal of Virology. Band 61, Nr. 4, 1. April 1987, S. 972–981, PMID 3029431, PMC 254052 (freier Volltext).
- B. Yakobson, T. A. Hrynko, M. J. Peak, E. Winocour: Replication of adeno-associated virus in cells irradiated with UV light at 254 nm. In: Journal of Virology. Band 63, Nr. 3, 1. März 1989, S. 1023–1030, PMID 2536816, PMC 247794 (freier Volltext).
- M. Agbandje, C. R. Parrish, M. G. Rossmann: The recognition of parvovirus capsids by Antibodies. In: Seminars in Virology. Band 6, 1995, S. 219–231, doi:10.1006/smvy.1995.0027 (ohiolink.edu [PDF]).
- K. Excoffon u. a.: Directed evolution of adeno-associated virus to an infectious respiratory virus. In: Proceedings on the National Academy of Science. Band 106, Nr. 10, 2009, S. 3865–3870, doi:10.1073/pnas.0813365106, PMID 19237554, PMC 2646629 (freier Volltext) – (cell.com).
- A. Ghosh, Y. Yue, Y. Lai, D. Duan: A Hybrid Vector System Expands Adeno-associated Viral Vector Packaging Capacity in a Transgene-Independent manner. In: Molecular Therapy. Band 16, Nr. 1, 2008, S. 124–130, doi:10.1038/sj.mt.6300322, PMID 17984978 (nature.com [PDF]).
- D. M. McCarty: Self-complementary AAV vectors; advances and applications. In: Mol Ther. Band 16, Nr. 10, 2008, S. 1648–1656. PMID 18682697.
- R. M. Kotin: Large-scale recombinant adeno-associated virus production. In: Hum Mol Genet. Band 20(R1), 2011, S. R2–R6. PMID 21531790; PMC 3095058 (freier Volltext).
- F. Mingozzi, K. A. High: Immune responses to AAV in clinical trials. In: Curr Gene Ther. Band 11, Nr. 4, 2011, S. 321–330. PMID 21557723.
- I. Kwon, D. V. Schaffer: Designer gene delivery vectors: Molecular engineering and evolution of adeno-associated viral vectors for enhanced gene transfer. In: Pharmaceut Res. Band 25, 2008, S. 489–499.
- E. Ayuso, F. Mingozzi, F. Bosch: Production, purification and characterization of adeno-associated vectors. In: Curr Gene Ther. Band 10, Nr. 6, 2010, S. 423–436. PMID 21054248.
- J. Wang, S. M. Faust, J. E. Rabinowitz: The next step in gene delivery: molecular engineering of adeno-associated virus serotypes. In: Journal of Molecular and Cellular Cardiology. Band 50, Nr. 5, 2011, S. 793–802. PMID 21029739.
- D. R. Deyle, D. W. Russell: Adeno-associated virus vector integration. In: Curr Opin Mol Ther. Band 11, Nr. 4, 2009, S. 442–447. PMID 19649989; PMC 2929125 (freier Volltext).
- A. Donsante, D. G. Miller, Y. Li, C. Vogler, E. M. Brunt, D. W. Russell, M. S. Sands: AAV vector integration sites in mouse hepatocellular carcinoma. In: Science. Band 317, Nr. 5837, 2007, S. 477. PMID 17656716.