Clostridium paradoxum
Clostridium paradoxum[1] ist ein moderat thermophiles alkaliphiles Bakterium, das dem paraphyletischen Taxon der Clostridien zugeordnet wird. Es erhielt die Bezeichnung paradoxum (von altgriechisch παράδοξον, unerwartet, widersprüchlich), da bei ihm begeißelte Endosporen vorkommen.
| Clostridium paradoxum | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
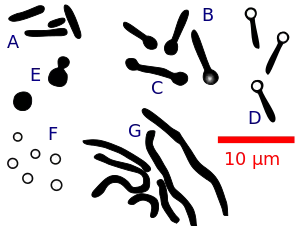
Abb. 1. Morphologie von Clostridium paradoxum unter verschiedenen Wachstumsbedingungen. Mehr im Text. | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Clostridium paradoxum | ||||||||||||
| Li, Mandelco & Wiegel, 1993 |
Erscheinungsbild
Clostridium paradoxum kann 2–6 peritriche Geißeln und Endosporen bilden. In statischer Kultur bei der für das Bakterium optimalen Temperatur (56 °C) verändert es beim Durchlaufen der Wachstumsphasen seine Zellform (siehe Abb. 1).
Exponentiell in Flüssigkultur (pH 10,1) wachsende Zellen sind zunächst 2–4,5 µm lang und stäbchenförmig (Abb. 1. A). In der späten exponentiellen Phase setzt die Sporenbildung ein (B und C). Sie kommt in der stationären Phase zum Ende (D). Dann treten auch teilweise begeißelte L-Formen auf (E). Danach werden die Sporen freigesetzt (F).
Am Rande des pH-Maximum pH 11 wachsen die Bakterien als Filamente (G) mit bis zu 50 µm Länge. Der Effekt tritt auch auf, wenn die Zellen am Temperatur-Maximum von ca. 60–63 °C kultiviert werden.
Wachstum und Stoffwechsel
Clostridium paradoxum ist obligat anaerob und wächst in Reinkultur nur auf sauerstofffreien Medien. Es kann Sulfat nicht reduzieren.
Das Bakterium nutzt chemotroph Hefeextrakt und Trypton.
- Nutzbare Substrate (unter Zugabe von 0,3 % Hefeextrakt): Glucose, Fructose, Saccharose, Maltose und Pyruvat.
- Nicht nutzbar u. a.: Glycerin, Milchsäure, Ameisensäure, Stärke, Cellulose, Ethanol, Methanol, Aminosäuren.
Abbauprodukte bei pH-Werten zwischen 7,5 und 10 sind hauptsächlich Acetat, CO2 und H2. Das Bakterium gewinnt Energie durch Gärung und nicht durch chemiosmotisch betriebene ATP-Synthasen.
Der Organismus wächst bei pH-Werten zwischen 7,0 und 11,1. Das Optimum liegt bei pH 10,1. Bei diesem pH-Wert wächst das Bakterium bei Temperaturen zwischen 30 °C und 63 °C, das Optimum liegt bei 56 °C. Unter optimalen Bedingungen wächst das Bakterium mit einer Generationszeit von 16 Minuten.
Die molekularen Grundlagen für die Toleranz gegenüber alkalischen Medien von Clostridium paradoxum werden behandelt in
Vorkommen
Clostridium paradoxum und physiologisch ähnliche Stämme wurden in Belebtschlamm- und Faulschlamm-Proben nachgewiesen, in verschiedenen US-amerikanischen Städten aus Kläranlagen entnommen wurden. In diesem Biotop treten zwar für die Organismen regelmäßig anaerobe Bedingungen auf. Es wurden dort aber nie die für Clostridium paradoxum optimalen Temperaturen und pH-Werte gemessen.
Chemotaxonomische Merkmale und externe Taxonomie
Clostridium paradoxum hat keine Lipopolysaccharide und wird deshalb den grampositiven Organismen zugeordnet, auch wenn es bei der Gram-Färbung negativ reagiert. Es weist gegenüber den meisten anderen Clostridien weder auffällige Antibiotika-Empfindlichkeiten noch Resistenzen auf. Es konnten keine Häme nachgewiesen werden.
Der GC-Gehalt der genomischen DNS beträgt ca. 30 mol%.
Die taxonomische Zuordnung basiert der 16S-RNA-Sequenz. Bei der Erstbeschreibung wurde der vorläufige Gattungsname Clostridium gewählt, da die Autoren mit einer baldigen umfassenden taxonomischen Restrukturierung der Clostridien rechneten. Sie ist (2015) noch nicht abgeschlossen.
Es wurden bei den Clostridien 19 genetisch klar abgrenzbare Cluster gefunden, denen die einzelnen Gattungen zugeordnet werden können. Clostridium paradoxum (im Cluster XI) gehört, obwohl es aktuell der Gattung Clostridium innerhalb der Clostridiaceae zugeordnet wird, danach zu der monophyletischen Familie der Peptostreptococcaceae[2] und ist eng mit dem Darmbakterium Peptrostreptococcus anaerobius verwandt[3]:
| Clostridiales |
| ||||||||||||||||||||||||
Typ-Stamm ist JW-YL-7 (DSM 7308).[4]
Literatur
- Rainey FA, Ward-Rainey NL, Janssen PH, Hippe H, Stackebrandt E: Clostridium paradoxum DSM 7308T contains multiple 16S rRNA genes with heterogeneous intervening sequences.. In: Microbiology. 142, Nr. 8, 1996, S. 2087–95. doi:10.1099/13500872-142-8-2087. PMID 8760921.
- Cook, Gregory M., et al.: The Intracellular pH of Clostridium paradoxum, an Anaerobic, Alkaliphilic, and Thermophilic Bacterium. Applied and environmental microbiology 62.12 (1996): 4576–4579.
- S. A. Ferguson, S. Keis, G. M. Cook: Biochemical and Molecular Characterization of a Na+-Translocating F1Fo-ATPase from the Thermoalkaliphilic Bacterium Clostridium paradoxum. In: Journal of Bacteriology. 188, Nr. 14, 2006, ISSN 0021-9193, S. 5045–5054. doi:10.1128/JB.00128-06.
Referenzen
- Li, Youhong, Mandelco, Linda, Wiegel, Juergen: Isolation and Characterization of a Moderately Thermophilic Anaerobic Alkaliphile, Clostridium paradoxum sp. nov. (Seite nicht mehr abrufbar, Suche in Webarchiven) Info: Der Link wurde automatisch als defekt markiert. Bitte prüfe den Link gemäß Anleitung und entferne dann diesen Hinweis. 1993 International Journal of Systematic and Evolutionary Microbiology, 43,3 S. 450–460. doi:10.1099/00207713-43-3-450
- Ludwig, Wolfgang, Karl-Heinz Schleifer, and William B. Whitman. Revised road map to the phylumFirmicutes. Bergey’s Manual® of Systematic Bacteriology. Springer New York, 2009. 1-13. Archivlink (Memento des Originals vom 1. Juli 2015 im Internet Archive) Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis. (S. 10)
- COLLINS M, LAWSON P, WILLEMS A, CORDOBA J, FERNANDEZ-GARAYZABAL J, GARCIA P, CAI J, HIPPE H, FARROW: The Phylogeny of the Genus Clostridium: Proposal of Five New Genera and Eleven New Species Combinations. Int J Syst Evol Microbiol 44(4):812-826 doi:10.1099/00207713-44-4-812
- Y. Li, L. Mandelco, J. Wiegel: Isolation and Characterization of a Moderately Thermophilic Anaerobic Alkaliphile, Clostridium paradoxum sp. nov.. In: International Journal of Systematic Bacteriology. 43, Nr. 3, 1993, ISSN 0020-7713, S. 450–460. doi:10.1099/00207713-43-3-450.