TIM-Fass
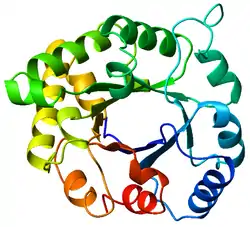
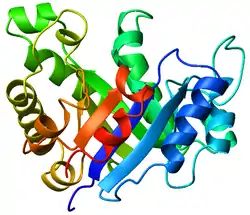
Das TIM-Fass (engl. TIM barrel, auch als α/β-Fass[1] oder (β/α)8-Fass[2] bekannt) ist in der Biochemie und Molekularbiologie eine konservierte Proteinfaltung, die aus acht α-Helices und acht parallelen β-Strängen besteht, die sich entlang des Peptidrückgrats abwechseln. Die Struktur ist nach Triosephosphatisomerase, einem konservierten Stoffwechselenzym, benannt. TIM-Fässer sind eine der häufigsten Proteinfaltungen. Eines der Merkmale unter den Mitgliedern dieser Klasse von Proteinen ist, dass obwohl sie alle die gleiche Faltung in ihrer Tertiärstruktur aufweisen, besteht zwischen ihnen nur eine sehr geringe Sequenzähnlichkeit. Mindestens 15 verschiedene Enzymfamilien verwenden das TIM-Fass, um die geeignete Geometrie des aktiven Zentrums zu generieren, und zwar immer am C-terminalen Ende der acht parallelen β-Stränge des Fasses.
Struktur und Zusammensetzung
TIM-Fässer werden als α/β-Proteinfaltungen angesehen, da sie ein alternierendes Muster von α-Helices und β-Strängen in einer einzelnen Domäne enthalten. In einem TIM-Fass bilden die Helices und Stränge (normalerweise jeweils 8) eine Art „Zylinderspule“, die sich in Form eines „Donuts“, der topologisch als Toroid bezeichnet wird, krümmt und so sich selbst schließt. Die parallelen β-Stränge bilden die Innenwand des „Donuts“, während die α-Helices die Außenwand des „Donuts“ bilden. Jeder β-Strang ist durch eine lange rechtsgängige Schleife mit dem nächsten benachbarten Strang verbunden, wobei die Schleife eine der α-Helices enthält. Man kann sich auch vorstellen, dass das TIM-Fass aus 8 überlappenden rechtshändigen β-α-β-Supersekundärstrukturen besteht.[3]
Der Kern des Proteins ist eng gepackt, hauptsächlich mit sperrigen hydrophoben Aminosäureresten, obwohl ein paar Glycine benötigt werden, damit das stark eingeschränkte Zentrum der ungefähr 8 Wiederholungen zusammenpassen kann. Die Wechselwirkungen zwischen den Strängen und Helices werden auch von der Hydrophobizität dominiert und die verzweigten aliphatischen Reste Valin, Leucin und Isoleucin machen etwa 40 % der Gesamtreste in den β-Strängen aus.[3]
Bei vielen TIM-Proteinen sind die katalytische Stelle und die β-Fasskomponenten starrer als die helikalen Teile, deren Positionen bei bestimmten Proteinen häufig variieren können.[4] Ein ideales 4-fach symmetrisches TIM-Fass-Enzym wurde von der Rosetta@home-Gruppe mithilfe von verschiedenen Berechnungsmethoden entwickelt.[5]
Schleifen-Regionen
Von den ungefähr 200 Resten, die erforderlich sind, um ein TIM-Fass vollständig zu bilden, werden ungefähr 160 als strukturell äquivalent zwischen verschiedenen Proteinen angesehen, die diese Faltung teilen. Die verbleibenden Reste befinden sich auf den Schleifenregionen, die die Helices und Stränge verbinden.
Die Schleifen am C-terminalen Ende der Stränge neigen dazu, das aktive Zentrum zu enthalten, was ein Grund dafür ist, dass diese Faltung häufig stattfindet. Die zur Aufrechterhaltung der Struktur erforderlichen Reste und die Reste, welche die enzymatische Katalyse bewirken, gehören größtenteils zu verschiedenen Untergruppen.[6] Die Schleifen können in der Tat so lang sein, dass sie andere Proteindomänen enthalten. Kürzlich wurde gezeigt, dass katalytische Schleifen zwischen verschiedenen TIM-Fass-Enzymen als halbautonome Einheiten funktioneller Gruppen ausgetauscht werden können.[7]
Einzelnachweise
- Donald Voet, Judith G. Voet: Biochemistry. 4. Auflage. John Wiley & Sons, 2011, ISBN 978-0-470-91745-9, 8. Three-Dimensional Structures of Proteins, S. 252.
- D. Mehta, T. Satyanarayana: Bacterial and Archaeal α-Amylases: Diversity and Amelioration of the Desirable Characteristics for Industrial Applications. In: Frontiers in Microbiology. Band 7, 2016, S. 9, doi:10.3389/fmicb.2016.01129 (englisch).
- C. Branden, J. Tooze: Introduction to Protein Structure. 2. Auflage. Garland Publishing, New York 1999, ISBN 978-0-8153-2305-1, S. 47–50.
- S. P. Tiwari, N. Reuter: Similarity in Shape Dictates Signature Intrinsic Dynamics Despite No Functional Conservation in TIM Barrel Enzymes. In: PLoS Computational Biology. Band 12, Nummer 3, März 2016, S. e1004834, doi:10.1371/journal.pcbi.1004834, PMID 27015412, PMC 4807811 (freier Volltext).
- P. S. Huang, K. Feldmeier, F. Parmeggiani, D. A. Velasco, B. Höcker, D. Baker: De novo design of a four-fold symmetric TIM-barrel protein with atomic-level accuracy. In: Nature chemical biology. Band 12, Nummer 1, Januar 2016, S. 29–34, doi:10.1038/nchembio.1966, PMID 26595462, PMC 4684731 (freier Volltext).
- A. Ochoa-Leyva, X. Soberón, F. Sánchez, M. Argüello, G. Montero-Morán, G. Saab-Rincón: Protein design through systematic catalytic loop exchange in the (beta/alpha)8 fold. In: Journal of molecular biology. Band 387, Nummer 4, April 2009, S. 949–964, doi:10.1016/j.jmb.2009.02.022, PMID 19233201.
- A. Ochoa-Leyva, F. Barona-Gómez, G. Saab-Rincón, K. Verdel-Aranda, F. Sánchez, X. Soberón: Exploring the Structure-Function Loop Adaptability of a (β/α)(8)-Barrel Enzyme through Loop Swapping and Hinge Variability. In: Journal of molecular biology. Band 411, Nummer 1, August 2011, S. 143–157, doi:10.1016/j.jmb.2011.05.027, PMID 21635898.