PML-Körper
PML-Körper (PML-Body, PML-Körperchen, PML oncogenic domain (POD), nuclear domain 10 (ND 10) oder Kremer (Kr) Körper) sind kugelförmige Gebilde mit einem Durchmesser von 0,1–1,0 µm, die im Zellkern in den meisten Zelllinien und vielen Geweben vorkommen. In Kernen von Zellkultur-Wirbeltierzellen finden sich durchschnittlich 5 bis 30 PML-Kernkörperchen mit einer Größe von ~0,2 bis 1 µm.
Der Name PML-Körper leitet sich von der akuten Promyelozyten-Leukämie her, da sie bei dieser Erkrankung, aber auch bei verschiedenen Virusinfektionen zerstört werden. PML-Körper wurden in den frühen 1960er Jahren entdeckt. Die Identifizierung von PML, einem Gen, das an einer onkogenen chromosomalen Translokation beteiligt ist, als Schlüsselorganisator dieser Objekte weckte weiteres Interesse an ihnen. Die verschiedenen Ebenen der Regulation durch eine spezifische posttranslationale Modifikation, der SUMOylierung, haben mehrere ungelöste Probleme aufgeworfen. Funktionell können PML-Körper Partnerproteine binden, modifizieren oder abbauen.
Mehrere Untertypen dieser Körper wurden auf morphologischer Basis definiert. Sie haben alle eine elektronendichte Hülle und enthalten einen inneren Kern. PML-Körper rückten mit der Beobachtung in den Vordergrund, dass das onkogene PML/RARA-Protein sie auf eine behandlungsreversible Weise zerstört. PML-Körper werden durch zellulären Stress reguliert: Virusinfektion, DNA-Schäden, Transformation und oxidativer Stress. Darüber hinaus wird die Transkription von PML und mehreren Genen, die für Partnerproteine kodieren, durch Interferone dramatisch verbessert.[1]
Geschichte
Mit Hilfe von Elektronenmikroskopie und Einsatz von Autoantikörpern wurden die ersten Forschungen in diesem Feld getätigt. In den frühen 1960er Jahren zeigten Forscher das Vorhandensein von dichten kugelförmigen Objekten mittels Elektronenmikroskopie. Zwei Klassen von Körpern wurden damals beschrieben: Leere (fibrilläre) und körnige Körper. Die körnigen Körper enthalten im Inneren ein mikrogranulares Material, für das Ribonukleoproteine vorgeschlagen wurden. PML-Nukleärkörper wurden später durch die Immunfluoreszenz-Methode mit Autoimmunseren von Patienten mit Primär biliäre Zirrhose beobachtet. Diese ermöglichten die Identifizierung des ersten assoziierten Proteins, SP100, und eine erste Charakterisierung dieser Strukturen.
Die Identifizierung eines anti-nukleären Matrix-Antikörpers, der die gleichen Strukturen wie SP100 markierte, stellte die erste Verbindung zwischen diesen Körpern und der Kernmatrix her. Die Lokalisierung von PML-Proteinen auf die gleichen Stellen wie SP100 führte zu einem erneuten Interesse an diesen Bereichen. PML ist ein Protein, welches mit dem Retinsäure-Rezeptor α(RARA) im PML/RARA-Onkoprotein der akuten promyelozytären Leukämie (APL) fusioniert ist.
In den 1990er Jahren wurde beobachtet, dass in APL-Zellen PML/RARA diese Bereiche störte. Die führte in der wissenschaftlichen Gemeinschaft zu einer intensiveren Erforschung dieser Körper. Darüber hinaus wurden PML-Körper durch zwei verschiedene Anti-APL-Therapien, Retinsäure und Arsentrioxid, wiederhergestellt. Hierbei wurde später der PML/RARA-Abbau ausgelöst. Dadurch konnte man die erste auffällige Parallele zwischen dem Status der Körper und dem der Zelle identifizieren. Viele weitere Studien in den 2000er Jahren zeigten, dass sich PML-Körper unter Stressbedingungen veränderten, insbesondere bei Virusinfektionen, Hitzeschock und Exposition gegenüber Schwermetallen.
Beteiligte Proteine
PML-Körper rekrutieren eine ständig wachsende Anzahl von Partnerproteinen (heute im Bereich von 100), wobei eines der am besten untersuchten DAXX ist, ein starker Repressor der Transkription und Modulator der Apoptose. Entscheidend ist, dass PML der eigentliche Organisator dieser Körper ist. Unter diesen rekrutierten Proteinen verdient man eine besondere Erwähnung: Ein Ubiquitin-ähnliches Protein namens SUMO, da die PML-Konjugation durch SUMO eine entscheidende Rolle bei der Rekrutierung von Partnern spielt, von denen viele selbst sumoyliert sind.
PML
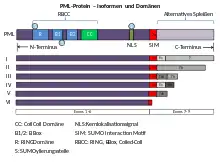
B: B-Box, CC: Coiled-coil Domäne, NLS: Kernlokalisierungssignal, S: Sumoylierungsstellen, SIM: SUMO-Interaktionsmotiv. Unten: Darstellung der sechs nukleären PML-Isoformen (I bis VI). Die Exons 1–6 werden von allen Isoformen geteilt, während ihre C-Termini sich durch das alternative Spleißen der Exons 7–9 individuell unterscheiden
Das PML-Protein ist ein Tumorsuppressorprotein aus der TRIM/RBCC-Proteinfamilie und der Organisator der PML-Kernkörper. Wie viele Proteine aus dieser Familie ist das PML-Protein eine Ubiquitin-Ligase. Diese kann subzelluläre Strukturen durch Autoassemblierung erzeugen. Die Transkription des PML-Gens wird durch Interferone α/β oder γ, aber auch durch das p53-Protein kontrolliert. Beide bewirken einen dramatischen Anstieg der Anzahl und Größe der PML-Körper.
PML verfügt über eine aminoterminalen RING-Fingerdomäne, die eine SUMO-Ligase direkt bindet, und eine Coiled-coil (Doppelwendel-Struktur), welche die Homodimerisierung vermittelt. Dieser Abschnitt des Proteins wird als RBCC bezeichnet (RING-Finger, B box and coiled coil-domain). Zudem besitzt sie einen als SIM (SUMO-Interaktionsmotiv) bezeichneten Proteinabschnitt.
Durch alternatives Spleißen werden mehrere Varianten (Isoformen) des PML-Proteins hergestellt. Diese haben unterschiedliche carboxyterminalen Domänen. Zusätzlich zum nukleären Lokalisierungssignal (NLS), welches in allen PML-Isoformen vorhanden ist, verfügt PML-I über ein nukleäres Exportsignal, das es ermöglicht, alle Isoformen durch Heterodimerbildung zwischen den Zellkern und Cytoplasma hin und her zu bewegen. Die am häufigsten vorkommende Isoform, PML-I, beherbergt auch eine Exonuklease-III-Domäne. In menschlichen Zellen werden alle sechs Isoformen exprimiert. Hiervon machen die PML-Isoformen I und II den größten Anteil aus.[2]
PML durchläuft mehrere kritische posttranslationale Modifikationen, insbesondere durch Phosphorylierung und Sumoylierung. Die PML-Sumoylierung ist insbesondere wichtig für den Aufbau von PML-Körperchen. Verschiedene Kinasen, die durch Schäden an der DNA oder durch Stress aktiviert werden, phosphorylieren PML. Sie regulieren dadurch möglicherweise die PML-Stabilität, die Biogenese von Nukleärkörpern und tragen zur DNA-Reparatur oder Apoptose-Kontrolle bei.
Die überwiegende Mehrheit des PML-Proteinpools in einer Zelle ist nicht in den PML-Körpern gebunden. In den meisten Zelllinien sind mehr als 90 % der PML-Proteine im Zellkern verteilt aufzufinden.[1]
| PML-Isoform | Lokalisierung in pml -/- Zellen |
|---|---|
| PML I | Nukleär bei Stress, Zytoplasma |
| PML II | Fibrilläre/nukleäre Hülle (Lamina?) |
| PML IV | Nukleär bei Stress |
| PMLV | Dicke Schale / Verankerung in der Matrix |
| PMLVII | Zytoplasmatische/frühe Endosomen |
Die Expression von PML-Isoformen in pml-/--Zellen offenbart jeweils eigene Lokalisationen. Dies deutet darauf hin, dass der Carboxy-Terminus Interaktionen mit spezifischen, aber noch unbekannten Partner steuert.[1]
SUMO
SUMO-Proteine sind eine Familie von kleinen Proteinen, die kovalent an andere Proteine in Zellen gebunden sind, um ihre Funktion zu verändern. Sumoylierung ist eine posttranslationale Modifikation, die an verschiedenen zellulären Prozessen beteiligt ist, wie z. B. Chromatinorganisation, Transkription, Signaltransduktion, Apoptose, Proteinstabilität und Reaktion auf Stress.[3]
SP100
Dieses Gen kodiert für eine subnukleäre Organelle und Hauptkomponente der PML-Körper. PML und SP100 werden kovalent durch den SUMO-1-Modifikator modifiziert. Das kodierte Protein bindet Heterochromatin-Proteine und soll eine Rolle bei der Tumorgenese, Immunität und Genregulation spielen. Für dieses Gen wurden alternativ gespleißten Varianten identifiziert, von denen eine ein hochmobiles Gruppenprotein kodiert.[4]
DAXX
DAXX (Death-associated protein 6) ist ein mit der Todesdomäne assoziiertes Protein. Es wurde erstmals durch seine zytoplasmatische Interaktion mit dem Rezeptor Fas entdeckt, welcher die Zell-Apoptose einleitet. Es wurde mit Heterochromatin und PML-Körpern in Verbindung gebracht und ist an vielen Kernprozessen beteiligt, einschließlich Transkription und Zellzyklusregulation.
p53
Der humane Tumorsuppressor p53 reguliert als Transkriptionsfaktor nach DNA-Schädigung die Expression von Genen, die an der Kontrolle des Zellzyklus, an der Induktion der Apoptose (des programmierten Zelltods) oder an der DNA-Reparatur beteiligt sind.
Struktur
Typen von PML-Körpern
Es wird davon ausgegangen, dass es mehrere Typen von PML-Körpern gibt. Tatsächlich aggregiert sich das PML-Protein als Reaktion auf eine Vielzahl von Belastungen in unterschiedlicher Form. Der am ausführlichsten untersuchte Faktor, der auf die PML-Verteilung Auswirkung hat, ist Arsentrioxid. Es sind auch mehrere durch DNA-Schäden aktivierte Kinasen als wichtig erkannt worden. Stressinduzierte Aggregation kann die Aggregation typischer Nukleärkörper fördern oder umgekehrt in Mikrokörper zerlegen. Die Unterschiede zwischen diesen Typen von PML-Körpern basieren auf der Form oder deren Inhalt. Dies führt zu einer dynamischeren Sicht auf die PML-Körper, als bisher angenommen.[1]
| Stressfaktor[1] | PML NB Erscheinungsbild | Zelltypen |
|---|---|---|
| IFNs | Erhöhte Anzahl und Größe | Alle |
| As2O3 | Große PML Hülle, verminderte Anzahl | Alle |
| CdCl2/Hitzeschock | Verteilte Mikrokörper | Alle |
| Proteasominhibitor | Zunahme im Nukleolus | Primäre Zelllinien |
| Erhöhte Anzahl und Größe | Transformierte Zelllinien | |
| Actinomycin D | Groß, in der Nähe des Nukleolus | Primäre und transformierte Zelllinen |
| Verteilte Mikrokörper | Transformierte Zelllinien | |
| Ionisierende Strahlung (Gamma, UVc) | Groß, in der Nähe des Nukleolus | Primäre und transformierte Zelllinen |
Struktur eines üblichen PML-Körpers
Der klassische PML-Körper ist ein sphärisches Objekt mit einem Durchmesser von 0,1–1 µm, das ein granulares Zentrum aufweisen kann oder auch nicht. Diese Körper, von fünf bis 15 pro Kern, sind meist proteinartig und enthalten im Allgemeinen keine RNA oder DNA. Das PML-Protein bildet die äußere Hülle dieser Körper und die Partner-Proteine befinden sich in der Regel im Inneren. Dies lässt sich leicht bei einer Überexpression von PML- oder dessen Partnerproteine zeigen. Proteine können diese Hülle mit nur moderater Einschränkung ihrer Mobilität passieren.[5]
Wie mehrere andere Körper im Zellkern sind auch PML-Nukleärkörper im interchromosomalen Raum vorhanden. Dies erklärt wahrscheinlich, warum sie oft in der Nähe oder in der Nähe anderer Körper vorkommen. Obwohl sie frei von DNA sind, können PML-Körper mit einigen spezifischen chromosomalen Loci assoziiert werden, wie z. B. der MHC-Klasse-I-Gencluster-Region. Für diese wurden die PML-Körper zur Modulation der Chromatinarchitektur und Transkription vorgeschlagen. Eine elegante Studie hat ergeben, dass ein PML-Körper ständig einem Repressor-Locus gegenübergestellt wird, was die Zusammenhänge mit der transkriptionellen Regulation unterstreicht[6]. Umgekehrt können Chromatinveränderungen, die während der Transkription oder des Zellzyklus auftreten, die Struktur und Anzahl der PML-Körper verändern. PML-Körper sind während vieler Virusinfektionen stark verändert. Sie können beispielsweise virale Genome an ihrer Peripherie oder in ihrem zentralen Kern während der Infektion ruhender Zellen ansammeln.[1]
In funktionell spezialisierten PML-Körpern, wie z. B. bei der alternativen Verlängerung von Telomer-assoziierten PML-Kernkörpern (APBs) enthält der innere Kern von PML-Körpern Chromatin, in diesem Fall telomere DNA.
Bei Zellen des Immundefizienz, centromerer Instabilität und Gesichtsdysmorphie (ICF)-Syndroms enthält der innere Kern der PML-Körper perizentrisches Satellitenheterochromatin des Chromosoms 1.[3]
Bildung
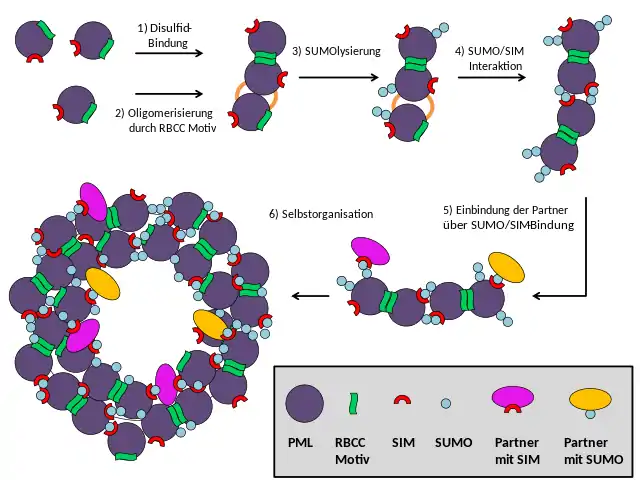
Die Bildung und strukturelle Integrität von PML-Körpern beruht auf mindestens fünf grundlegenden Prinzipien:
1) eine oxidationsgetriebene Disulfidvernetzung von PML
2) die selbstoligomerisierenden Eigenschaften des PML-RBCC-Motivs,
3) die Poly-SUMO-Ketten auf den drei Hauptziel-Lysinen
4) die nicht-kovalente Wechselwirkung von SUMO mit SUMO interagierenden Motiven (SIM) in kernkörperassoziierten Faktoren und
5) spezifische Sequenzen in verschiedenen PML-Proteinisoformen.
Im ersten Schritt der Zusammenbaus ermöglichen oxidierte PML-Monomere die Bildung von disulfidvernetzten kovalenten Multimeren. Diese organisieren sich selbst in der Kernkörper-Außenhülle. Die nicht-kovalente Homodimerisierung, die durch die RBCC-Domäne vermittelt wird, kann für den frühen PML Kernkörper-Montageschritt ähnlich wichtig sein.
Anschließend bilden Poly-Sumoylierungen, SUMO/SIM-Interaktionen und die Zugabe von SUMO- und/oder SIM-haltigen Bindungspartnern einen ausgereiften PML-Körper mit einem peripheren Gerüst, bestehend aus den sechs verschiedenen PML-Isoformen, deren SIM-Motiven und den Poly-SUMO-Ketten.
Das PML-Kernkörpergerüst bietet eine Vielzahl von potentiellen Stellen, an die ein Sortiment von PML-interagierenden, SIM-haltigen und/oder sumoylierten Partnerproteinen mehr oder weniger stark vorübergehend binden kann. Die unterschiedlichen Verweilzeiten der Bindungspartner bei den PML-Körpern sind abhängig von der Anzahl und Stärke ihrer einzelnen Interaktionsmodule. Dies steht im Einklang mit dem Vorhandensein mehrerer Sumoylierungsstellen und SIMs in wichtigen PML-Körper-Komponenten wie PML, SP100, DAXX, HIPK2, UBC9, PIASy und RNF4.[3]
Dynamik
Studien haben gezeigt, dass PML ein stabiler Bestandteil der Körper ist und dass Partnerproteine mobiler sind, obwohl sie vorübergehend in den Kernkörperchen gehalten werden. Die Austauschraten der verschiedenen PML-Isoformen zwischen Kernkörper und Karyoplasma zeigten einen deutlichen Unterschied für die PML-V Isoform. Dieses bildet eigenartige dickschalige Kernkörper und könnte die Körper in der Kernmatrix verankern. Die Körper selbst sind nicht sehr mobil, obwohl Fusionen und Spaltungen durch den Verlauf des Zellzyklus beobachtet werden können. Analysen der Kernkörper während des Zellzyklus haben Hinweise auf Duplizierung durch einen Spaltungsmechanismus während der S-Phase geliefert. Zudem haben sie die Körper-Neubildung während des M/G1-Übergangs analysiert. Während der Mitose bleiben die PML-Proteine aggregiert, werden aber phosphoryliert, desumoyliert und setzen ihre Partner frei. Vor dem Zerfall der Kernmembran in der Prometaphase verlieren PML-Körper ihre Chromatin-Anbindung, was zu einer erhöhten Mobilität führt. PML assoziiert mit Kernmembranen und Nukleoporinen während der Mitose, was die Neubildung der Kernhülle während des Übergangs von Telophase/G1 erleichtert. Schließlich, während des Übergangs von Telophase zu G1, treten SP100 und DAXX wieder in den Kern ein und binden dann an die vorgeformten Körper, SP100 zuerst und später DAXX.
Abgesehen von Arsentrioxid, das die Kernkörper-Bildung fördert, induzieren Hitzeschock oder Schwermetalle eine reversible Kernkörper-Fragmentierung durch die Bildung von hochmobilen Mikrokörpern, die frei von SUMO und den meisten Partnern sind. Einzelzellstudien nach der Stressbewältigung haben gezeigt, dass die anfängliche Größe, Lage und Anzahl der Körper irgendwie wiederhergestellt wurde, was darauf hindeutet, dass sich PML-Körper an vorbestimmten Stellen zusammensetzen können.[1][3]
Die zellzyklusabhängige Demontage von PML-Körpern beginnt mit der De-Sumoylierung von PML zu Beginn der Mitose. Die sphärische Schalenstruktur der PML-Körper bricht zusammen und andere Komponenten wie SUMO, SP100 und DAXX lösen sich oder werden entfernt. Während der Mitose aggregiert sich PML zu sogenannten mitotischen Ansammlungen von PML-Protein (MAPPs).[3]
Funktionen
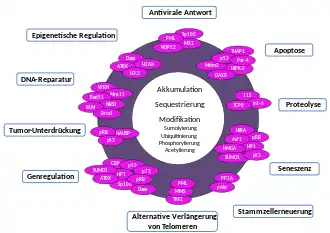
Die funktionale Vielfalt der vorübergehenden PML-Körper-Komponenten ist wahrscheinlich die Grundlage für die vielen verschiedenen biologischen Rollen, die diesen Kernstrukturen zugeschrieben werden. PML-Körper wurden funktionell mit Apoptose, Nukleärproteolyse, Seneszenz, Stammzellerneuerung, Regulierung der Genexpression, Tumorsuppression, DNA-Schadenreaktion, Telomerverlängerung und -stabilität, epigenetischer Regulierung und antiviralen Reaktionen verknüpft. Zusätzlich spielen sie eine Rolle bei der Kontrolle des Zellzyklus, zellulärer Stressreaktion, DNA-Reparatur und Proteinmodifikationsprozessen. Generell weisen die verschiedenen Aspekte der PML-Körper-Funktionen vor allem auf ihre Rolle bei der Erhaltung des Genoms hin.
Eine Hypothese für die Integration all dieser Funktionen in ein einheitliches Konzept basiert auf der Idee, dass PML-Körper ein stabiles Proteingerüst bereitstellen, auf dem die Bindungspartner für ihre effiziente posttranslationale Modifikation oder Sequestrierung assoziieren. Die kontrollierte Anreicherung bei oder Freisetzung spezifischer nukleärer Faktoren aus den Körpern kann ihre funktionelle Interaktion auf der Grundlage von massengesetzlichen Maßnahmen verbessern und dadurch die Signalkaskaden durch das Karyoplasma verfeinern.[3]
Ein Forschungsbericht legt nahe, dass sich durch die PolySUMO/polySIM-Schnittstelle in PML-Körpern phasenseparierte Flüssigkeitstropfenstrukturen in lebenden Zellen bilden können.[7] So gehören PML-Körper zur Familie der viskosen, membranfreien Kernräume, die als Phasentrennungskondensate fungieren können, die den Lipidtröpfchen entsprechen.
Die biochemische Umgebung innerhalb eines phasenseparierenden PML-Körpers unterscheidet sich von derjenigen im umgebenden Karyoplasma, und dieser Unterschied könnte einzigartige Strategien zur Regulierung nukleärer Reaktionswege ermöglichen, einschließlich der Regulation von Enzymreaktionskinetiken (d. h. posttranslationale Modifikationen), der Regulation der Spezifität biochemischer Reaktionen, der Sequestrierung von Molekülen und der Pufferung der Zellkonzentration von Molekülen.[3]
DNA-Reparatur: Das PML-Protein wird von mehreren durch DNA-Schäden aktivierten Kinasen phosphoryliert. Mehrere Studien haben gezeigt, dass PML-Körper in die DNA-Reparatur einbezogen wird, indem unterschiedliche Proteine rekrutiert oder aus Kernkörperchen freigesetzt wurden. Einige dieser Proteine werden in Kernkörperchen unter unbelasteten Bedingungen lokalisiert, während andere erst nach DNA-Schäden mit den Kernkörperchen assoziiert werden. Als Reaktion auf DNA-Schäden scheinen PML-Körper die beschädigten Stellen zu erfassen und durch einen Spaltungsmechanismus eine erhöhte Anzahl von Mikrokörpern zu bilden. Die genaue Rolle von PML (Zuschauer oder Akteur) in diesen verschiedenen Prozessen ist noch unklar.[1]
Posttranslationale Modifikation von Partner-Proteinen: Die vielleicht am meisten untersuchten posttranslationalen Modifikationen waren diejenigen am Tumorsuppressor p53. Ein auffälliger Befund war die Konzentration von p53-modifizierenden Enzymen (CBP, HDM2, HIPK2 und HAUSP) innerhalb der Kernkörper. PML-verstärkte Acetylierung, Sumoetylierung und Phosphorylierung in den Kernkörpern scheinen alle die p53-Funktion zu verbessern.
Die Kinase-Aktivität von einigen Proteinen kann durch die Translokation in die PML-Körper beeinflusst werden: Es wurden Auto-Phosphorylierung und Dephosphorylierung von Kinasen beobachtet. Es gibt auch einige Hinweise darauf, dass PML die Protein-Sumoylation in Hefe direkt verbessern kann. Die PML-Kernkörper wurden zur Verbesserung der Sumoylation spezifischer Partnerproteine vorgeschlagen. Da viele Kernkörper-assoziierte Proteine eine SIM enthalten, kann dies die Partner-Sequestrierung innerhalb der Kernkörper verbessern.[1]
Partner-Sequestrierung: Sequestrierung oder "Einlagerung" war die erste vorgeschlagene Funktion von PML-Körpern. Diese Sequestrierung zeigt sich in der relativen Akkumulation der nucleoplasmatischen und der Kernkörper-assoziierten Form von PML-Partnern, die zwischen den einzelnen Partnern und den Ebenen der PML-Expression sowie der Sumoylation sehr stark variiert. Ein gut untersuchter, abgesonderter Partner ist DAXX, ein starker Repressor, der zwischen sumolylierten Proteinen, einschließlich PML und vielen Transkriptionsfaktoren, aufteilt. Die Sequestrierung von DAXX durch Kernkörper-assoziierte und sumoylierte PMLs setzt die transkriptionelle Repression durch DNA-gebundene sumoylierte Transkriptionsfaktoren frei. Die Sequestrierung von DAXX reguliert auch die Apoptose.[1]
Partnerprotein-Abbau: Mehrere instabile Proteine wurden an PML-Körpern lokalisiert, während Proteine, die in ihrem Abbau beeinträchtigt sind sich mit PML, SUMO und Ubiquitin anhäufen. Der durch Arsentrioxid induzierte PML-Abbau hat eine Verbindung zwischen PML-Körpern und dem Proteinabbau hergestellt. Arsentrioxid löst eine anfängliche Sumoylation von K160 aus, gefolgt von einem proteasomabhängigen Abbau.[1]
Tumor-Suppressor: Neben dem korrelativen Zusammenhang zwischen Karzinogenese und PML-Expression gibt es viele experimentelle Hinweise auf eine direkte tumorsuppressive Rolle von PML. Mehrere unabhängige Studien haben gezeigt, dass die Überexpression von PML die Progression des Zellzyklus bei einer Vielzahl von Krebszelllinien verlangsamen oder blockieren kann. Weitere Analysen typischer Stress-Reaktionswege zeigten die Beteiligung der Tumorsuppressoren pRb und p53 an der PML-Überexpressions-induzierten zellulären Seneszenz. Die molekularen Details der PML-Wirkung entlang der pRB- und/oder p53-tumorsuppressiven Pfade bleiben jedoch schwer fassbar. Neben der zellulären Seneszenz spielt PML eine wesentliche funktionelle Rolle bei der Apoptose. Der PML-Verlust korreliert mit dem Fortschreiten vieler Krebsarten und in den meisten Fällen ist eine niedrige PML-Expression mit einer schlechten Prognose verbunden. Die Tumorsuppressorfunktion von PML-Körpern kann mit ihrer Fähigkeit verknüpft werden, viele Proteine zu akkumulieren, die an der Reaktion auf DNA-Schäden und Reparaturwegen beteiligt sind. Dies stabilisiert vermutlich DNA-Reparaturkomplexe und verstärkt ihre Aktivitäten. Obwohl die physiologische Funktion von PML und der PML-Nukleärkörper noch nicht vollständig aufgeklärt ist, kann ihre tumorunterdrückende Rolle durch die Unterstützung von DNA-Schadensverläufen für alle diese potenziellen Funktionen gemeinsam sein.[3]
Regulation der Transkription: Es gibt verschiedene Hinweise für den Einfluss von PML und PML-Kernkörperchen auf die Transkriptionsregulation. Bei einer Untersuchung der molekularen Wechselwirkungen in der Zelle wurden 166 verschiedene Proteine erkannt, die mit PML physisch oder funktionell interagieren können. Die Hälfte dieser Proteine ist direkt oder indirekt in der Transkriptionskontrolle involviert. Transkriptionsfaktoren wie CBP/p300, Daxx und p53 lokalisieren vorübergehend an den PML-Kernkörperchen, wo sie modifiziert und/oder kompartmentalisiert werden. Die PML Isoform IV kann auch als Transkriptionsregulator agieren, wenn es künstlich an Promotoren rekrutiert wird.[2]
Antivirale Antwort: PML-Körper sind in der Lage, eine intrinsische Repression der Virusreplikation zu vermitteln, indem sie eine epigenetische Stilllegung von Virusgenomen induzieren oder durch Einschließungsmechanismen. Darüber hinaus entwickeln sich PML-Körper als Co-Aktivatoren von zellulären Genen, die antivirale Aktivitäten wie Zytokine und ISGs ausüben. Komponenten des PML-Körpers werden selbst durch die IFN-Behandlung hochreguliert. Um diese antiviralen Aktivitäten zu überwinden, haben viele Viren antagonistische Proteine entwickelt, die einzelne PML-Körper-Komponenten oder die gesamte Zellstruktur beeinflussen.[8] Die Proteine von humanen Papilloma-Viren können spezifisch an einige PML-Isoformen binden, so dass diese dem proteasomalen Abbau zugeführt werden. Das Protein des Adenovirus interagiert ebenfalls spezifisch mit einer PML-Isoform. Diese Interaktion führt zum proteasomalen Abbau von PML und damit zur Auflösung der Kernkörperchen.[2]
Alternative Verlängerung von Telomeren: Das unbegrenzte Wachstumspotenzial von Krebszellen erfordert die Erhaltung ihrer Telomere. Dies wird häufig durch Reaktivierung der Telomerase erreicht. In einem signifikanten Anteil der Tumore ist jedoch eine alternative Verlängerung des Telomer-(ALT-)Mechanismus aktiv. Der molekulare Mechanismus des ALT-Weges bleibt schwer fassbar. Insbesondere die Rolle der charakteristischen Komplexe von PML-Körpern mit Telomeren, den ALT-assoziierten PML-Körpern (APBs), wird derzeit untersucht. PML-Körper in normalen Zellen enthalten keine Nukleinsäuren. In ALT-positiven Zellen lokalisiert sich jedoch eine Teilmenge von PML-Körpern mit telomerer DNA.[9]
Einzelnachweise
- V. Lallemand-Breitenbach, H. de The: PML Nuclear Bodies. In: Cold Spring Harbor Perspectives in Biology. Band 2, Nr. 5, 1. Mai 2010, ISSN 1943-0264, S. a000661–a000661, doi:10.1101/cshperspect.a000661, PMID 20452955, PMC 2857171 (freier Volltext) – (cshlp.org [abgerufen am 13. März 2019]).
- Tobias Ulbricht: Die Rolle der PML-Kernkörperchen bei der Regulation der MHC Klasse II-Expression. (PDF) In: Dissertation. 31. März 2010, abgerufen am 2. April 2019.
- Peter Hemmerich, Klaus Weisshart, Shamci Monajembashi, Christian Hoischen: Multimodal Light Microscopy Approaches to Reveal Structural and Functional Properties of Promyelocytic Leukemia Nuclear Bodies. In: Frontiers in Oncology. Band 8, 2018, ISSN 2234-943X, doi:10.3389/fonc.2018.00125 (frontiersin.org [abgerufen am 5. April 2019]).
- sp100 Protein, SP100 nuclear antigen - Creative BioMart. Abgerufen am 7. April 2019.
- M. Lang, T. Jegou, I. Chung, K. Richter, S. Munch: Three-dimensional organization of promyelocytic leukemia nuclear bodies. In: Journal of Cell Science. Band 123, Nr. 3, 1. Februar 2010, ISSN 0021-9533, S. 392–400, doi:10.1242/jcs.053496 (biologists.org [abgerufen am 13. April 2019]).
- Toshiro Tsukamoto, Noriyo Hashiguchi, Susan M. Janicki, Tudorita Tumbar, Andrew S. Belmont: Visualization of gene activity in living cells. In: Nature Cell Biology. Band 2, Nr. 12, Dezember 2000, ISSN 1465-7392, S. 871–878, doi:10.1038/35046510 (nature.com [abgerufen am 24. März 2019]).
- Salman F. Banani, Allyson M. Rice, William B. Peeples, Yuan Lin, Saumya Jain: Compositional Control of Phase-Separated Cellular Bodies. In: Cell. Band 166, Nr. 3, Juli 2016, S. 651–663, doi:10.1016/j.cell.2016.06.010 (elsevier.com [abgerufen am 5. April 2019]).
- Myriam Scherer, Thomas Stamminger: Emerging Role of PML Nuclear Bodies in Innate Immune Signaling. In: Journal of Virology. Band 90, Nr. 13, 1. Juli 2016, ISSN 0022-538X, S. 5850–5854, doi:10.1128/JVI.01979-15, PMID 27053550, PMC 4907236 (freier Volltext) – (asm.org [abgerufen am 5. April 2019]).
- Inn Chung, Sarah Osterwald, Katharina I. Deeg, Karsten Rippe: PML body meets telomere: The beginning of an ALTernate ending? In: Nucleus. Band 3, Nr. 3, Mai 2012, ISSN 1949-1034, S. 263–275, doi:10.4161/nucl.20326, PMID 22572954, PMC 3414403 (freier Volltext) – (tandfonline.com [abgerufen am 13. April 2019]).