Lipid Raft
Lipid Rafts (zu deutsch ‚Lipidflöße‘) nennt man spezielle Bereiche in Zellmembranen. Sie zeichnen sich durch einen relativ hohen Gehalt an Sphingomyelinen, Glycosphingolipiden und Cholesterin aus.[1]
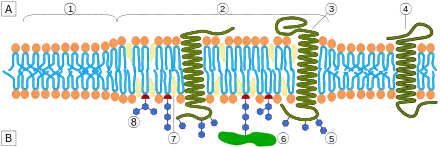
Eigenschaften
Lipid Rafts ordnen sich als flüssigkristalline Phase in eigenen Bereichen der Zellmembran an. Sie sind an verschiedenen Prozessen beteiligt, z. B. Sortierung von Proteinen für die Zellmembran im Golgi-Apparat, Endocytose und Signaltransduktion. Sie werden zurzeit als dynamische geordnete Nanostrukturen aus Sterolen und Sphingolipiden mit bestimmten Membranproteinen definiert, die durch Lipid-Lipid-, Protein-Lipid- und Protein-Protein-Interaktionen verschmelzen können.[2] Lipid rafts wurden aufgrund ihrer relativen Stabilität gegenüber einer Extraktion mit Tensiden auch als detergent-insoluble membranes, detergent-resistant membranes, glycosphingolipids-enriched membranes, detergent-insoluble glycolipid-rich membranes, und Triton-insoluble floating fraction bezeichnet. Proteine mit GPI-Anker reichern sich bevorzugt in lipid rafts an.
Obwohl das genaue Modell unbekannt ist, minimieren die lipid rafts die freie Energie zwischen den beiden Phasen – es bilden sich Domänen einer typischen Größe, die sich weder mischen, noch weiter fusionieren.[3] Die lipid rafts bilden dabei eine geordnete Phase innerhalb der ungeordneten Phase der Phospholipide der restlichen Zellmembran. Gut beobachtbar ist die Entmischung zwischen der "Liquid Ordered Phase" (geordnete Phase, L0) und der "Liquid Disordered Phase"" target="_blank" rel="nofollow" (ungeordnete Phase, Ld oder Lα).[4] Möglicherweise bieten die Lipid rafts aufgrund ihrer vergleichsweise höheren Dicke einen Sortiermechanismus für Membranproteine nach der Länge ihrer Transmembrandomäne.[5]
Analyse
Probleme bei der Darstellung von lipid rafts ist das Fehlen des thermodynamischen Gleichgewichts in der Zusammensetzung der Zellmembran,[6] was die Untersuchung der Zusammensetzung erschwert.[7] Zudem besitzen künstliche Membranen eine niedrigere Anzahl an Membranproteinen als natürliche.
Ihre Beobachtung ist aufgrund ihrer geringen Größe von 10–200 nm schwierig,[8] weil sie nahe am Auflösungsvermögen klassischer Lichtmikroskope) liegt. Jedoch bietet Fluoreszenzmikroskopie eine Möglichkeit lipid rafts sichtbar zu machen. Benutzt werden z. B. Farbstoffe, die sich zwischen den Domänen einlagern wie Laurdan,[9] Filipin[10] oder kopfgelabelte Farbstoffe wie Texas Red, die sich aufgrund ihrer Größe bevorzugt in der ungeordneten Phase einlagern. Die B-Untereinheit des Choleratoxins bindet an das Gangliosid GM1 in lipid rafts.[11][12] Durch eine Fluoreszenzmarkierung können Proteine markiert werden, die sich in lipid rafts anreichern, z. B. Lck-GFP. Cholesterol kann durch Filipin, Nystatin oder Amphotericin B gebunden werden. Durch Methyl-beta-Cyclodextrin kann der Zellmembran Cholesterol entzogen werden. Durch HMG-CoA-Reduktase-Hemmer kann die Biosynthese des Cholesterols gehemmt werden.[6] Im Gegensatz zu den anderen Bereichen der Zellmembran sind lipid rafts in einer 1 % (m/V) Triton X-100-Lösung bei 4 °C unlöslich und können daher mit milden Tensiden isoliert werden.[13]
Zur Analyse häufig verwendete Methoden sind z. B. die Fluoreszenzmikroskopie, die Fluoreszenzkorrelationsspektroskopie und Kreuzkorrelationsspectroskopie (FCS/FCCS) zur Messung der Mobilität, der Förster-Resonanzenergietransfer zur Messung der Nachbarschaft zweier Moleküle und die optische Pinzette zur Messung der Viskosität.[14][6] Weiterhin wird die Rasterkraftmikroskopie, die Scanning Ion Conductance Microscopy (SICM) und die Dual Polarisation Interferometry, Kernspinresonanzspektroskopie sowie ELISA, Western Blot und FACS verwendet.[15][16] Per FLIP oder FRAP kann die seitliche Mobilität verfolgt werden.
Kritik am Lipid Raft-Konzept
Am Konzept der lipid rafts wurden verschiedene Punkte kritisiert.[17] Obwohl Lipid Rafts im Fluoreszenzmikroskop in Modellmembranen beobachtet werden können, ist deren Nachweis in lebenden Zellen bisher nicht eindeutig gelungen. Über deren durchschnittliche Größe (1–1000 nm) und Lebensdauer herrscht bei Forschern bisher keine Einigkeit. Daher ist es unklar, in welcher Form Lipid rafts existieren.
Geschichte
Spezielle Bereiche einer Zellmembran (englisch membrane microdomain) wurden erstmals in den 1970er Jahren durch Stier und Sackmann[18] sowie Klausner und Karnovsky postuliert.[19] Die Arbeitsgruppe von Karnovsky konnte den ungleichmäßigen Aufbau der Zellmembran anhand der unterschiedlichen Fluoreszenzlebensdauer von 1,6-Diphenyl-1,3,5-hexatrien in der Zellmembran zeigen.[8] 1988 stellten Kai Simons in Deutschland und Gerrit van Meer in den Niederlanden ein Konzept von Mikrodomänen in Lipidmembranen vor, in denen sich Cholesterin, Glycolipide und Sphingolipide anreichern.[20][21] Sie nannten diese Domänen „Lipid Rafts“, da diese wie Flöße auf der zweidimensionalen Lipiddoppelschicht der Zellmembran im Fluid-Mosaik-Modell schwimmen.
Einzelnachweise
- S. Thomas, R. S. Kumar, T. D. Brumeanu: Role of lipid rafts in T cells. In: AITE. 52, 2004, S. 215–224.
- D. Lingwood, K. Simons: Lipid rafts as a membrane-organizing principle. In: Science. Band 327, Nummer 5961, Januar 2010, S. 46–50, ISSN 1095-9203. doi:10.1126/science.1174621. PMID 20044567.
- Linda J Pike: The Challenge of Lipid Rafts. In: Journal of Lipid Research. Okt 2008, S. 1–17.
- A. Rietveld, K. Simons: The differential miscibility of lipids as the basis for the formation of functional membrane rafts. In: Biochim. Biophys. Acta. 1376 (3), November 1998, S. 467–479. PMID 9805010.
- K. Simons, J. L. Sampaio: Membrane organization and lipid rafts. In: Cold Spring Harbor perspectives in biology. Band 3, Nummer 10, Oktober 2011, S. a004697, ISSN 1943-0264. doi:10.1101/cshperspect.a004697. PMID 21628426. PMC 3179338 (freier Volltext).
- J. A. Allen, R. A. Halverson-Tamboli, M. M. Rasenick: Lipid raft microdomains and neurotransmitter signalling. In: Nature reviews. Neuroscience. Band 8, Nummer 2, Februar 2007, S. 128–140, ISSN 1471-003X. doi:10.1038/nrn2059. PMID 17195035.
- Ken Jacobson, Ole G. Mouritsen, Richard G. W.Anderson: Lipid rafts: At a crossroad between cell biology and physics. In: Nature Cell Biology. Band 9, Nr. 1, 2007, S. 7–14, doi:10.1038/ncb0107-7, PMID 17199125.
- L. J. Pike: The challenge of lipid rafts. In: The Journal of Lipid Research. Band 50, 2008, S. S323, doi:10.1194/jlr.R800040-JLR200, PMID 18955730, PMC 2674732 (freier Volltext).
- L. A. Bagatolli: To see or not to see: lateral organization of biological membranes and fluorescence microscopy. In: Biochim. Biophys. Acta. Band 1758, Nr. 10, 2006, S. 1451–1456, doi:10.1016/j.bbamem.2006.05.019.
- G. Gimpl, K. Gehrig-Burger: Cholesterol reporter molecules. In: Bioscience Reports. Band 27, Nummer 6, Dezember 2007, S. 335–358, ISSN 0144-8463. doi:10.1007/s10540-007-9060-1. PMID 17668316.
- P. A. Orlandi, P. H. Fishman: Filipin-dependent inhibition of cholera toxin: evidence for toxin internalization and activation through caveolae-like domains. In: Journal of Cell Biology. Band 141, Nummer 4, Mai 1998, S. 905–915, ISSN 0021-9525. PMID 9585410. PMC 2132770 (freier Volltext).
- J. Sanchez, J. Holmgren: Cholera toxin - a foe & a friend. In: The Indian journal of medical research. Band 133, Februar 2011, S. 153–163, ISSN 0971-5916. PMID 21415489. PMC 3089046 (freier Volltext).
- K. B. Kim, J. S. Lee, Y. G. Ko: The isolation of detergent-resistant lipid rafts for two-dimensional electrophoresis. In: Methods in molecular biology (Clifton, N.J.). Band 424, 2008, S. 413–422, ISSN 1064-3745. doi:10.1007/978-1-60327-064-9_32. PMID 18369879.
- E. Klotzsch, G. J. Schütz: A critical survey of methods to detect plasma membrane rafts. In: Philosophical transactions of the Royal Society of London. Series B, Biological sciences. Band 368, Nummer 1611, Februar 2013, S. 20120033, ISSN 1471-2970. doi:10.1098/rstb.2012.0033. PMID 23267184. PMC 3538433 (freier Volltext).
- S. Thomas, R. S. Kumar, S. Casares, T.-D. Brumeanu: Sensitive detection of GM1 lipid rafts and TCR partitioning in the T cell membrane. In: Journal of Immunological Methods. Band 275, Nr. 1–2, 2003, S. 161–168, doi:10.1016/S0022-1759(03)00014-0, PMID 12667680.
- Sunil Thomas, Rajeev Kumar, Anca Preda-Pais, Sofia Casares, Teodor-D. Brumeanu: A Model for Antigen-Specific T-Cell Anergy: Displacement of CD4-p56lck Signalosome from the Lipid Rafts by a Soluble, Dimeric Peptide-MHC Class II Chimera1. In: The Journal of Immunology. Band 170, Nr. 12, 2003, S. 5981–5992, PMID 12794125.
- Sean Munro: Lipid rafts: elusive or illusive? In: Cell. Band 115, Nr. 4, 2003, S. 377–388, doi:10.1016/S0092-8674(03)00882-1, PMID 14622593.
- A. Stier, E. Sackmann: Spin labels as enzyme substrates Heterogeneous lipid distribution in liver microsomal membranes. In: Biochimica et Biophysica Acta (BBA) - Biomembranes. Band 311, Nr. 3, 1973, S. 400–408, doi:10.1016/0005-2736(73)90320-9, PMID 4354130.
- Morris J. Karnovsky, Alan M. Kleinfeld, Richard L. Hoover, Richard D. Klausner: The concept of lipid domains in membranes. In: The Journal of Cell Biology. Band 94, Nr. 1, 1982, S. 1–6, doi:10.1083/jcb.94.1.1, PMID 6889603, PMC 2112185 (freier Volltext).
- S. Thomas, A. P. Pais, S. Casares, T. D. Brumeanu: Analysis of lipid rafts in T cells. In: Molecular Immunology. 41, 2004, S. 399–409.
- Zeljka Korade: Lipid rafts, cholesterol, and the brain. In: Neuropharmacology. 55, 2008, S. 1265–1273.
Weblinks
- Differentielle Assoziation von Psychopharmaka mit Membranmikrodomänen in Bezug auf die Modulation liganden-gesteuerter Ionenkanäle. (Memento vom 6. Januar 2013 im Webarchiv archive.today) Bedeutung von lipid rafts und Transportproteinen für die Wirkung von Psychopharmaka