Haloquadratum walsbyi
Haloquadratum walsbyi ist eine Art (Spezies) halophiler (salzliebender) Archaeen.[2][3] Sie ist die bisher einzige offiziell beschriebene Spezies (und damit Typusspezies) der Gattung Haloquadratum (mit Schreibvariante Haloquadra).[3][4][5]
| Haloquadratum walsbyi | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
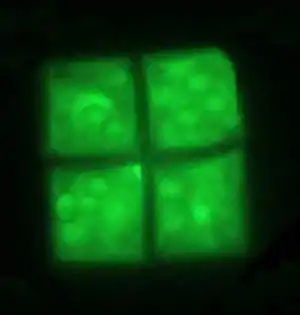
Blatt (englisch sheet) mit 4 Zellen | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name der Gattung | ||||||||||||
| Haloquadratum | ||||||||||||
| Burns et al. 2007 | ||||||||||||
| Wissenschaftlicher Name der Art | ||||||||||||
| Haloquadratum walsbyi | ||||||||||||
| Burns et al. 2007 |
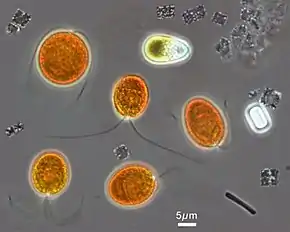
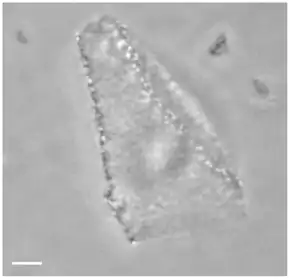
Die Archaeenspezies H. walsbyi (auch als Hqr. walsbyi abgekürzt) ist insofern ungewöhnlich, als ihre Zellen die Form flacher quadratischer Kästchen haben.[6][4] Außer durch diese flachen, quadratischen Zellen zeichnet sich die Art auch durch ihre ungewöhnliche Fähigkeit aus, in wässriger Umgebung mit hohen Konzentrationen von Natrium- und Magnesiumchlorid (NaCl respektive MgCl2) zu überleben.[7][8]
H. walsbyi wurde 1980 von Anthony E. Walsby im Gavish Sabkha, einem hypersalinen Küstenbecken nördlich von Scharm El-Scheich auf der Sinai-Halbinsel in Ägypten, entdeckt,[9][10][11] aber erst 2004 von Burns et al. kultiviert[12] und 2007 offiziell beschrieben.[13] Nach ihrem Entdecker werden sie gelegentlich „Walsby-Haloarchaeen“ (englisch Walsby’s square archaea; früher auch „Walsby-Bakterien“ etc.) genannt.[7][3] H. walsbyi ist außer im Gavish Sabkha in etlichen anderen Solebecken auf der ganzen Erde nachweisbar und gilt in hypersalinen Gewässern als Kosmopolit als weltweit vertreten. Die Art ist wahrscheinlich einer der häufigsten Vertreter in diesen salzigen Ökosystemen.[14] Beispielfundorte sind die Salinen des Roten Meeres und Salinen in Niederkalifornien.[4]
Morphologie und Stoffwechsel
H. walsbyi war zunächst durch ihre mit etwa 0,15 μm extrem dünne quadratische Morphologie (Gestalt) aufgefallen.[3] Inzwischen wurden aber auch andere Vertreter der Halobacteria (Haloarchaeen) mit ähnlicher Morphologie gefunden, insbesondere in der Gattung Haloarcula (H. quadrata, H. californiae und H. sinaiiensis – letztere Spezies ebenfalls aus dem Gavish Sabkha).[11][15]
Die Zellen besitzen eine Fülle von Gasvesikel (en. gas vacuoles, intracellular refractile bodies). Durch die Gasfüllung bekommen die Zellen den nötigen Auftrieb, um eine obere Position in der Wassersäule einzunehmen und möglichst viel Licht aufnehmen zu können.[16][3] Gleichzeitig sorgen spezielle Vorkehrungen für den erforderlichen Schutz vor der Ultraviolettstrahlung der Sonne (s. u.).
Neben der großen Anzahl an lichtbrechenden, gasgefüllten Vakuolen enthalten die Zellen typischerweise Granulen (Einlagerungen) von Polyhydroxyalkanoaten (PHA).[3]
Die einzelne quadratischen Zellen sind miteinander zu großen „Blättern“ (englisch sheets) verbunden, die manchmal bis zu 40 µm groß sind. Die Breite liegt hierbei zwischen 0,1 und 0,5 µm, es handelt sich also um extrem flache und dünne Zellen. Das Oberflächen-Volumen-Verhältnis ist hierbei also sehr hoch, wodurch wahrscheinlich die Aufnahme von Stoffen erleichtert wird.[17]
Diese Blätter sind äußerst zerbrechlich und die Verbindungen zwischen den Zellen können leicht getrennt werden.[3]
Wegen der schwierigen Kultivierbarkeit geht die Erforschung des Stoffwechsels von H. walsbyi nur langsam voran, es dürften noch etliche Einzelheiten auf ihre Entdeckung warten, nachdem das Genom sequenziert wurde.
Entdeckung und Beschreibung
H. walsbyi wurde erstmals 1980 von Anthony E. Walsby in dem küstennahen Solebecken (arabisch سبخة Sabkha, auch Sabcha oder Sebkha, DMG Sabḫa) Gavish (arabisch جافيش) entdeckt. Diese Saline, benannt nach Eliezer Gavish (1937–1981), einem Erforscher solcher Solebecken, wurde früher auch englisch Nabq sea-marginal pool genannt. Sie ist Teil des Nabeq-Naturschutzgebiets (en. Nabq Protected Area)[18] auf der Sinai-Halbinsel nördlich von Scharm El-Scheich (Ägypten) am Golf von Akaba.[10][11]
Die Spezies wurden jedoch erst 2004 im Labor kultiviert[12] und 2007 von Burns et al. offiziell beschrieben.[13]
Systematik
Haloquadratum_walsbyi wurde früher in die Familie Halobacteriaceae (Ordnung Halobacteriales) gestellt, wird aber inzwischen zusammen mit der Gattung Haloferax der neu eingerichteten Familie Haloferaceae (Ordnung Haloferacales) zugeordnet.[5][19]
Die hier angegebene Taxonomie (mit Stand Februar 2022) basiert auf folgenden Quellen:
- B – Bacterial Diversity (BacDive), Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ)[20]
- L – List of Prokaryotic names with Standing in Nomenclature (LPSN), Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ)[5]
- N – National Center for Biotechnology Information (NCBI)[19]
- M – MicrobeWiki, Kenyon College, Department of Biology[3]
In neuer Taxonomie (N) gehört die Gattung zur Familie Haloferacaceae Gupta et al. 2015 emend. Gupta et al. 2016 in der Ordnung Haloferacales Gupta et al. 2015 der Klasse Halobacteria (Haloarchaeen). Das manchmal angefügte hochgestellte ‚T‘ bei einem Stamm kennzeichnet den Typus- oder Referenzstamm. Die Gattung ist monotypisch, wenn man von Stämmen (etwa aus der Metagenomik) absieht, die weitere Spezies darstellen könnten:
Gattung: Haloquadratum Burns et al. 2007 (N,M) mit Synonym (M) Haloquadra
- Spezies: Haloquadratum walsbyi Burns et al. 2007 (N) bzw. Bolhuis et al. 2004 (N) – Typus
- Stamm: Haloquadratum walsbyi C23T (B,N[21]) – alias DSM 16854T (B,N) und JCM 12705 (B,N) – Australien[22][13][23]
- Stamm: Haloquadratum walsbyi J07HQW1 (N) – Australien (Victoria), gefiltertes Oberflächenwasser aus dem Lake Tyrrell[1]
- Stamm: Haloquadratum walsbyi J07HQW2 (N) – Australien (Victoria), gefiltertes Oberflächenwasser aus dem Lake Tyrrell
- Stamm: Haloquadratum walsbyi DSM 16790 (B,N) alias HBSQ001 (B,N[24]) und JCM 12895 (B) – Spanien[13][23]
- Stamm: Haloquadratum walsbyi JCM 15557 (B)
- Stamm: Haloquadratum walsbyi LT53-19[25]
- Spezies: Haloquadratum sp. J07HQX50 (N) – Australien (Victoria), gefiltertes Oberflächenwasser aus dem Lake Tyrrell (Metagenomik)[26]
- Spezies: Haloquadratum sp. JCM 15065 (B)[25] alias Haloquadratum sp. Bajool9[25] und Haloarchaeon BJCX4_extB9.1 (B,N[27]) – Bajool-Salinen,[28] Queensland, Australien
Etymologie
Der Gattungsname Haloquadratum leitet sich ab von altgriechisch ἅλς háls, deutsch ‚Salz, Meer‘, Genitiv halos; und lateinisch quadratum ‚quadratisch‘ (als Neutrum), bedeutet also etwas „salziges/Salz benötigendes Quadratisches“.[5]
Ökologie
H. walsbyi wurde zum ersten Mal in salzhaltigen Tümpeln in Ägypten entdeckt und entnommen, ist aber in hypersalinen Gewässern auf der ganzen Welt zu finden.[3]
Wenn Meerwasser verdunstet, kommt es zu einer hohen Konzentration und Ausfällung von Kalziumkarbonat und Kalziumsulfat, was zu einer hypersalinen, natriumchloridreichen Sole führt. Weitere Verdunstung führt zur Ausfällung von Natriumchlorid oder Halit und in der Folge zu einer konzentrierten Magnesiumchlorid-Sole, die englisch auch als bittern (vgl. Bittersalz alias Magnesiumsulfat) oder japanisch als Nigari bezeichnet wird. In der letzten Phase der Halitbildung, bevor die Magnesiumchloridkonzentration die Sole steril werden lässt, gedeiht H. walsbyi und kann bis zu 80 % der Biomasse der Sole ausmachen.[3] Diese hohen Salzkonzentrationen stellen daher für H. walsbyi eine optimale Wachstumsumgebung dar. Laut Bolhuis et al. sind sie die bei ihrer Entdeckung hyperhalophilsten (bei Weitem salzverträglichsten) bekannten Organismen. Dies macht sie für die Erforschung ihrer genauen Rolle im Ökosystem interessant. Auch aufgrund ihrer spezifischen Morphologie sind weitere Erkenntnisse über die Evolution und morphologische Anpassung von Archaeen zu erwarten.[3][29]
Das Art-Epitheton walsbyi ist neulateinischer Genitiv von walsbyus, meint also „des (Forschers) Walsby“.[5]
Genom
Inzwischen wurde die Kartierung des Genoms von H. walsbyi für mehrere Stämme, u. a. auch den Referenzstamm, abgeschlossen, was zu einem besseren Verständnis der Genealogie und Taxonomie dieser Organismen und ihrer Rolle im Ökosystem geführt hat. Ein Genomvergleich der spanischen und australischen Isolate (Stämme HBSQ001 respektive C23T) zeigt eine große Übereinstimmung, insbesondere auch was die Anordnung der Gene (Syntänie) anbetrifft. Dies deutet stark auf eine rasche globale Ausbreitung der Art hin.[23]
H. walsbyi HBSQ001
Das 3,1 Mbp (Megabasenpaare) große Genom des Stammes H. walsbyi HBSQ001 (alias DSM 16790) wurde durch Bolhuis et al. (2006) ausführlich beschrieben.[8][3] Das Genom dieses Stammes besteht aus einem großen DNA-Doppelstrang („Chromosom“) mit einer Länge von 3.132.494 bp mit 2738 offenen Leserahmen (en. open reading frames, ORFs) und einem Plasmid mit der Bezeichnung PL47 (46.867 bp, 39 ORFs).[24][8][3] Das Genom hat eine gut konservierte „Haloarchael“-Region, die den Replikationsursprung und das Zellteilungskontrollprotein (en. cell division control protein bzw. cell division cycle) CDC6_1 ([en]) enthält.[8][3]
Das Genom hat einen GC-Gehalt von 47,9 %, was im Vergleich zu dem allgemein hohen GC-Gehalt von 60–70 % anderer Haloarchaea bemerkenswert niedrig ist. Dieser GC-Gehalt ist im gesamten Genom konstant. Es wird vermutet, dass der niedrigere GC-Gehalt durch die große Anzahl von Photolyasen kompensiert wird, die helfen, mit der hohen UV-Bestrahlung in den flachen Küstenlagunen fertig zu werden. Obwohl sich der GC-Gehalt von dem der meisten anderen Haloarchaea unterscheidet, sind die meisten von H.walsbyi kodierten typischen Haloarchaea-Proteine auf der Ebene der Aminosäuresequenz (also im Proteom, nach der Translation (Biologie) an den Ribosomen) hoch konserviert.[8][3] Weiter bemerkenswert an diesem Genom sind unter anderem
- die geringe Kodierungsdichte (en. coding density) von 75 % (im Vergleich zu 86–91 % bei anderen Haloarchaea),
- die Tatsache, dass es für zwei unterschiedliche Bacteriorhodopsin-Proteine kodiert,
- sowie die Nutzung eines Dihydroxyacetons über ein von Phosphoenolbrenztraubensäure (en. phosphoenolpyruvate) abhängigen Phosphotransferase-Systems (PTS).[30][8][3]
Das ca. 47 kbp große Plasmid von H. walsbyi HBSQ001 (alias DSM 16790) zeigt ebenfalls eine konstante GC-Verteilung. Es enthält viele Gene, von denen viele vermutlich konserviert (en. hypothetical or conserved hypothetical) sind. Die meisten der identifizierten Gene kodieren Proteine, die an der Erhaltung, Replikation und Restriktionsmodifikation (en. Restriction modification system) des Plasmids beteiligt sind. Interessanterweise haben die meisten dieser Gene einen bakteriellen oder vciralen Ursprung und stammen nicht von (irgendwelchen) Archaeen.[8][3]
H. walsbyi C23T
Burns et al. (2007) und Dyall-Smith et al. (2011) zeigten, dass der Referenzstamm H. walsbyi C23T gleich drei Plasmide besitzt, neben einem großen mit der Bezeichnung PL100 (Länge 100.258 bp) gibt es noch zwei kleine: PL6A (6129 bp) und PL6B (6056 bp), beide zirkulär.[21][25]
Die Plasmide der PL6-Familie von Haloquadratum sind nun deshalb interessant, weil sie Virus-verwandt sind.[25]
Insbesondere sind PL6A und PL6B von H. walsbyi C23T untereinander eng verwandt und zeigen die gleiche Gensyntänie (-anordnung), haben aber im Allgemeinen nur wenig Ähnlichkeit mit anderen bekannten Sequenzen – mit bemerkenswerten Ausnahmen:
Ein Paar (anhand der Gensequenz) vorhergesagter Proteine (Hqrw_6002 und sein Ortholog Hqrw_7002) ähneln Proteinen, wie sie von vielen Haloarchaeen kodiert werden und sich in der Nähe von mit Haloviren verwandten Genclustern befinden. Zwar ist die Funktion derartiger Gene noch unbekannt (Stand 2018), aber man hat festgestellt, dass solche Gene immer von Mitgliedern der Virusgattung Betapleolipovirus (Familie Pleolipoviridae) übertragen werden, wie zum Beispiel HRPV-3 (Spezies Betapleolipovirus HHPV3[31]).
Ein zweites Paar von Plasmidproteinen (Hqrw_6005 und sein Ortholog Hqrw_7005) zeigte sich als schwach verwandt mit einem anderen Halovirus-Protein bisher unbekannter Funktion, HisV_gp16, das man von dem zitronen- oder spindelförmigen Halovirus His1 (Spezies Salterprovirus His1[32]) aus der Gattung Salterprovirus (Familie Halspiviridae) findet.[25]
Auch in anderen Proben aus hypersalinen Umgebungen Australiens wurden seitdem weitere Vertreter der PL6-Plasmidfamile gefunden (Dyall-Smith et al, 2018). Alle Vertreter zeigen eine starke Konservierung des Geninhalts und Syntänie; sowie ein Protein (F3), das sehr ähnlich zu einem der Betapleolipoviren ist; sowie ein Protein (R6), das einem His1-Protein (His1V_gp16) ähnelt.[25]
H. walsbyi LT53-19
Dieser Stamm hat aus der PL6-Familie das Plasmid pLT53-7, Länge: 7.045 bp, GC-Gehalt: 51.[33][25]
Haloquadratum sp. Bajool9
Dieser Stamm und mögliche Spezies hat aus der PL6-Familie das Plasmid PBAJ9-6, Länge: 6.213 bp, GC-Gehalt: 53.[27][25]
Plasmid pLTMV-6
Aus der Metagenomik (zugehöriges Archaeon 2018 noch unbekannt) stammt das (vorhergesagte) Plasmid pLTMV-6, Länge: 5884 bp, GC-Gehalt: 53.[34][25]
Viren
Ein bedeutendes Merkmal hypersaliner Umgebungen ist die hohe Anzahl an virusähnlichen Partikeln (en. viruslike particles, VLPs), die sie beherbergen. Solche Solen sind sogar die Wassersysteme mit der höchsten bekannten Konzentration an Viren (bzw. VLPs), nämlich bis zu 1010 VLPs/mℓ. Diese Umgebungen zeigen oft auch die höchsten Verhältnisse von Viren zu Zellen — bis zu 300:1, während in normalen aquatischen Systemen das Verhältnis bei 10:1 oder darunter legt. Darüber hinaus zeigen Studien, dass auch die Vielfalt an VLP-Morphologien (Gestalten oder Formen) in diesen Systemen sehr groß ist.[35]
Die einzigen bisher (Stand 2021) verfügbaren isolierten Haloarchaea-Viren infizieren Wirte mit hohem GC-Gehalt. Abgesehen von den VLPs sind tatsächlich keine Viren von Archaeen mit niedrigem GC-Gehalt (wie beispielsweise H. walsbyi) bekannt.
Haloarchaeen mit hohem GC-Gehalt (z. B. Halorubrum, Halobacterium, Haloferax usw.) sind in Kultursammlungen gut vertreten. H. walsbyi ist zwar in vielen Umgebungen weltweit sehr verbreitet, aber ähnlich wie die Nanohaloarchaea nur schwer kultivierbar. Dies dürfte der Grund sein, dass es für diese als Wirte keine bekannten Viren gibt.[35]
Die oben erwähnten Plasmid-Studien und die Hohe Viruslast der Habitate geben aber einen sehr deutlichen Hinweis, dass es zumindest zwei Gruppen von Haloquadratum-Viren geben sollte: zum einen Mitglieder der Gattung Salterprovirus der Familie Halspiviridae, zum anderen solche aus der Gattung Betapleolipovirus der Familie Pleolipoviridae.
Literatur
- David G. Burns, Helen M. Camakaris, Peter H. Janssen, Mike L. Dyall-Smith: Cultivation of Walsby's square haloarchaeon. In: FEMS Microbiol. Lett.. 238, Nr. 2, 15. September 2004, S. 469–473. PMID 15358434. PDF
- Aharon Oren, Antonio Ventosa: International Committee on Systematic Bacteriology Subcommittee on the taxonomy of Halobacteriaceae. Minutes of the meetings, 16 August 1999, Sydney, Australia. In: Int. J. Syst. Evol. Microbiol.. 50, Nr. 3, 1. Mai 2000, S. 1405–1407. doi:10.1099/00207713-50-3-1405. PMID 10843089.
- Norman Edwin Gibbons: Family V. Halobacteriaceae fam. nov.. In: Robert Earle Buchanan, N. E. Gibbons (Hrsg.): Bergey's Manual of Determinative Bacteriology, 8. Auflage, The Williams & Wilkins Co., Baltimore 1974, ISBN 0-683-01117-0.
- Lenin Maturrano, Fernando Santos, Ramon Rosselló-Mora, Josefa Antón: Microbial Diversity in Maras Salterns, a Hypersaline Environment in the Peruvian Andes. In: ASM Applied and Environmental Microbiology, Band 72, Nr. 6, 2. Juni 2006, S. 3887–3895; doi:10.1128/AEM.02214-05, PMC 1489619 (freier Volltext), PMID 16751493.
- M. Alam, M. Claviez, D. Oesterhelt, M. Kessel: Flagella and motility behaviour of square bacteria. In: European Molecular Biology Organization Journal, Band 3, Nr. 12, 1. Dezember 1984, S. 2899-2903; PMID 6526006, PMC 557786 (freier Volltext)
- Henk Bolhuis, Evelien M. te Poele, Francisco Rodriguez-Valera: Isolation and cultivation of Walsby's square archaeon. In: Environmental Microbiology, Band 6, Nr. 12, 24. September 2004, S. 1287–1291; doi:10.1111/j.1462-2920.2004.00692.x, PMID 15560825.
- Haloquadratum walsbyi. Max-Planck-Institut für Biochemie, Department of Membrane Biochemistry.
- Bernhard Tschitschko et al.: Genomic variation and biogeography of Antarctic haloarchaea, in: Microbiome, Band 6, Nr. 113, 2018; doi:10.1186/s40168-018-0495-3.
Ricardo Cavicchioli: Behind the paper: Antarctic haloarchaea – a unique Microbiome, in: Microbiology, 19. Juni 2018.
Weblinks
- PubMed-Referenzen zu Haloquadratum
- PubMed-Central-Referenzen zu Haloquadratum
- GoogleScholar-Referenzen zu Haloquadratum
- Genome-Sequence, JCVI, Memento im Webarchiv vom 3. August 2008.
- NCBI: Haloquadratum, Details: Haloquadratum Burns et al. 2007 (genus); graphisch: Haloquadratum, auf LifeMap NCBI Version.
- Haloquadra, MicrobeWiki, Kenyon College, Department of Biology.
- LPSN: Genus Haloquadratum Burns et al. 2007
Einzelnachweise
- MapCarta: Lake Tyrrell, Australia (englisch) / Lake Tyrrell, Australian (deutsch)
- OneZoom: Haloquadratum walsbyi
- Haloquadra syn. Haloquadratum (Gattung) und Haloquadratum walsbyi (Spezies), MicrobeWiki, Kenyon College, Department of Biology.
- Lenin Maturrano, Fernando Santos, Ramon Rosselló-Mora, Josefa Antón: Microbial Diversity in Maras Salterns, a Hypersaline Environment in the Peruvian Andes. In: ASM Applied and Environmental Microbiology, Band 72, Nr. 6, 2. Juni 2006, S. 3887–3895; doi:10.1128/AEM.02214-05, PMC 1489619 (freier Volltext), PMID 16751493.
- LPSN: Haloquadratum Burns et al. 2007, Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ)
- W. Stoeckenius: Walsby's square bacterium: fine structure of an orthogonal procaryote. In: J. Bacteriol.. 148, Nr. 1, 1. Oktober 1981, S. 352–360. doi:10.1128/JB.148.1.352-360.1981. PMID 7287626. PMC 216199 (freier Volltext).
- Aharon Oren, Antonio Ventosa, M. Carmen Gutiérrez, Masahiro Kamekura: Haloarcula quadrata sp. nov., a square, motile archaeon isolated from a brine pool in Sinai (Egypt). In: International Journal of Systematic Bacteriology, Band 49, Nr. 3, 1. Juli 1999, S. 1149–1155; doi:10.1099/00207713-49-3-1149, PMID 10425773, ResearchGate.
- Henk Bolhuis, Peter Palm, Andy Wende, Michaela Falb, Markus Rampp, Francisco Rodriguez-Valera, Friedhelm Pfeiffer, Dieter Oesterhelt: The genome of the square archaeon Haloquadratum walsbyi: life at the limits of water activity. In: BMC Genomics, Band 7, Nr. 1, S. 169, 4. Juli 2006; doi:10.1186/1471-2164-7-169, PMID 16820047, PMC 1544339 (freier Volltext).
- Anthony Edward Walsby: A square bacterium. In: Nature. 283, Nr. 5742, 3. Januar 1980, S. 69–71. bibcode:1980Natur.283...69W. doi:10.1038/283069a0.
- Gerald M. Friedman: Hypersaline Ecosystems: The Gavish Sabkha, Springer-Verlag Berlin Heidelberg, 1985, Serie: Ecological Studies 53, ISBN 3-642-70292-9; doi:10.1007/978-3-642-70290-7. Siehe insbes. Fig. 3.18.
- Barbara Javor, Carol Requadt, Walther Stoeckenius: Box-shaped halophilic bacteria. In: Journal of Bacteriology. Band 151, Nr. 3, September 1982, ISSN 0021-9193, S. 1532–1542, doi:10.1128/jb.151.3.1532-1542.1982, PMID 6286602, PMC 220435 (freier Volltext) – (Epub 2. Februar 2021).
- David G. Burns, Helen M. Camakaris, Peter H. Janssen, Mike L. Dyall-Smith: Cultivation of Walsby's square haloarchaeon. In: FEMS Microbiol. Lett.. 238, Nr. 2, 15. September 2004, S. 469–473. PMID 15358434. PDF
- David G. Burns, Peter H. Janssen, Takashi Itoh, Masahiro Kamekura, Zhuo Li, Grant Jensen4, Francisco Rodríguez-Valera, Henk Bolhuis, Mike L. Dyall-Smith: Haloquadratum walsbyi gen. nov., sp. nov., the square haloarchaeon of Walsby, isolated from saltern crystallizers in Australia and Spain. In: International Journal of Systematic and Evolutionary Microbiology. 57, Nr. 2, 1. Februar 2007, S. 387–392. doi:10.1099/ijs.0.64690-0. PMID 17267984.
- Elleuche, Skander: Extreme Lebensräume: Wie Mikroben unseren Planeten erobern. Springer Verlag GmbH., Berlin 2018, Kapitel: Überleben in gesättigten Salzlösungen.
- Marina Torreblanca, F. Rodriguez-Valera, Guadalupe Juez, Antonio Ventosa, Masahiro Kamekura, Morris Kates: Classification of Non-alkaliphilic Halobacteria Based on Numerical Taxonomy and Polar Lipid Composition, and Description of Haloarcula gen. nov. and Haloferax gen. nov. In: Systematic and Applied Microbiology, Band 8, Nr. 1–2, Juli 1986, S. 89–99; doi:10.1016/S0723-2020(86)80155-2, PDF.
- Aharon Oren, Nuphar Pri-El, Orr Shapiro, Nachshon Siboni: Buoyancy studies in natural communities of square gas-vacuolate archaea in saltern crystallizer ponds. In: Saline Systems. 2, 14. April 2006, S. 4. doi:10.1186/1746-1448-2-4. PMID 16613609. PMC 1459177 (freier Volltext).
- A. Stolz: Extremophile Mikroorganismen, 2017, Springer Spektrum, Berlin, Heidelberg, ISBN 978-3-662-55595-8 doi:10.1007/978-3-662-55595-8_7
- MapCarta: Nabq, Village in Eqypt (englisch) / Nabq, Dorf in Ägypten (deutsch)
- NCBI: Haloquadratum, Details: Haloquadratum Burns et al. 2007 (genus); graphisch: Haloquadratum, auf LifeMap NCBI Version.
- BacDive: Haloquadratum - Bacterial Diversity Metadatabase, Deutsche Sammlung von Mikroorganismen und Zellkulturen (DSMZ)
- NCBI: , Haloquadratum walsbyi DSM 16790 Genomes
- Peter Palm: Haloquadratum walsbyi: Limited Diversity in a Global Pond, auf: Science Buzz, Juli 2019.
- Mike L. Dyall-Smith, Friedhelm Pfeiffer, Kathrin Klee, Peter Palm, Karin Gross, Stephan C. Schuster, Markus Rampp, Dieter Oesterhelt: Haloquadratum walsbyi: Limited Diversity in a Global Pond. In: PLOS ONE. 6, Nr. 6, 20. Juni 2011, S. e20968. bibcode:2011PLoSO...620968D. doi:10.1371/journal.pone.0020968. PMID 21701686. PMC 3119063 (freier Volltext).
- NCBI: , Haloquadratum walsbyi C23 Genomes
- Mike L. Dyall-Smith, Friedhelm Pfeiffer: The PL6-Family Plasmids of Haloquadratum Are Virus-Related. In: Frontiers in Microbiology, Band 23, Nr. 9, 23. Mai 2018, 1070; doi:10.3389/fmicb.2018.01070, PMID 29875763, PMC 5974055 (freier Volltext)
- Sheila Podell, Juan A. Ugalde, Priya Narasingarao, Jillian F. Banfield, Karla B. Heidelberg, Eric E. Allen: Assembly-driven community genomics of a hypersaline microbial ecosystem. In: PLoS ONE, Band 8, Nr. 4, E61692, 18. April 2013; doi:10.1371/journal.pone.0061692, PMID 23637883.
- NCBI: haloarchaeon BJCX4_extB9.1 (species), insbes. Haloquadratum walsbyi strain Bajool9 genome assembly, plasmid: I
- MapCarta: Bajool, Australia (englisch) / Bajool, Australian (deutsch)
- Henk Bolhuis, Evelien M. te Poele, Francisco Rodriguez-Valera: Isolation and cultivation of Walsby's square archaeon. In: Environmental Microbiology, Band 6, Nr. 12, 24. September 2004, S. 1287–1291; doi:10.1111/j.1462-2920.2004.00692.x, PMID 15560825.
- Phosphotransferase-System, Lexikon der Biologie, auf spektrum.de
- ICTV: ICTV Taxonomy history: Betapleolipovirus HRPV3, Stand: MSL#36, veröffentlicht 2021.
- ICTV: ICTV Taxonomy history: Salterprovirus His1, Stand: MSL#36, veröffentlicht 2021.
- NCBI: Haloquadratum walsbyi isolate LT53-19 genome assembly, plasmid: I
- NCBI: viral metagenome genome assembly, plasmid: pLTMV-6
- FernandoSantosMaría D.Ramos-BarberoJosefaAntón: Metagenomes of Archaeal Viruses in Hypersaline Environments. In: Encyclopedia of Virology (Fourth Edition), Band 4, 2021, S. 414–418; doi:10.1016/B978-0-12-809633-8.21227-2.