Bacteriorhodopsin
Bacteriorhodopsin (BR) ist ein integrales Membranprotein in der Zellmembran des extremophilen Organismus Halobacterium salinarum (Halobakterien). Das Protein BR ist der Lichtenergie-Konverter der phototrophen Energiegewinnung von Halobacterium salinarum. Die von Halobacterium salinarum vollzogene Phototrophie unterscheidet sich grundlegend von Photosynthesen, zum Beispiel von der oxygenen Photosynthese der Pflanzen. Die Lichtenergie wird hier nicht zur Spaltung von Wasser genutzt, sondern dient über den Energiekonverter BR zum Aufbau eines Protonen-Konzentrationsunterschieds zwischen dem Innenraum (Cytoplasma) und Außenraum des Bakteriums. Dieser Konzentrationsunterschied ist die Energiequelle für die ATP-Synthase. Aufgrund seiner außergewöhnlichen Funktionalität ist das Protein von großem wissenschaftlichem Interesse, zudem wurde für BR eine Reihe technischer Anwendungen vorgeschlagen.
| Bacteriorhodopsin | ||
|---|---|---|
| ||
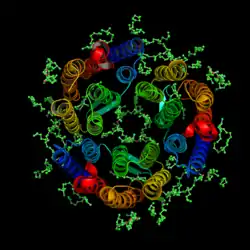
| Biologischer Aufbau, nach PDB 1M0K. | ||
| Masse/Länge Primärstruktur | 249 Aminosäuren | |
| Sekundär- bis Quartärstruktur | Homotrimer; multipass Membranprotein | |
| Bezeichner | ||
| Gen-Name(n) | bop | |
| Externe IDs | ||
| Transporter-Klassifikation | ||
| TCDB | 3.E.1 | |
| Bezeichnung | ionenverlagerndes mikrobielles Rhodopsin | |
| Vorkommen | ||
| Homologie-Familie | Bacteriorhodopsin | |
| Übergeordnetes Taxon | Halobakterien | |
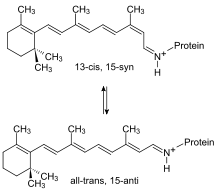
die beiden Grundformen des Retinal, in denen es im Dunkeln vorkommt
Geschichte
Den Namen „Bacteriorhodopsin“ für ein Protein der Purpurmembran (purple membrane) von Halobacterium salinarum (Halobakterien) schlugen 1971 der Biochemiker Dieter Oesterhelt und der Elektronenmikroskopiker Walther Stoeckenius vor. Gemeinsam mit dem Biophysiker Allen E. Blaurock hatten sie nachgewiesen, dass die Purpurmembran von Halobacterium ein kristallin vorliegendes Retinalprotein enthält.[1][2][3] Zwei Jahre später charakterisierten Oesterhelt und Stoeckenius die Funktion der Purpurmembran als lichtabhängigen Protonentransport, und interpretierten dies als eine einfache Form der Photosynthese.[4] Spätestens nachdem Richard Henderson und Nigel Unwin 1975 mittels Elektronenmikroskopie ein Strukturmodell des Proteins erstellt hatten, avancierte Bakteriorhodopsin zu einem Modellobjekt von Bioenergetik, Membran- und Strukturbiologie.[5] Ab Mitte der 1970er-Jahre erschienen zum Thema mehr als hundert Publikationen pro Jahr, und es wurden verwandte Proteine wie Halorhodopsin oder Sensorhodopsine beschrieben. Pläne zur technischen Nutzung der das Bakteriorhodopsin enthaltenden Purpurmembran finden sich seit den späten 1970er-Jahren.[6]
Aufbau des Proteins
Das Protein des BR besteht aus 248 Aminosäuren, die, in sieben näherungsweise parallelen alpha-Helices angeordnet, die Zellmembran durchziehen und eine Pore bilden. In dieser Pore befindet sich ein an das Protein gebundenes Retinalmolekül. Retinal ist das Chromophor des Moleküls und über eine Amidbindung, in diesem Zusammenhang meist als Schiffsche Base bezeichnet, an die Aminfunktion der Aminosäure Lys216 gebunden. Unter physiologischen Bedingungen liegt das Chromophor nur als all-trans- und 13-cis-Isomer vor. Die Isomerisierung erfolgt unter Lichteinwirkung.
BR bildet in der Zellmembran des Halobacterium salinarum, zusammengelagert zu Trimeren, zweidimensional kristalline Bereiche aus. Diese bis zu fünf Mikrometer großen Bereiche, in denen BR-Trimere in zweidimensional hexagonaler Anordnung in der Lipiddoppelschicht vorliegen, heißen Purpurmembran (PM). Die Einbettung des BR in die Purpurmembran führt zu einer bemerkenswerten Stabilität des Proteins gegenüber physikalisch-chemischen Einflüssen. So bleiben Farbe und photochemische Aktivität der PM in Gegenwart von Sauerstoff sowie im trockenen Zustand erhalten.
Funktion des Proteins
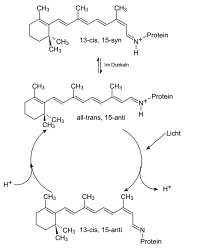
BR kann als eine von Lichtenergie getriebene molekulare Maschine betrachtet werden, die Protonen pumpt. In einem mehrstufigen Prozess, initiiert durch die lichtinduzierte Isomerisierung des Chromophors und angetrieben durch Veränderungen der Protonenaffinitäten von Aminosäurefunktionen, werden Protonen von der cytoplasmatischen zur extracellulären Seite durch die Pore des Proteins verschoben. Auslösend für die gerichtete Protonenverschiebung ist die Isomerisierung des Retinal-Chromophors infolge von Lichtabsorption. Das Chromophor liegt im unbelichteten Zustand als Mischung von all-trans- und 13-cis-Retinal, nach Belichtung lediglich in der 13-cis-Konfiguration vor. Dies hat, aufgrund der Einbettung des Chromophors, strukturelle Veränderungen des Proteins zur Folge, was sich unmittelbar auf den zunächst protonierten Zustand der Schiffschen Base auswirkt. Dieses Proton befindet sich nach der Isomerisierung in einer energetisch ungünstigen Umgebung und wird an den unmittelbaren Wechselwirkungspartner der Schiffschen Base, Asp85, in extrazelluläre Richtung abgegeben. Daran ist eine Folge von vier weiteren unidirektionalen Protonenverschiebungen geknüpft, bevor abschließend der Ausgangszustand des Proteins wiederhergestellt wird und ein neuer Zyklus durchlaufen werden kann. Dieses lichtgetriebene Pumpen von Protonen ist an eine zyklische Folge spektroskopisch unterscheidbarer Zustände des Proteins geknüpft. Diese Folge wird Photozyklus genannt. Das Durchlaufen des Photozyklus infolge von Belichtung ist mit einem reversiblen Farbwechsel von Purpur (B-Zustand, Absorptionsmaximum 570 nm) nach Gelb (M-Zustand, Absorptionsmaximum 410 nm) verbunden.[7]
Technische Anwendungen
Sicherheitsdruckpigment
Eine erste Anwendung fand Bacteriorhodopsin als photochromes Pigment in Sicherheitsdruckfarben.[8]
Proteinspeicher mit 50 Terabyte Kapazität
Möglich wird dieser enorme Speicherplatz durch die Lichtempfindlichkeit des BR-Proteins. Fällt Licht auf das Protein, so verwandelt es sich in eine Reihe unterschiedlicher Moleküle, von denen jedes eine charakteristische Form und Farbe aufweist, bevor es in den Ausgangszustand zurückkehrt. Dieses Zwischenstadium dient der Gewinnung von chemischer Energie und hält in der Natur etwa eine Stunde an. Durch Modifizierung der DNA des Bakteriums kann dieser Zustand mehrere Jahre lang aufrechterhalten werden. Übertragen auf das binäre System der Computertechnik, bedeutet der Grundzustand eine 0 und der veränderte Zustand eine 1.[9]
Literatur
- Jean-Baptiste Waldner: Nano-informatique et intelligence ambiante. Hermes Science Publications, London 2006, ISBN 978-2-7462-1516-0
Einzelnachweise
- Allen E. Blaurock, Walther Stoeckenius: Structure of the Purple Membrane. In: Nature New Biology. Band 233, Nr. 39, September 1971, S. 152–155, doi:10.1038/newbio233152a0.
- Dieter Oesterhelt, Walther Stoeckenius: Rhodopsin-like Protein from the Purple Membrane of Halobacterium halobium. In: Nature New Biology. Band 233, Nr. 39, September 1971, S. 149–152, doi:10.1038/newbio233149a0.
- W. Stoeckenius: From membrane structure to bacteriorhodopsin. In: The Journal of Membrane Biology. Band 139, Nr. 3, Mai 1994, S. 139–148, doi:10.1007/BF00232619.
- Dieter Oesterhelt, Walther Stoeckenius: Functions of a New Photoreceptor Membrane. In: Proceedings of the National Academy of Sciences. Band 70, Nr. 10, Oktober 1973, S. 2853–2857 (pnas.org).
- R. Henderson, P. N. T. Unwin: Three-dimensional model of purple membrane obtained by electron microscopy. In: Nature. Band 257, Nr. 5521, September 1975, S. 28–32, doi:10.1038/257028a0.
- Mathias Grote, Maureen A. O'Malley: Enlightening the life sciences: the history of halobacterial and microbial rhodopsin research. In: FEMS Microbiology Reviews. Band 35, Nr. 6, November 2011, S. 1082–1099, doi:10.1111/j.1574-6976.2011.00281.x.
- Heiko Patzelt, Bernd Simon, Antonius terLaak, Brigitte Kessler, Ronald Kühne, Peter Schmieder, Dieter Oesterhelt, Hartmut Oschkinat: The structures of the active center in dark-adapted bacteriorhodopsin by solution state NMR spectroscopy. In: Proceedings of the National Academy of Sciences. Bd. 99, Nr. 15, 2002, S. 9765–9770, doi:10.1073/pnas.132253899.
- Martin Neebe: Bakteriorhodopsin als multifunktionales photochromes Farbpigment für die Sicherheitstechnik. Dissertation Philipps-Universität Marburg 2003, DNB 968682308.
- David J. Mehrl, Thomas F. Krile: Multiplexed Holographic Data Storage in Bacteriorhodopsin. Juni 1999 (nasa.gov).