G-Quadruplex
Nucleinsäuresequenzen, die besonders viel Guanin enthalten, sind in der Lage, viersträngige Strukturen auszubilden, die G-Quadruplexe, G-Tetraden oder G4-DNA genannt werden.

Eigenschaften
G-Quadruplexe bestehen aus einer quadratischen Anordnung von Guaninmolekülen, die von Wasserstoffbrückenbindungen durch Ausbildung von Hoogsteen-Basenpaaren stabilisiert werden. Zusätzlich werden sie durch ein monovalentes Kation, meist Kalium, im Zentrum der Tetrade stabilisiert. Sie können von DNA, RNA, LNA und PNA erzeugt werden und können dabei intramolekular, bimolekular und tetramolekular sein, das heißt aus einer, zwei oder vier Untereinheiten zusammengesetzt sein. Abhängig von der Richtung der Stränge oder Teilstränge, welche die Tetraden bilden, werden die Strukturen als parallel oder antiparallel beschrieben.
Die allgemeine Sequenz von G-Quadruplexen ist
- G3–5 NL1 G3–5 NL2 G3–5 NL3 G3–5
mit den beliebigen Schleifen NL1–3, deren Länge zwischen einem und sieben Nukleotiden ist.[1][2]
Quadruplexe kommen seltener in Exonen vor.[3] An G-Quadruplexe bindet die RecQ-Helicase, die das Werner-Syndrom verursacht und an das Bloom-Syndrom-Protein bindet.[3] Ein künstliches Zinkfingerprotein mit dem Namen Gq1 wurde auch entwickelt, das auf G-Quadruplexe passt, ebenso deren spezifische Antikörper.[3]
Kationische Porphyrine binden ebenso an G-Quadruplexe, ebenso das Molekül Telomestatin.
Quadruplexe in den Telomeren

Die sich wiederholenden DNA-Sequenzen der Telomere einer Vielzahl von Organismen bilden G-Quadruplex-Strukturen. Dies konnte vielfach in vitro („im Reagenzglas“), in einigen Fällen auch in vivo (in lebenden Zellen) gezeigt werden. Telomere im Menschen und allen Vertebraten bestehen aus vielen Wiederholungen der DNA-Sequenz TTAGGG. Die von dieser Struktur gebildeten Quadruplexe wurden mittels NMR und Röntgenstrukturanalyse inzwischen gut erforscht. Die Anordnung dieser Quadruplexe in den Telomeren scheint die Aktivität des Enzymes Telomerase zu reduzieren, das verantwortlich für die Erhaltung der Länge der Telomere ist und in 85 % aller Krebsarten eine Rolle spielt. Dies ist ein wichtiger Ansatz für die Entwicklung von Medikamenten.
Nichttelomerische Quadruplexe
Ebenso stieg in letzter Zeit das Interesse an Quadruplexen, die nicht in Telomeren vorkommen. Verantwortlich dafür ist die Arbeit von Hurley mit dem Protoonkogen c-Myc, welches anscheinend einen Quadruplex in einer Region ausbildet, die für Nuklease überempfindlich ist und die für die Genaktivität eine wichtige Rolle spielt.[4] Danach fand man heraus, dass viele andere Gene G-Quadruplexe in Regionen der Promotoren haben, dazu zählte auch das Beta-globin Gen der Hühner, die menschliche Ubiquitin-Ligase RFP2 und die Protoonkogene c-kit, bcl-2, VEGF, H-ras und N-ras.
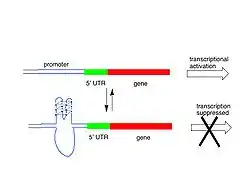
Gesamtuntersuchungen von Genomen auf die Ausbildung von G-Quadruplexen wurden durchgeführt, welche 376.000 Putative Quadruplex Sequenzen (PQS) im menschlichen Genom identifiziert haben, obwohl wahrscheinlich nicht alle davon in vivo so auftreten.[3] Eine ähnliche Studie hat Putative G-Quadruplexes in Prokaryonten entdeckt. Es gibt mehrere denkbare Möglichkeiten, wie Quadruplexe die Genaktivität kontrollieren könnten, entweder durch Herabregulation oder Heraufregulation. Ein mögliches Modell wird nebenstehend anschaulich dargestellt, hierbei blockiert ein G-Quadruplex in oder nahe bei einem Promotor die Transkription des Genes, dadurch wird dieses also deaktiviert. In einem anderen Modell hilft ein Quadruplex am nichtcodierenden DNA-Strang eine offene Gestalt der codierenden DNA zu erhalten und verbessert so die Expression des jeweiligen Gens.
Literatur
- Stephen Neidle, Shankar Balasubramanian (Hrsg.): Quadruplex Nucleic Acids. Cambridge Royal Society of Chemistry, 2006, ISBN 978-0-85404-374-3, doi:10.1039/9781847555298.
- Jay E. Johnson, Jasmine S. Smith, Marina L. Kozak, F. Brad Johnson: In vivo veritas: Using yeast to probe the biological functions of G-quadruplexes. In: Biochimie. Band 90, Nr. 8, 2008, S. 1250–1263, doi:10.1016/j.biochi.2008.02.013, PMID 18331848.
- Pooja Rawal, Veera Bhadra Rao Kummarasetti, Jinoy Ravindran, Nirmal Kumar, Kangkan Halder, Rakesh Sharma, Mitali Mukerji, Swapan Kumar Das, Shantanu Chowdhury: Genome-wide prediction of G4 DNA as regulatory motifs: Role in Escherichia coli global regulation. In: Genome Research. Band 16, Nr. 5, 2006, S. 644–655, doi:10.1101/gr.4508806, PMID 16651665.
- Xu Hou, Wei Guo, Fan Xia, Fu-Qiang Nie, Hua Dong, Ye Tian, Liping Wen, Lin Wang, Liuxuan Cao, Yang Yang, Jianming Xue, Yanlin Song, Yugang Wang, Dongsheng Liu, Lei Jiang: A Biomimetic Potassium Responsive Nanochannel: G-Quadruplex DNA Conformational Switching in a Synthetic Nanopore. In: Journal of the American Chemical Society. Band 131, Nr. 22, 2009, S. 7800–7805, doi:10.1021/ja901574c.
Weblinks
- Greglist – Auflistung mit möglichen von Quadruplexen regulierten Gene
- GRSDB – Datenbank mit G-quadruplexen an RNA Erzeugung
- G-quadruplex Resource Site
- Cockayne-Syndrom B (CSB) und G-Quadruplex:
Long-range four-stranded DNA structures found to play a role in rare ageing disease, auf: EurekAlert! vom 6. Dezember 2021
Carly Cassella: Unusual 'Quadruple Helix' Structure in DNA May Be Behind Rare Aging Syndrome, auf sciencealert vom 8. Dezember 2021
Einzelnachweise
- A. K. Todd, M. Johnston, S. Neidle: Highly prevalent putative quadruplex sequence motifs in human DNA. In: Nucleic Acids Res. (2005), Band 33, Nr. 9, S. 2901–2907. PMID 15914666; PMC 1140077 (freier Volltext).
- S. Burge, G. N. Parkinson, P. Hazel, A. K. Todd, S. Neidle: Quadruplex DNA: sequence, topology and structure. In: Nucleic Acids Res. (2006), Band 34, Nr. 19, S. 5402–5415. PMID 17012276; PMC 1636468 (freier Volltext).
- J. L. Huppert, S. Balasubramanian: Prevalence of quadruplexes in the human genome. In: Nucleic Acids Res. (2005), Band 33, Nr. 9, S. 2908–2916. PMID 15914667; PMC 1140081 (freier Volltext).
- A. Siddiqui-Jain, C. L. Grand, D. J. Bearss, L. H. Hurley: Direct evidence for a G-quadruplex in a promoter region and its targeting with a small molecule to repress c-MYC transcription. In: Proc Natl Acad Sci U S A. (2002), Band 99, Nr. 18, S. 11593–11598. PMID 12195017; PMC 129314 (freier Volltext).