Bradyrhizobium
Bradyrhizobium (früher auch Agromonas Ohta & Hattori 1985[1] oder Photorhizobium Eaglesham et al. 1990[2]) ist eine Gattung gramnegativer Bodenbakterien, von denen viele Stickstoff (d. h. atmosphärischen Stickstoff: Distickstoff N2) fixieren. Die Stickstofffixierung ist ein wichtiger Teil des Stickstoffkreislaufs. Pflanzen können atmosphärischen Stickstoff selbst nicht nutzen; sie sind auf Stickstoffverbindungen wie Nitrate oder stickstofffixierende Symbiosepartner angewiesen.
| Bradyrhizobium | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
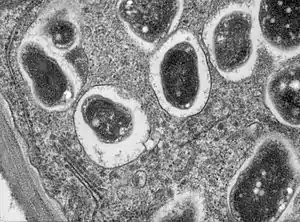
Querschnitt durch eine Sojabohnenwurzel (Glycine max ‚Essex‘). Bradyrhizobium japonicum infiziert die Wurzeln und geht eine stickstofffixierende Symbiose ein. Teil einer Zelle mit einzelnen Bakteroiden in ihren Symbiosomen. | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| Bradyrhizobium | ||||||||||||
| Jordan 1982 |
Beschreibung
Die Bakterien der Gattung Bradyrhizobium sind gramnegative und stäbchenförmig mit beweglich (motil) vermöge einer einzigen polaren (endständigen) oder subpolaren (nicht ganz polaren) Geißel. Sie sind weit verbreitete bodenbewohnende Mikroorganismen, die mit Leguminosen (Hülsenfrüchtlern) symbiotische Beziehungen eingehen können, bei denen sie Stickstoff (N2) im Austausch gegen Kohlenhydrate der Pflanze fixieren. Wie andere Rhizobien sind viele Mitglieder dieser Gattung in der Lage, atmosphärischen Stickstoff in solcher Weise zu fixieren, dass die Produkte für andere Organismen leicht verwertbar sind. Bradyrhizobium-Bakterien sind auch wichtige Bestandteile mikrobieller Gemeinschaften in Waldböden, wobei die aus diesen Böden isolierten Stämme allerdings in der Regel nicht zur Stickstofffixierung oder Knöllchenbildung fähig sind.[3] Sie sind langsam wachsend, im Gegensatz zu den schnell wachsenden Rhizobium-Arten. In einem flüssigen Medium benötigen Bradyrhizobium-Arten etwa 3–5 Tage, um eine mäßige Trübung zu erzeugen, und 6–8 Stunden, um ihre Populationsgröße zu verdoppeln. Sie wachsen am besten mit Pentosen als Kohlenstoffquelle.[4] Einige Stämme (z. B. USDA 6 und CPP) sind in der Lage, Kohlenmonoxid (CO) aerob zu Kohlendioxid (CO2) zu oxidieren.[5]
Etymologie
Die Vorsilbe des Gattungsnamens ‚Brady-‘ bezieht sich auf das lateinisch/griechische Adjektiv bradus für ‚langsam‘. Es weist auf die langsame Wachsgeschwindigkeit von Bradyrhizobium hin. Der restliche Namensteil ‚-rhizobium‘ verweist auf die vergleichbare Lebensweise wie bei der Gattung Rhizobium, nämlich ein Leben (‚-bio‘) in den Wurzelknöllchen von Pflanzen (rhizo bedeutet ‚Wurzel‘).
Systematik
Der folgenden Systematik liegt die List of Prokaryotic names with Standing in Nomenclature (LPSN) mit Stand vom 23. Mai 2021 zugrunde.[6]
Gattung: Bradyrhizobium Jordan 1982 (syn. Agromonas Ohta & Hattori 1985)[7]
Bestätigte Arten
Die Gattung Bradyrhizobium umfasst die folgenden Arten (Spezies):[6]
- B. agreste Klepa et al. 2021[8]
- B. algeriense Ahnia et al. 2019
- B. americanum Ramírez-Bahena et al. 2017
- B. amphicarpaeae Bromfield et al. 2019
- B. arachidis Wang et al. 2013
- B. archetypum Helene et al. 2020
- B. australiense Helene et al. 2020
- B. betae Rivas et al. 2004[9][Anm. 1]
- B. cajani Araújo et al. 2017
- B. canariense Vinuesa et al. 2005[9][Anm. 2]
- B. centrosematis corrig. Ramírez-Bahena et al. 2017[Anm. 3]
- B. cosmicum Wasai-Hara et al. 2020
- B. cytisi Chahbourne et al. 2011
- B. daqingense JY Wang et al. 2012
- B. denitrificans (Hirsch & Müller 1986) van Berkum et al. 2011
- B. diazoefficiens Delamuta et al 2013[9][Anm. 4]
- B. diversitatis Serenato Klepa et al. 2021[8]
- B. elkanii Kuykendall et al. 1993[9][Anm. 4]
- B. embrapense Delamuta et al. 2015
- B. erythrophlei Yao et al. 2015
- B. ferriligni Yao et al. 2015
- B. frederickii de Oliveira Urquiaga et al. 2019
- B. ganzhouense Lu et al. 2014
- B. glycinis Serenato Klepa et al. 2021[8]
- B. guangdongense Li et al. 2015
- B. guangxiense Li et al. 2015
- B. hipponense Rejili et al. 2020
- B. huanghuaihaiense Zhang et al. 2012
- B. icense Durán et al. 2014
- B. ingae da Silva et al. 2014
- B. iriomotense Islam et al. 2010
- B. ivorense Fossou et al. 2020
- B. japonicum (Kirchner 1896) Jordan 1982 – Typus[9][Anm. 5]
- B. jicamae Ramírez-Bahena et al. 2009
- B. kavangense Lasse Grönemeyer et al. 2015
- B. lablabi Chang et al. 2011
- B. liaoningense Xu et al. 1995[9][Anm. 4]
- B. lupini Peix et al. 2015
- B. manausense Silva et al. 2014
- B. mercantei Helene et al. 2017
- B. murdochi Helene et al. 2020
- B. namibiense Grönemeyer et al. 2017
- B. nanningense Li et al. 2020
- B. neotropicale Zilli et al. 2014[11][Anm. 6]
- B. niftali Klepa et al. 2019
- B. nitroreducens Jang et al. 2020
- B. oligotrophicum (Ohta & Hattori 1985) Ramírez-Bahena et al. 2013
- B. ottawaense Yu et al. 2014
- B. pachyrhizi Ramírez-Bahena et al. 2009
- B. paxllaeri Durán et al. 2014
- B. retamae Guerrouj et al. 2013
- B. rifense Chahboune et al. 2012
- B. ripae Bünger et al. 2018
- B. shewense Aserse et al. 2018
- B. stylosanthis Marçon Delamuta et al. 2016
- B. subterraneum Gronemeyer et al. 2015
- B. symbiodeficiens Bromfield et al. 2020
- B. tropiciagri Delamuta et al. 2015
- B. vignae Grönemeyer et al. 2016
- B. viridifuturi Helene et al. 2015
- B. yuanmingense Yao et al. 2002[9][Anm. 7]
Vorläufig zugewiesene Arten
Die folgenden Arten wurden zwar beschrieben, aber nicht nach dem International Code of Nomenclature of Bacteria (ICNB) validiert:[6]
- „B. brasilense“ Martins da Costa et al. 2017
- „B. campsiandrae“ Cabral Michel et al. 2021
- „B. centrolobii“ Michel et al. 2017
- „B. forestalis“ Martins da Costa et al. 2018
- „B. guangzhouense“ Li et al. 2019
- „B. macuxiense“ Michel et al. 2017
- „B. sacchari“ de Matos et al. 2017
- „B. uaiense“ Cabral Michel et al. 2020
- „B. valentinum“ Durán et al. 2014
- „B. zhanjiangense“ Li et al. 2019
- „B. sp. USDA 4362“ (syn. Photorhizobium thompsonianum BTAi1[12][2])
Eine Gattung „Photorhizobium“ wird bei LPSN nicht gelistet, nur beim National Center for Biotechnology Information (NCBI), dort aber mit einer ganzen Reihe vorläufig benannter Arten.[13] Es ist daher nicht ganz klar, ob die gesamte (vorgeschlagene oder vorläufige) Gattung Photorhizobium nun Bradyrhizobium zugeschlagen werden kann, oder nur die eine bezeichnete Spezies.
Phylogenie
Die gegenwärtig akzeptierte Taxonomie der Gattung Bradyrhizobium basiert auf der List of Prokaryotic names with Standing in Nomenclature (LPSN),[6] die Phylogenie basiert auf der Analyse des gesamten Genoms:[14]
| Nitrobacteraceae |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
Knöllchenbildung
Knöllchen (englisch nodules) sind Wucherungen an den Wurzeln von Hülsenfrüchten, in denen sich die symbiotischen Bakterien befinden. Die Pflanzenwurzeln scheiden Aminosäuren und Zucker in die Rhizosphäre (Wurzelbereich im Erdboden) aus. Die symbiotischen Bakterien[Anm. 8] wandern zu den Wurzeln und heften sich an die Wurzelhaare. Die Pflanze setzt daraufhin Flavonoide frei, wodurch in den Bakterien die Expression der so genannten Nod-Genen auslöset wird. Die Expression dieser Gene führt zur Produktion der sog. Nod-Faktoren: Enzymen, die das Aufrollen der Wurzelhaare einleiten. Während dieses Prozesses werden die Bakterien mit den Wurzelhaaren zusammengerollt. Die Bakterien durchdringen die Wurzelhaarzellen mit einem Infektionsfaden (en. infection thread), der durch das Wurzelhaar in die Hauptwurzel wächst. Dies führt dazu, dass sich die infizierten Zellen teilen und ein Knöllchen bilden. Die Bakterien finden sich dann wie bei den Rhizobiaceae in speziell eingerichteten Pflanzenzellen (Bacteroiden) innerhalb Vakuolenstruktur, die Symbiosom genannt wird.[15] Die Hyphomicrobiales/Rhizobiales-Bakterien können nun mit der Stickstofffixierung beginnen.
Es sind über 55 Gene bekannt, die mit der Knöllchenbildung (en. nodulation) in Verbindung stehen (Nod-Gene).[16] Das nodD ist für die Expression der anderen nod-Gene unerlässlich.[17] Es sind insgesamt zwei nodD-Gene bekannt: nodD1 und nodD2, aber nur nodD1 wird für eine erfolgreiche Knöllchenbildung benötigt.[16]
Stickstofffixierung
Bradyrhizobium und andere in den Knöllchen lebende Rhizobiales-Bakterien nehmen atmosphärischen Stickstoff (N2) auf und binden ihn in Ammoniak (NH3) oder Ammonium (NH4+). Die Pflanzen selbst können atmosphärischen Stickstoff nicht nutzen, sie müssen eine Verbindung des Elements verwenden. Nach der Photosynthese ist die Stickstofffixierung der wichtigste Prozess für das Wachstum und die Entwicklung von Pflanzen.[18] Der Gehalt an Ureid-Stickstoff in einer Pflanze korreliert mit der Menge des fixierten Stickstoffs, den die Pflanze aufnimmt.[19]
Wichtige bei Bradyrhizobium an der Stickstofffixierung beteiligte Gene sind nif und fix. Die nif-Gene sind den Genen von Klebsiella pneumoniae, einem freilebenden diazotrophen Bakterium der Klasse Gammaproteobacteria, in Funktion und Struktur sehr ähnlich, obwohl sie nicht wie Bradyrhizobium der Klasse Alphaproteobacteria angehören. Die für die symbiotische Stickstofffixierung wichtigen fix-Gene wurden zuerst bei den symbiotischen Bakterien der Ordnung Hyphomicrobiales (alias Rhizobiales) gefunden. Die nif- und fix-Gene befinden sich in mindestens zwei verschiedenen Clustern auf dem Bakterien-Chromosom. Cluster I enthält die meisten Stickstofffixierungsgene. Cluster II enthält drei fix-Gene, die in der Nähe der nod-Gene (s. o.) liegen.[20]
Artenvielfalt und Wirtsspezifität
Bradyrhizobium-Arten können entweder spezifische oder allgemeine Symbiosen bilden.[4] D. h. eine dieser Spezies ist möglicherweise nur in der Lage, eine einzige Leguminosenart als Wurzelknöllchen-Symbiont zu dienen, während andere Bradyrhizobium-Spezies in der Lage sein kann, mehrere verschiedene Leguminosenarten zu bedienen. Die ribosomale RNA (rRNA) ist in dieser Gruppe von Mikroben stark konserviert, was die Verwendung von Bradyrhizobium als Indikator für die Artenvielfalt äußerst schwierig macht. Stattdessen wurden DNA-DNA-Hybridisierungen verwendet, die eine größere Vielfalt zeigen. Allerdings werden nur wenige phänotypische Unterschiede festgestellt, so dass nicht viele Arten benannt werden konnten.[9]
Einige Bradyrhizobium-Stämme sind auch photosynthetisch aktiv, diese bilden dann oft Knöllchen in den Stängeln von semiaquatischen Leguminosen der Gattung Aeschynomene[Anm. 9] und wurden auch in den Knöllchenwurzeln des afrikanischen Wildreises Oryza breviligulata (syn. Oryza barthii) gefunden.[21][22]
Bedeutung
Körnerleguminosen werden auf etwa 1,5 Mio. km² Land pro Jahr angebaut.[18] Die Menge des jährlich fixierten Stickstoffs beträgt weltweit etwa 44–66 Mio. Tonnen, was fast der Hälfte des gesamten in der Landwirtschaft verwendeten Stickstoffs entspricht.[23] Es sind auch kommerzielle Bradyrhizobium-Inokulanzien erhältlich.
In der Molekularbiologie wurde Bradyrhizobium (wie auch Rhizobium) als Verunreinigung (Kontamination) von Reagenzien für DNA-Extraktionskits und Reinstwassersystemen identifiziert, was dazu führen kann, dass es fälschlicherweise in Mikrobiota- oder Metagenom-Datensätzen erscheint.
Bradyrhizobium wurde auch als Verunreinigung von Reagenzien für DNA-Extraktionskits und Reinstwassersystemen identifiziert, was dazu führen kann, dass es fälschlicherweise in Mikrobiota- oder Metagenom-Datensätzen erscheint.[3] Das Vorhandensein von stickstofffixierenden Bakterien als Verunreinigungen kann auf die Verwendung von Stickstoffgas bei der Reinstwasserproduktion zurückzuführen sein, um das mikrobielle Wachstum in Lagertanks zu hemmen.[24] Das Vorhandensein von stickstofffixierenden Bakterien als Verunreinigung kann auf die Verwendung von Stickstoffgas bei der Reinstwasserproduktion zurückzuführen sein, um das mikrobielle Wachstum in Lagertanks zu hemmen.[25]
Anmerkungen
- B. betae wurde aus tumorartigen Wurzeldeformationen an Zuckerrüben isoliert, ihr symbiotischer Status ist unbekannt.
- B. canariense bildet Wurzelknöllchen bei genistoiden Schmetterlingsblütlern ([en]), die auf den Kanarischen Inseln heimisch sind. Die Art wurde auch in Knöllchen von Lupinen und Serradella in Westaustralien und im südlichen Afrika gefunden. Zur Klade der genistoiden Schmetterlingsblütler siehe Genistoid clade, tolweb.org (Tree of Life Web Project).
- Bradyrhizobium centrosemae ist eine Fehlschreibung von Bradyrhizobium centrosematis.
- B. elkanii, B. diazoefficiens und B. liaoningense bilden Symbiosen mit Sojabohnen.
- B. japonicum erzeugt Wurzelknöllchen bei Sojabohnen, Augenbohnen, Mungobohnen und Siratro (Macroptilium atropurpureum). Zu Siratro siehe
- R. Hegnauer: Chemotaxonomie der Pflanzen, Band 33, Birkhäuser, Basel, 1994, S. 1–97, doi:10.1007/978-3-0348-8494-5_1, Online ISBN 978-3-0348-8494-5; hier: Bemerkung #335.
- Elisabeth Piras: Reis ist Leben, Wie Öko-Landbau und Fairer Handel zu Ernährungssouveränität führen, April 2011, auf: naturland.de
- B. neotropicale bildet Symbiosen mit dem Baum Centrolobium paraense aus dem Amazonas-Regenwald in Brasilien.
- B. yuanmingense bildet Wurzelknöllchen bei Buschklee (Lespedeza)
- Knöllchenbakterien oder Rhizobien im weiteren Sinne sind die eigentlichen Rhizobien (Knöllchenbakterien, Familie Rhizobiaceae), sowie verwandte Bakterien (aus einigen anderen Familien der gemeinsamen Ordnung Hyphomicrobiales, manchmal auch als Rhizobiales bezeichnet).
- Zu den Aeschynomene-Spezies gehören die Indische Schampflanze (Aeschynomene indica), die Shola-Pflanze (Aeschynomene aspera) und der Ambatch-Baum (Aeschynomene elaphroxylon).
Weblinks
- Just a Few Common Bacterial Groups Gobble Up the Majority of Carbon in Soil, auf: SciTechDaily vom 11. September 2021, Quelle: Lawrence Livermore National Laboratory; sowie Just a Few Common Bacteria Gobble Up Most of the Carbon in Soil, ebenda vom 23. Januar 2022, Quelle: U.S. Department of Energy.
Einzelnachweise
- M.-H. Ramirez-Bahena, R. Chahboune, A. Peix, E. Velazquez: Reclassification of Agromonas oligotrophica into the genus Bradyrhizobium as Bradyrhizobium oligotrophicum comb. nov. In: International Journal of Systematic and Evolutionary Microbiology. 63, Nr. Pt 3, 2012, S. 1013–1016. doi:10.1099/ijs.0.041897-0. PMID 22685107.
- E. R. Eaglesham, J. M. Ellis, W. R. Evans, D. E. Fleishman, M. Hungria, K. W. Hardy; P. M. Gresshoff, L. E. Koth, G. Stacey, W. E. Newton: The first photosynthetic N2-fixing Rhizobium: Characteristics. In: Nitrogen Fixation: Achievements and Objectives. Springer, Boston, MA 1990, ISBN 978-1-4684-6434-4, S. 805–811, doi:10.1007/978-1-4684-6432-0_69.
- David VanInsberghe, Kendra Maas, Erick Cardenas, Cameron Strachan, Steven Hallam, William Mohn: Non-symbiotic Bradyrhizobium ecotypes dominate North American forest soils. In: The ISME Journal. 9, Nr. 11, 2015, S. 2435–2441. doi:10.1038/ismej.2015.54. PMID 25909973. PMC 4611507 (freier Volltext).
- P. Somasegaran: Handbook for rhizobia: Methods in legume–rhizobium technology. Springer-Verlag, New York, 1994, ISBN 978-0-387-94134-9, S. 1–6, 167.
- King Gary: Molecular and culture-based analyses of aerobic carbon monoxide oxidizer diversity. In: Applied and Environmental Microbiology. 69, Nr. 12, 2003, S. 7257–7265. doi:10.1128/aem.69.12.7257-7265.2003. PMID 14660374. PMC 309980 (freier Volltext).
- LPSN: Genus Bradyrhizobium. Abgerufen am 23. Mai 2021. List of Prokaryotic names with Standing in Nomenclature
- NCBI: Bradyrhizobium Jordan 1982 (genus, syn. Agromonas Ohta and Hattori 1985); graphisch: Bradyrhizobium, auf: Lifemap, NCBI Version.
- M. S. Klepa, L. G. Ferraz Helene, G. O'Hara, M. Hungria: Bradyrhizobium agreste sp. nov., Bradyrhizobium glycinis sp. nov. and Bradyrhizobium diversitatis sp. nov., isolated from a biodiversity hotspot of the genus Glycine in Western Australia. In: Int J Syst Evol Microbiol. 2021. doi:10.1099/ijsem.0.004742. PMID 33709900.
- Raul Rivas, Miet Martens, Philippe De Lajudie, Anne Willems: Multilocus sequence analysis of the genus Bradyrhizobium. In: Systematic and Applied Microbiology. 32, Nr. 2, 2009, S. 101–110. doi:10.1016/j.syapm.2008.12.005. PMID 19201125.
- M. Kalita, W. Małek: Genista tinctoria microsymbionts from Poland are new members of Bradyrhizobium japonicum bv. genistearum. In: Systematic & Applied Microbiology. 33, Nr. 5, 2010, S. 252–259. doi:10.1016/j.syapm.2010.03.005. PMID 20452160.
- Jerri E. Zilli, Alexandre C. Baraúna, Krisle da Silva, Sofie E. De Meyer, Eliane N. C. Farias, Paulo E. Kaminski, Ismaele B. da Costa, Julie K. Ardley, Anne Willems, Natália N. Camacho, Fernanda dos S. Dourado, Graham O’Hara: Bradyrhizobium neotropicale sp. nov., isolated from effective nodules of Centrolobium paraense. In: International Journal of Systematic and Evolutionary Microbiology. 64, Nr. Pt 12, 9. September 2014, S. 3950–3957. doi:10.1099/ijs.0.065458-0. PMID 25205796.
- NCBI: Bradyrhizobium sp. USDA 4362 (species)
- NCBI: Photorhizobium (genus), und Photorhizobium (list/token)
- Anton Hördt, Marina García López, Jan P. Meier-Kolthoff, Marcel Schleuning, Lisa-Maria Weinhold, Brian J. Tindall, Sabine Gronow, Nikos C. Kyrpides, Tanja Woyke, Markus Göker: Analysis of 1,000+ Type-Strain Genomes Substantially Improves Taxonomic Classification of Alphaproteobacteria. In: Frontiers in Microbiology. 11, 7. April 2020, S. 468. doi:10.3389/fmicb.2020.00468. PMID 32373076. PMC 7179689 (freier Volltext).
- A. F. Haag et al.: Molecular insights into bacteroid development during Rhizobium-legume symbiosis. In: FEMS Microbiology Reviews. 37, Nr. 3, Mai 2013, S. 364–383. doi:10.1111/1574-6976.12003. PMID 22998605.
- Gary Stacey: Bradyrhizobium japonicum nodulation genetics. In: FEMS Microbiology Letters. 127, Nr. 1–2, 1995, S. 1–9. doi:10.1111/j.1574-6968.1995.tb07441.x. PMID 7737469.
- Gary Stacey, J. Sanjuan, S. Luka, T. Dockendorff, R. W. Carlson: Signal exchange in the Bradyrhizobium–soybean symbiosis. In: Soil Biology and Biochemistry. 27, Nr. 4–5, 1995, S. 473–483. doi:10.1016/0038-0717(95)98622-U.
- G. Caetanoanolles: Molecular dissection and improvement of the nodule symbiosis in legumes. In: Field Crops Research. 53, Nr. 1–3, 1997, S. 47–68. doi:10.1016/S0378-4290(97)00022-1.
- P. van Berkum, C. Sloger, D. F. Weber, P. B. Cregan, H. H. Keyser: Relationship between Ureide N and N2 Fixation, Aboveground N Accumulation, Acetylene Reduction, and Nodule Mass in Greenhouse and Field Studies with Glycine max (L.) Merr. In: Plant Physiol.. 77, Nr. 1, 1985, S. 53–58. doi:10.1104/pp.77.1.53. PMID 16664027. PMC 1064455 (freier Volltext).
- Hauke Hennecke: Nitrogen fixation genes involved in the Bradyrhizobium japonicum–soybean symbiosis. In: FEBS Letters. 268, Nr. 2, 1. August 1990, S. 422–426. doi:10.1016/0014-5793(90)81297-2. PMID 2200721.
- Clémence Chaintreuil, Eric Giraud, Yves Prin, Jean Lorquin, Amadou Bâ, Monique Gillis, Philippe de Lajudie, Bernard Dreyfus: Photosynthetic Bradyrhizobia Are Natural Endophytes of the African Wild Rice Oryza breviligulata. In: Applied and Environmental Microbiology. 66, Nr. 12, Dezember 2000, S. 5437-5447. doi:10.1128/AEM.66.12.5437-5447.2000.
- Der Afrikanische Wildreis: Oryza barthii, auf: flora-obscura.de
- O. Alberton, G. Kaschuk, M. Hungria: Sampling effects on the assessment of genetic diversity of rhizobia associated with soybean and common bean. In: Soil Biology and Biochemistry. 38, Nr. 6, 2006, S. 1298–1307. doi:10.1016/j.soilbio.2005.08.018.
- Susannah J. Salter, Michael J. Cox, Elena M. Turek, Szymon T. Calus, William O. Cookson, Miriam F. Moffatt, Paul Turner, Julian Parkhill, Nicholas J. Loman, Alan W. Walker: Reagent and laboratory contamination can critically impact sequence-based microbiome analyses, in: BMC Biology, Band 12, Nr. 87, 12. November 2014, doi:10.1186/s12915-014-0087-z. PrePrint doi:10.1101/007187.
- Leonid A. Kulakov, Morven B. McAlister, Kimberly L. Ogden, Michael J. Larkin, John F. O'Hanlon: Analysis of bacteria contaminating ultrapure water in industrial systems. In: Applied and Environmental Microbiology. 68, Nr. 4, April 2002, S. 1548–1555. doi:10.1128/AEM.68.4.1548-1555.2002. PMID 11916667. PMC 123900 (freier Volltext).