Single Guide RNA
Single Guide RNA (sgRNA) ist eine künstliche RNA, die in der CRISPR/Cas-Methode, der CRISPRi oder der CRISPRa in Kombination mit Cas9 oder Cas12b verwendet wird.
Eigenschaften
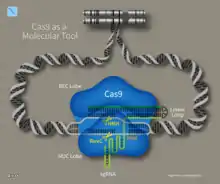
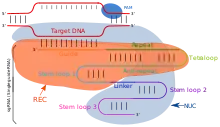
Die sgRNA bildet eine Sekundärstruktur, die als R-loop bezeichnet wird.[1] Sie kann in Bakterien,[2][3][4] Hefen,[5] Fruchtfliegen,[6] Zebrafischen[7] und Mäusen[8] verwendet werden. Daraufhin wird sie von Cas-Proteinen des Typs I und II gebunden.[1] Natürlicherweise bindet Cas9 zwei RNA, die crRNA und die tracrRNA, während bei der Methode nur eine aus Sequenzen der beiden RNA bestehende sgRNA verwendet wird.[9] Dadurch muss für die CRISPR/Cas-Methode nur eine RNA kloniert werden. Eine sgRNA besteht aus den 20 Nukleotiden strangaufwärts (in 5'-Richtung) von einem Protospacer Adjacent Motif (PAM) der zu schneidenden Ziel-DNA und einem Teil der tracrRNA. Bevorzugt befindet sich am 5'-Ende der 20 Nukleotide (Position 1) ein Guaninnukleotid (GC-clamp) und vier Nukleotide vor dem PAM (Position 17) ein Adenin- oder Thyminnukleotid.[10] Programme zur Identifikation von 20 Nukleotiden vor einem PAM bzw. zum Entwerfen einer sgRNA sind beispielsweise CHOPCHOP,[11] CasOFFinder,[12] FlyCRISPR,[13] CRISPR-ERA,[14] SgRNA Designer,[15] CRISPOR,[16] E-CRISP[17] und CRISPRdirect.[18]
Meistens werden zum Einschleusen der sgRNA in eukaryotische Zellen virale Vektoren transduziert oder Plasmide transfiziert. Bei Verwendung zur Gentherapie in Eukaryoten wird ein eukaryotischer Promotor verwendet. Im Falle einer Einschleusung des Cas-Proteins und der sgRNA in eine Zelle wird die sgRNA vorher durch In-vitro-Transkription aus einem Vektor beispielsweise mit einem T7-Promotor erzeugt.
Weblinks
- How to Design Your gRNA for CRISPR Genome Editing, abgerufen am 29. Januar 2019.
Einzelnachweise
- F. Jiang, D. W. Taylor, J. S. Chen, J. E. Kornfeld, K. Zhou, A. J. Thompson, E. Nogales, J. A. Doudna: Structures of a CRISPR-Cas9 R-loop complex primed for DNA cleavage. In: Science. Band 351, Nummer 6275, Februar 2016, S. 867–871, doi:10.1126/science.aad8282, PMID 26841432, PMC 5111852 (freier Volltext).
- W. Jiang, D. Bikard, D. Cox, F. Zhang, L. A. Marraffini: RNA-guided editing of bacterial genomes using CRISPR-Cas systems. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 233–239, doi:10.1038/nbt.2508, PMID 23360965, PMC 3748948 (freier Volltext).
- J. M. Peters, A. Colavin, H. Shi, T. L. Czarny, M. H. Larson, S. Wong, J. S. Hawkins, C. H. Lu, B. M. Koo, E. Marta, A. L. Shiver, E. H. Whitehead, J. S. Weissman, E. D. Brown, L. S. Qi, K. C. Huang, C. A. Gross: A Comprehensive, CRISPR-based Functional Analysis of Essential Genes in Bacteria. In: Cell. Band 165, Nummer 6, Juni 2016, S. 1493–1506, doi:10.1016/j.cell.2016.05.003, PMID 27238023, PMC 4894308 (freier Volltext).
- X. T. Li, Y. Jun, M. J. Erickstad, S. D. Brown, A. Parks, D. L. Court, S. Jun: tCRISPRi: tunable and reversible, one-step control of gene expression. In: Scientific Reports. Band 6, 12 2016, S. 39076, doi:10.1038/srep39076, PMID 27996021, PMC 5171832 (freier Volltext).
- J. E. DiCarlo, J. E. Norville, P. Mali, X. Rios, J. Aach, G. M. Church: Genome engineering in Saccharomyces cerevisiae using CRISPR-Cas systems. In: Nucleic acids research. Band 41, Nummer 7, April 2013, S. 4336–4343, doi:10.1093/nar/gkt135, PMID 23460208, PMC 3627607 (freier Volltext).
- G. W. Thickbroom, F. L. Mastaglia: Cerebral events preceding self-paced and visually triggered saccades. A study of presaccadic potentials. In: Electroencephalography and clinical neurophysiology. Band 62, Nummer 4, Juli 1985, S. 277–289, doi:10.1016/0168-5597(85)90005-x, PMID 2408874.
- W. Y. Hwang, Y. Fu, D. Reyon, M. L. Maeder, S. Q. Tsai, J. D. Sander, R. T. Peterson, J. R. Yeh, J. K. Joung: Efficient genome editing in zebrafish using a CRISPR-Cas system. In: Nature Biotechnology. Band 31, Nummer 3, März 2013, S. 227–229, doi:10.1038/nbt.2501, PMID 23360964, PMC 3686313 (freier Volltext).
- H. Wang, H. Yang, C. S. Shivalila, M. M. Dawlaty, A. W. Cheng, F. Zhang, R. Jaenisch: One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. In: Cell. Band 153, Nummer 4, Mai 2013, S. 910–918, doi:10.1016/j.cell.2013.04.025, PMID 23643243, PMC 3969854 (freier Volltext).
- M. Jinek, K. Chylinski, I. Fonfara, M. Hauer, J. A. Doudna, E. Charpentier: A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. In: Science. Band 337, Nummer 6096, August 2012, S. 816–821, doi:10.1126/science.1225829, PMID 22745249, PMC 6286148 (freier Volltext).
- How to design sgRNA sequences. In: takarabio.com. Abgerufen am 2. Februar 2019.
- CHOPCHOP. In: chopchop.cbu.uib.no. Abgerufen am 30. Januar 2019 (englisch).
- CRISPR RGEN Tools. In: rgenome.net. Abgerufen am 30. Januar 2019 (englisch).
- C. Dustin Rubinstein, Ed O'Connor-Giles, Kate O': CRISPR Optimal Target Finder. In: tools.flycrispr.molbio.wisc.edu. Abgerufen am 30. Januar 2019 (englisch).
- CRISPR-ERA. In: crispr-era.stanford.edu. Abgerufen am 30. Januar 2019.
- sgRNA Designer: CRISPRko. In: portals.broadinstitute.org. Abgerufen am 30. Januar 2019 (englisch).
- CRISPOR. In: crispor.tefor.net. Abgerufen am 30. Januar 2019.
- E-CRISP Design. In: e-crisp.org. 1. Januar 2013, abgerufen am 30. Januar 2019 (enc).
- S. E. Mohr, Y. Hu, B. Ewen-Campen, B. E. Housden, R. Viswanatha, N. Perrimon: CRISPR guide RNA design for research applications. In: The FEBS journal. Band 283, Nummer 17, 09 2016, S. 3232–3238, doi:10.1111/febs.13777, PMID 27276584, PMC 5014588 (freier Volltext).