Eukaryotischer Promotor
Der eukaryotische Promotor ist ein Teil der Erbinformation bei Lebewesen mit Zellkern (Eukaryoten). Es handelt sich um denjenigen Abschnitt der DNA, der direkt vor dem Transkriptionsstart des jeweiligen Gens liegt und Information darüber enthält, wann und in welchem Zelltyp das jeweilige Gen exprimiert werden soll. Im Vergleich zum Promotor anderer Lebewesen ist der eukaryotische Promotor wesentlich komplizierter.
Struktur
Der eukaryotische Promotor ist in drei Abschnitte eingeteilt: am nächsten beim Transkriptionsstart des Gens liegt der Kernpromotor, es folgt (gegen die Leserichtung) der proximale Promotor und weiter entfernt schließlich die Enhancer sowie mögliche Locus-Kontrollregionen. Nicht bei allen eukaryotischen Genen findet man alle Abschnitte.[2][3]
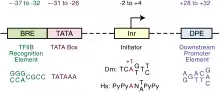
Kernpromotor
Der Kernpromotor erstreckt sich maximal zwischen Position −37 bis +32, wenn +1 der Transkriptionsstart des Gens festgelegt wird. Im Kernpromotor finden sich häufig eine TATA-Box, und oft ein Downstream Promoter Element. Manchmal gibt es zwei TATA-Boxen oder zur TATA-Box ein BRE-Element.
Proximaler Promotor
Der proximale Promotor ist zwischen Position −50 und −200 lokalisiert. Er kann eine CAAT-Box, eine GC-Box oder eine CpG-Insel enthalten. Im proximalen Promotor befinden sich außerdem die Bindungssequenzen für Transkriptionsfaktoren, die ihrerseits mit Coaktivatoren oder Corepressoren komplexiert sind.
Enhancer
Enhancer- bzw. Silencer-Regionen sind von den meisten Genen höherer Eukaryoten bekannt. Sie gehören wie die Promotoren zu den cis-Elementen. Aufgrund des Supercoilings eukaryotischer DNA müssen Enhancer nicht nahe am Transkriptions-Beginn gelegen sein. Entfernungen bis zu 100 kbp sind bekannt. Enhancer enthalten weitere Bindungsstellen für Transkriptionsfaktoren.
Locus-Kontrollregionen (LCR)
LCR liegen viele Tausend Basenpaare vom Transkriptionsstart entfernt und bestehen aus DNase I-sensiblen Regionen. Auch hier spielt das Supercoiling eine Rolle. Es sind 20 Genfamilien beim Menschen bekannt, deren Expression über LCR kontrolliert wird.
Einzelnachweise
- Jennifer E.F. Butler, James T. Kadonaga: The RNA polymerase II core promoter: a key component in the regulation of gene expression. In: Genes & Development. 16, Nr. 20, 15. Oktober 2002, S. 2583–2592. doi:10.1101/gad.1026202. PMID 12381658.
- Jochen Graw, Wolfgang Hennig: Genetik. 4. vollständig überarbeitete Auflage. Springer-Verlag, Berlin u. a. 2006, ISBN 3-540-24096-9, S. 320–329.
- reactome: Generic Transcription Pathway doi:10.3180/REACT_12627.1