Salmonella Typhimurium
Salmonella Typhimurium (eigentlich: Salmonella enterica subsp. enterica ser. Typhimurium oder auch Salmonella enterica subsp. I ser. Typhimurium) zählt zu den Serovaren der Art Salmonella enterica. Es ist ein begeißeltes und daher bewegliches, gramnegatives Bakterium[1] und beinhaltet, wie alle Serovare der Subspezies I, das O-Antigen, das Bestandteil des Lipopolysaccharid-Komplexes der Bakterienzellwand ist. Salmonella Typhimurium ist eines der hauptverursachenden Bakterien menschlicher Gastroenteritis. Salmonella Typhimurium befällt, im Gegensatz zu seinem nahen Verwandten, Salmonella Typhi, nicht nur Menschen, sondern auch viele andere Säugetiere (lateinisch: typhi murium = des Typhus der Mäuse).
| Salmonella Typhimurium | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|
Kolonien von Salmonella Typhimurium auf Hektoen-Enteroagar | ||||||||||||
| Systematik | ||||||||||||
| ||||||||||||
| Wissenschaftlicher Name | ||||||||||||
| S. enterica subsp. enterica ser. Typhimurium | ||||||||||||
| (ex Kauffmann and Edwards 1952) Le Minor and Popoff 1987 |
Vorkommen
Salmonella Typhimurium wurde in den USA, dem Vereinigten Königreich, Kanada, Deutschland, Frankreich, Österreich und Dänemark gefunden[2] und gehört zu den beiden in Deutschland bei menschlichen Infektionen am häufigsten nachgewiesenen Serovaren.[3] Das Bakterium besitzt einen geringeren Risikostatus für die Bevölkerung als Salmonella Typhi, allerdings kommen mehr Menschen durch nicht-typhoide Salmonellose, welche durch Salmonella Typhimurium hervorgerufen wird, zu Tode. Besonders betroffen sind Kleinkinder und ältere Menschen.[4] Salmonella Typhimurium kann sich im Temperaturbereich von 5–45 °C vermehren. Salmonellen sind widerstandsfähig gegen oxidativen Stress, dadurch können sie der Phagozytose entgehen.[5] Temperaturen über 70 °C und gebräuchliche Desinfektionsmittel töten Salmonellen allerdings sehr schnell ab.
Aufbau und Genom
LT2 ist der Stamm von Salmonella Typhimurium, der hauptsächlich für molekularbiologische und zelluläre Untersuchungen verwendet wird. Sie verfügen über eine stark abgeschwächte Virulenz.[6]
Das Genom von Salmonella Typhimurium Stamm LT2 gliedert sich im Wesentlichen in zwei Bestandteile: das Chromosom und das Plasmid pSLT. Das Chromosom (Größe: 4.857.432 bp) besitzt insgesamt 4.489 codierende Sequenzen, darunter 39 Pseudogene. Darüber hinaus enthält es 85 tRNA, sieben tRNA-Cluster, 11 strukturelle RNA und ein tRNA-Pseudogen. Die Pseudogene sind für Maltoseregulation und Trehalose-Metabolismus zuständig. Auf dem Chromosom sind 11 Operons (stc, bcf, fim, lpf, saf, stb, std, stf, sth, sti und stj) lokalisiert. Sie codieren Fimbrien auf der Zelloberfläche, welche den Kontakt zum Darmepithel herstellen.
Antibiotikaresistenzgene gegen Ampicillin, Streptomycin, Chloramphenicol, Sulfonamides und Tetracycline sowie Resistenzen gegen Trimethoprim und Ciprofloxacin sind im Chromosom verschlüsselt.[2]
Der GC-Gehalt beträgt im Chromosom und im Plasmid 53 %.
Das Plasmid pSLT (Größe 93.939bp) enthält dagegen nur eine strukturelle RNA und 108 codierende Sequenzen, darunter sechs Pseudogene. Auch das Plasmid besitzt ein Operon (pef) zur Bildung von Fimbrien. Salmonella Typhimurium beinhaltet vier Prophagen (Gifsy-1, Gifsy-2, Fels-1 und Fels-2), die eine Rolle bei den Infektionen spielen.
Entdeckung
Salmonella Typhimurium Stamm LT2 wurde in den 1940er Jahren isoliert und in Studien für phagenübertragende Transduktion verwendet. Dabei wurden abgeschwächte Bakterien der Gattung als Schluckimpfung verabreicht, um Antigene von anderen Pathogenen zu zeigen und Proteine zu Tumoren zu transportieren.
Die Sequenzierung des Salmonella Typhimurium LT2-Genoms erfolgte 2001 im Rahmen eines Experiments.[7] Es wurden zwei Methoden zur Sequenzierung verwendet: Whole Genom Shotgun Sampling und Sampling von gelgereinigten Restriktionsfragmente.
Hierbei ermittelte man zunächst die Homologie von Salmonella Typhimurium zu anderen Darmbakterien, indem man es mit den vollständigen Genomen von drei verwandten Bakterienstämmen (Escherichia coli K12 und O157:H7 und S. typhi) verglich. Dabei wurden sie auf einem Mikroarray, auf dem Gene von Salmonella Typhimurium LT2 aufgetragen waren, hybridisiert.
Homologie
Vergleiche mit den vier oben genannten Bakteriengenomen zeigte, dass sie in einem hohen Prozentsatz kollinear sind. Sie wiesen jedoch in den inversen Replikationstermini hohe Unterschiede auf.
Mindestens 55 % aller Pseudogene von Salmonella Typhimurium sind homolog zu den Genomen einiger Enterobakterien (Salmonella Typhi, Salmonella Paratyphi A und B, S. enterica subsp. arizonae, S. bongori, E. coli K12 und O157:H7 und K. pneumoniae), was 2,5 Mb des Salmonella Typhimurium LT2 Genoms entspricht. Von den 108 codierenden Sequenzen und Pseudogenen im Plasmid des Salmonella Typhimurium LT2 waren nur drei homolog zu den Enterobakterien Salmonella Typhi und Salmonella Paratyphi A.
Lateraler Gentransfer trat häufig auf: 11 % der S. typhi-Gene und 29 % der E. coli K12-Gene wanderten in das Salmonella-Typhimurium-LT2-Genom. Die Prophagen Gifsy-1 und -2, Fels-1 und -2 traten nur in Salmonella Typhimurium auf und sind in den anderen acht Enterobakterien trotz Homologie nicht vorhanden.
Mindestens eine der codierenden Sequenzen in Salmonella Typhimurium gleicht einer in den Enterobakterien Salmonella Typhi, Salmonella Paratyphi A/B. Weiterhin gibt es 121 codierende Sequenzen, die es in keinem der anderen Enterobakterien gibt.
Krankheitsbild
Die Infektion mit Salmonella Typhimurium entsteht durch verunreinigte Nahrung, hiermit ist sowohl die Verköstigung infizierter Nahrung (z. B. befallenes Obst oder verseuchte Erdnussbutter) als auch der Verzehr von Produkten, die von infizierten Tieren (z. B. Fleischprodukte) stammen, gemeint. Gut gegartes Fleisch minimiert das Infektionsrisiko, da alle Salmonellen hitzeempfindlich sind.[8][9]
Salmonella Typhimurium kann über den Kot erkrankter Tiere oder Menschen übertragen werden. Eine große Rolle spielt in diesem Zusammenhang die Hygiene. Mangelhaftes Händewaschen nach dem Entfernen von Haustierkot, Toilettengängen oder Füttern eines erkrankten Haustieres sorgt für ein erhöhtes Infektionsrisiko.[10]
Das Darmbakterium kann die Barriere des Magens durchdringen, da der pH-Wert des Magens abhängig ist von der aufgenommenen Nahrung und die Magensäure so variabel für Bakterien ist. Salmonella Typhimurium dringt in die Epithelzellen des Dünndarms ein und wird zum darunterliegenden Bindegewebe transportiert, wo es sich vermehrt. Die Symptome sind Durchfall, Fieber und Unterleibschmerzen, die 12–72 Stunden nach der Infektion beginnen und eine Woche andauern.[11][12][13]
Diagnostik
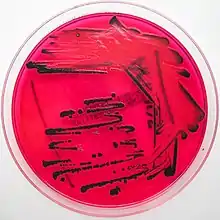
Salmonella Typhimurium kann in aller Regel ohne Schwierigkeiten kulturell aus Stuhlproben nachgewiesen werden. Die Stuhlprobe wird dabei im Normalfall über Nacht in einer Anreicherungsflüssigkeit (z. B. Selenitbouillon) vorbebrütet und anschließend auf einen selektiven Differenzierungsnährboden ausgestrichen (z. B. XLD-Agar). Verdächtige Kolonien werden dann am nächsten Tag auf einen Schrägagar nach Kligler überimpft, von dem zum Teil auch die serologische Differenzierung vorgenommen wird.
Auf dem Kligler-Agar zeigt Salmonella Typhimurium (wie andere enteritische Salmonellen auch) folgende Eigenschaften: Schrägschicht rot (Laktose negativ, daher auch auf MacConkey-Agar farblos), Hochschicht gelb (Glukose positiv), H2S positiv (schwarzer Niederschlag in der Hochschicht, die die Gelbfärbung vollständig überdecken kann), Gasbildung sichtbar. Falls keine vollständige biochemische oder massenspektrometrische Differenzierung durchgeführt wird, sollten auch noch die positive Katalase- und die negative Urease-Reaktion nachgewiesen werden, um Verwechslungen mit Bakterien der Gattungen Citrobacter und Proteus sicher auszuschließen. Hier können auch die positive Lysin-Decarboxylase und die negative Phenylalanin-Desaminase als biochemische Schlüsselreaktionen noch weiterhelfen.
In der immer erforderlichen serologischen Differenzierung zeigt Salmonella Typhimurium folgende zentrale Eigenschaften: Agglutination mit NaCl-Lösung negativ, mit polyvalentem Salmonellen-Antiserum („Omni“-Serum) positiv, mit O:4-Serum positiv (dies zeigt die Zugehörigkeit zur Gruppe O:4 bzw. Gruppe B nach alter Nomenklatur, siehe auch Kauffmann-White-Schema), mit dem Serum i der ersten H-Phase positiv sowie mit den Seren 1 und 2 der zweiten H-Phase positiv (für letztere Agglutinationsreaktion ist teilweise die Unterdrückung der ersten H-Phase mittels eines Spezialagars („Schwärmagar“) notwendig).[14][15]
Impfungen
Salmonella Typhimurium wird auch heute gern in abgeschwächter Variante in der Laborarbeit eingesetzt, da es in Mäusen dieselben Fiebersymptome hervorruft, wie im Menschen.[16]
Seit 2002 gibt es einen Typhimurium-Lebendimpfstoff, der unter dem Namen „SALMOPORC“ vertrieben und Saugferkeln gespritzt wird.
Literatur
- Michael McClelland, Kenneth E. Sanderson, John Spieth, Sandra W. Clifton, Phil Latreille, Laura Courtney, Steffen Porwollik, Johar Ali, Mike Dante, Feiyu Du, Shunfang Hou, Dan Layman, Shawn Leonard, Christine Nguyen, Kelsi Scott, Andrea Holmes, Neenu Grewal, Elizabeth Mulvaney, Ellen Ryan, Hui Sun, Liliana Florea, Webb Miller, Tamberlyn Stoneking, Michael Nhan, Robert Waterston, Richard K. Wilson: Complete genome sequence of Salmonella enterica serovar Typhimurium LT2. In: Nature. Band 413, Nr. 6858, September 2001, S. 852–856, doi:10.1038/35101614.
Einzelnachweise
- Pascal Le Gros Bin Beck Songhet: Salmonella Typhimurium diarrhea as a result of pathogen invasion and immune defence. Zürich 2010, doi:10.3929/ethz-a-006323804 (Dissertation, ETH Zürich).
- Salmonella Typhimurium DT104 Situation Assessment (PDF; 103 kB), Dezember 1997.
- Salmonellose (Salmonellen-Gastroenteritis). RKI-Ratgeber für Ärzte. Robert Koch-Institut, 16. August 2011, abgerufen am 19. Februar 2013.
- innovations-report.com: Salmonella bacteria sequenced.
- T. A. Halsey, A. Vazquez-Torres, D. J. Gravdahl, F. C. Fang, S. J. Libby: The ferritin-like Dps protein is required for Salmonella enterica serovar Typhimurium oxidative stress resistance and virulence. In: Infection and Immunity. Band 72, Nummer 2, Februar 2004, S. 1155–1158. PMID 14742565. PMC 321587 (freier Volltext).
- Stellungnahme der ZKBS: Salmonella Typhimurium LT2 aroA, galE oder cya, crp (1996)
- Nature: Complete genome sequence of: Salmonella enterica: serovar Typhimurium LT2
- CDC: Salmonella Typhimurium Infections Linked to Peanut Butter, 29. April 2009.
- BfR: Neue Studien zeigen: Salmonellen auch bei Mastschweinen und Mastputen ein Problem
- CDC: Q and A Related to the Salmonella Typhimurium Outbreak and Pets
- Bacteria Genomes – SALMONELLA TYPHIMURIUM (Memento vom 1. Juli 2012 im Internet Archive)
- Bundesamt für Verbraucherschutz und Lebensmittelsicherheit: Stellungnahme der ZKBS zur Einstufung von Salmonella Typhimurium LT2-Stämmen und von Salmonella Typhimurium-Stämmen mit stabilen Mutationen in den Genen aroA, galE oder cya und crp als Empfängerorganismen bei gentechnischen Arbeiten, Januar 1996.
- Matthias Eddicks: Überprüfung der Verträglichkeit des Salmonella Typhimurium – Lebendimpfstoffes Salmoporc® bei oraler Anwendung für drei Tage alte Saugferkel unter Berücksichtigung der Ausscheidung, Persistenz und Immunogenität des Impfstamms. (PDF; 461 kB) Dissertation, LMU München: Tierärztliche Fakultät 2006. urn:nbn:de:bvb:19-63308
- Helmut Tschäpe, Rolf Reissbrodt und Rita Prager: Salmonella spp. In: Birgid Neumeister, Heinrich K. Geiss, Rüdiger W. Braun und Peter Kimmig (Hrsg.): Mikrobiologische Diagnostik. 2. Auflage. Georg Thieme Verlag, Stuttgart – New York 2009, ISBN 978-3-13-743602-7, S. 450–454.
- Patrick A. D. Grimont und François-Xavier Weill: Antigenic Formulae of the Salmonella Serovars. (Nicht mehr online verfügbar.) WHO Collaboration Centre for Reference and Research on Salmonella, 2007, S. 19, archiviert vom Original am 1. Juli 2013; abgerufen am 20. Februar 2013 (englisch). Info: Der Archivlink wurde automatisch eingesetzt und noch nicht geprüft. Bitte prüfe Original- und Archivlink gemäß Anleitung und entferne dann diesen Hinweis.
- innovations-report.com: Salmonella bacteria sequenced