Konnektom
Als Konnektom (englisch connectome) bezeichnet man die Gesamtheit der Verbindungen im Nervensystem eines Lebewesens. Seinem Studium auf verschiedenen Skalen widmet sich die Konnektomik (englisch connectomics), ein Teilgebiet der Neurowissenschaften.
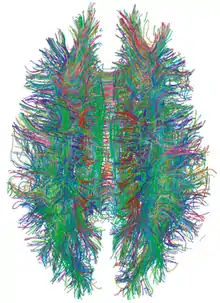
Da die Verbindungen einer Nervenzelle bei der Bestimmung ihrer Funktion eine zentrale Rolle spielen, ist ihre Untersuchung ein traditioneller Gegenstand biologischer Forschung. Das Begriffspaar „Konnektom“ und „Konnektomik“ gibt es jedoch erst seit 2005. Zu seiner Etablierung trug das im September 2010 bewilligte Human Connectome Project bei, in dessen Rahmen die National Institutes of Health die Erforschung des menschlichen Konnektoms mit insgesamt knapp 40 Millionen US-Dollar fördern.
Prägung des Begriffs
Der Begriff „Konnektom“ geht auf die Neurowissenschaftler Olaf Sporns (Indiana University) und Patric Hagmann (École polytechnique fédérale de Lausanne) zurück, die ihn im Jahr 2005 unabhängig voneinander vorgeschlagen hatten.[1][2]
Durch bewusste Anlehnung an die bestehenden Begriffe Genom und Proteom, die die Gesamtheit der Erbinformation beziehungsweise der Proteine in einem Lebewesen bezeichnen, soll der Name „Konnektom“ zum Ausdruck bringen, dass die einzelnen Verbindungen nur in ihrer gegenseitigen Beziehung zueinander verstanden werden können, ähnlich wie einzelne Gene oder Proteine im Organismus miteinander wechselwirken.[2] Ähnlich wie das Proteom ist das Konnektom nicht statisch, sondern aufgrund der neuronalen Plastizität ständigen Veränderungen unterworfen.
Für die synaptischen Verbindungen zwischen den Neuronen wird der aus der Elektrotechnik entlehnte Begriff Verdrahtung genutzt.
Das Konnektom auf verschiedenen Skalen
Die Konnektom-Forschung konzentriert sich auf die Netzwerkeigenschaften des Nervensystems. Das zu untersuchende Netzwerk wird hierbei meist durch einen Graphen im Sinne der Graphentheorie dargestellt, das heißt als abstrakte Menge so genannter Knoten, die durch Kanten verbunden sind. Welche konkrete Bedeutung die Knoten und Kanten haben, hängt von der Größenordnung ab, auf der das Nervensystem betrachtet wird. Die möglichen Größenordnungen werden grob in Mikroskala, Mesoskala und Makroskala unterteilt.
Mikroskala: Das Konnektom als Netzwerk von Nervenzellen
Auf feinster Ebene, der Mikroskala, besteht ein Nervensystem aus Neuronen, die durch Synapsen miteinander verbunden sind. Eine Untersuchung des Nervengewebes auf dieser Skala erfordert seine Darstellung mit einem Auflösungsvermögen im Mikrometer-Bereich. Die Knoten im Netzwerkgraphen entsprechen in diesem Fall den einzelnen Neuronen.
Eine vollständige Rekonstruktion des Konnektoms auf Mikroebene ist 1986 erstmals bei den Hermaphroditen des Fadenwurms Caenorhabditis elegans gelungen. Ihr Nervensystem besteht aus 302 Neuronen, die rund 5000 Synapsen, 2000 neuromuskuläre Endplatten sowie 600 Gap Junctions bilden. Es wurde 1986 auf Grundlage einer elektronenmikroskopischen Untersuchung serieller Schnitte durch ein Team um den Biologen John White beschrieben.[3]
Eine ähnlich umfassende Untersuchung komplexerer Organismen ist beim derzeitigen Stand der Technik nicht möglich, da die Anzahl ihrer Neurone typischerweise in die Milliarden geht. Aufgrund von Hochrechnungen ist davon auszugehen, dass beispielsweise das menschliche Gehirn rund 10 Milliarden Neurone enthält, die rund 100 Billionen Synapsen bilden;[4] im Vergleich dazu umfasst das menschliche Genom nur wenig mehr als 3 Milliarden Basenpaare. In diesem Maßstab ist es nicht mehr möglich, alle Axone in aufeinander folgenden elektromikroskopischen Schnittbildern manuell nachzuverfolgen. Daher werden für diese Aufgabe spezialisierte Methoden des maschinellen Sehens entwickelt, deren Qualität bislang jedoch noch nicht ganz die einer manuellen Segmentierung erreicht.[5] Zudem stellt bereits die Speicherung und Verarbeitung der anfallenden Datenmengen eine technische Herausforderung dar.
Mesoskala: Kortikale Säulen und Schichten
Die Großhirnrinde ist in kortikalen Säulen organisiert, Verbänden von einigen hundert oder tausend Neuronen, mit einem Gesamtdurchmesser von etwa 80 Mikrometern. Sie zeichnen sich durch gemeinsame afferente Nervenverbindungen aus, sind untereinander besonders stark verbunden und insbesondere in den primären sensorischen Arealen deutlich ausgeprägt. Sie bilden eine grundlegende Verarbeitungseinheit des Kortex.
Neben dieser vertikalen Organisation lassen sich histologisch darüber hinaus horizontale Schichten unterscheiden; aufgrund deren Anzahl unterteilt man die Großhirnrinde in den Isocortex (sechs Schichten) und den Allocortex (drei bis fünf Schichten). Während die Hirnforschung im Laufe ihrer jüngeren Geschichte wesentliche Fortschritte beim Verständnis auf Mikro- und Makroskala erreicht hat, gibt es noch immer nur wenige Ansätze, die Funktionsweise von Neuronenverbänden auf der mittleren Ebene zu erforschen.[6]
Makroskala: Verbindungen zwischen Hirnarealen
Aufgrund ihrer anatomischen Eigenschaften oder ihrer Funktion lassen sich verschiedene Hirnareale unterscheiden, die bei Betrachten des Konnektoms auf Makroebene die Knoten des Netzwerkgraphen bilden. Verbunden sind diese Zentren durch längere Nervenfasern, die die weiße Substanz bilden. Aufgrund von Erkenntnissen, die mittels spezieller Präparations- und Färbeverfahren gewonnen wurden, ist der generelle Verlauf vieler großer Nervenbündel auf der Makroebene bekannt.[7] Ziel aktueller Forschung (Stand: 2012) ist es, mittels bildgebender Verfahren das individuelle Konnektom lebender Probanden zu untersuchen und Einflüsse genetischer Faktoren, normaler Alterungsprozesse, sowie von Lernprozessen und Krankheiten aufzuklären. Neben wichtigen Beiträgen zahlreicher kleinerer Forschungsprojekte stellt das Human Connectome Project im Zeitraum 2010 bis 2015 das größte koordinierte Forschungsprogramm in diesem Bereich dar. Der Forschung stehen derzeit im Wesentlichen die folgenden drei Verfahren zur Verfügung:
Die Diffusions-Bildgebung ermöglicht eine direkte Untersuchung der weißen Substanz. Sie misst die brownsche Molekularbewegung von Wassermolekülen. Da diese durch die Mikrostruktur der Nervenfasern eingeschränkt ist, ermöglicht ihre Vorzugsrichtung eine Schätzung der lokalen Ausrichtung der Nervenfasern. Traktographie-Verfahren rekonstruieren aus solchen Daten algorithmisch den Verlauf der großen Nervenbahnen. Es wurden unterschiedliche Verfahren vorgeschlagen, um aus den Diffusions-Daten einen Netzwerkgraphen abzuleiten: Eine Möglichkeit besteht darin, die Hirnareale (d. h. die Knoten des Graphen) einer kortikalen Karte zu entnehmen; Gong u. a. definieren auf diese Weise 78 Regionen.[8] Eine feinere Unterteilung in 500 bis 4000 Knoten kann durch eine zufällige Gruppierung von je 8 bis 64 zusammenhängenden Voxeln erreicht werden, die in diesem Fall jedoch keine anatomische Bedeutung besitzen.[9] Zwei Knoten werden durch eine Kante verbunden, wenn die Traktographie eine Verbindung der entsprechenden Areale rekonstruiert hat; Kantengewichte können aus der Zahl der rekonstruierten Kurven abgeleitet werden.[9] Wenn das gemeinsame Konnektom einer ganzen Gruppe von Probanden dargestellt werden soll, wird eine Kante nur dann gesetzt, wenn die Verbindung innerhalb der Gruppe verlässlich reproduziert wurde.[8]
Ergänzend zu der Diffusions-Bildgebung, deren Netzwerkgraphen im Kontext der Konnektomik als Ausdruck einer „strukturellen“ Konnektivität betrachtet werden, kommt auch die funktionelle Magnetresonanztomographie (fMRT) zum Einsatz. Insbesondere werden Korrelationen im BOLD-Signal, das indirekter Ausdruck der Hirnaktivität ist, als Indiz für eine Verbindung der entsprechenden Hirnregionen gewertet. Besonders verbreitet sind Messungen im Ruhezustand (resting state); die dabei beobachteten Netzwerke werden daher als resting state networks bezeichnet. Von der funktionellen Bildgebung erhofft man sich im Kontext der Konnektomik insbesondere eine Darstellung polysynaptischer Verbindungen, die etwa eine Umschaltung im Thalamus enthalten und daher mit der Diffusions-Bildgebung nicht zuverlässig erkannt werden. Im Gegensatz zu Traktographie-Verfahren zeigt die fMRT zwar die verbundenen Hirnregionen, nicht aber den Verlauf der Nervenverbindung selbst; im Gegenzug leidet sie weniger unter kreuzenden oder auffächernden Faserbahnen, die die Genauigkeit und Verlässlichkeit von Traktographie-Verfahren einschränken.[10]
Ein dritter Ansatz führt systematische Korrelationen in der Dicke verschiedener Kortexregionen auf eine Verbindung der korrelierten Areale zurück. Diese Dicke lässt sich aus herkömmlichen T1-gewichteten Bildern der Magnetresonanztomographie schätzen.[11] Da diese Korrelationen sich nur durch den Vergleich zwischen Probanden berechnen lassen, ergibt sich in diesem Fall kein individuelles Konnektom, sondern ein gemeinsamer Graph für alle Probanden der untersuchten Gruppe.
Das Konnektom im Kontext funktioneller Spezialisierung und Integration
Der Theorie funktioneller Spezialisierung und Integration zufolge erfordern konkrete Funktionen des Gehirns eine Integration spezialisierter Hirnareale.[12] Eine wesentliche Motivation für die Erforschung des Konnektoms ist die Annahme, dass die Verbindungen der Nervenzellen untereinander wesentlich zu beiden Aspekten beitragen. So wird zum Beispiel einerseits die Funktion des visuellen Cortex primär durch seine afferente Verbindung zu den Sinneszellen im Auge bestimmt. Andererseits weiß man aus Experimenten mit Split-Brain-Patienten, dass die Faserbahnen im Corpus callosum zur Integration visueller Eindrücke des linken Gesichtsfelds mit den Sprachzentren unerlässlich sind.
Während die funktionelle Spezialisierung durch Methoden der Elektrophysiologie und der funktionellen Bildgebung inzwischen relativ gründlich erforscht und belegt ist, erhofft man sich von der Erforschung des Konnektoms insbesondere weitere Einsichten über die bislang weniger detailliert verstandenen Mechanismen der funktionellen Integration.[13]
Literatur
- Sebastian Seung: Das Konnektom – Erklärt der Schaltplan des Gehirns unser Ich? Springer Spektrum, 2013, ISBN 978-3-642-34294-3.
Weblinks
- Jan Dönges: Der Schaltplan der Denkmaschine. In: spektrumdirekt. 18. März 2011
- Konnektom – Die Verkabelung des Gehirns. Themenseite auf Spektrum (Mai 2011, zuletzt abgerufen am 4. Mai 2014)
- Olaf Sporns: Connectome. In: Scholarpedia. (englisch)
- Oliver Schmitt, Peter Eipert: neuroVIISAS. auf: neuroviisas.med.uni-rostock.de, Konnektomanalyse Software. (englisch)
- Oliver Schmitt, Peter Eipert: Rat connectome. auf: neuroviisas.med.uni-rostock.de, Zentrales und peripheres multiskalen Konnektom der Laborratte. (englisch)
Einzelnachweise
- O. Sporns, G. Tononi, R. Kötter: The Human Connectome: A Structural Description of the Human Brain. In: PLoS Comput Biol. 1(4), 2005, S. e42.
- P. Hagmann: From Diffusion MRI to Brain Connectomics. Dissertation. École polytechnique fédérale de Lausanne, 2005.
- J. G. White, E. Southgate, J. N. Thomson, S. Brenner: The structure of the nervous system of the nematode Caenorhabditis elegans. In: Phil. Trans. Royal Soc. London. B 314, 1986, S. 1–340.
- O. Sporns: Connectome. In: Scholarpedia. 5(2), 2009, S. 5584. Revision 91162.
- S. C. Turaga, J. F. Murray, V. Jain, F. Roth, M. Helmstaedter, K. Briggman, W. Denk, H. S. Seung: Convolutional networks can learn to generate affinity graphs for image segmentation. In: Neural Computation. Band 22, Nr. 2, 2010, S. 511–538, doi:10.1162/neco.2009.10-08-881, PMID 19922289.
- Das Manifest. Was wissen und können Hirnforscher heute? In: Gehirn&Geist. Juni 2004.
- E. Ludwig, L. Klingler: Atlas cerebri humani. S. Karger AG, Basel 1956.
- G. Gong, Y. He, L. Concha, C. Lebel, D. W. Gross, A. C. Evans, C. Beaulieu: Mapping Anatomical Connectivity Patterns of Human Cerebral Cortex Using In Vivo Diffusion Tensor Imaging Tractography. In: Cerebral Cortex. Band 19, 2009, S. 524–536, doi:10.1093/cercor/bhn102, PMC 2722790 (freier Volltext).
- P. Hagmann, M. Kurant, X. Gigandet, P. Thiran, V. J. Wedeen, R. Meuli, J.-P. Thiran: Mapping Human Whole-Brain Structural Networks with Diffusion MRI. In: PLoS ONE. 2(7), 2007, S. e597.
- K. R. A. Van Dijk, T. Hedden, A. Venkataraman, K. C. Evans, S. W. Lazar, R. L. Buckner: Intrinsic Functional Connectivity As a Tool For Human Connectomics: Theory, Properties, and Optimization. In: Journal of Neurophysiology. 103, 2010, S. 297–321.
- Y. He, Z. J. Chen, A. C. Evans: Small-World Anatomical Networks in the Human Brain Revealed by Cortical Thickness from MRI. In: Cerebral Cortex. 17, 2007, S. 2407–2419.
- K. Friston: Functional Integration. In: K. Friston u. a. (Hrsg.): Statistical Parametric Mapping. The Analysis of Functional Brain Images. Academic Press, 2011.
- O. Sporns: Discovering the Human Connectome. MIT Press, 2012, S. 20.