Immunpräzipitation
Eine Immunpräzipitation (IP, englisch immunoprecipitation) ist eine biochemische Methode, bei der in einem Pulldown-Assay mittels eines Antikörpers ein Antigen aus einer Lösung konzentriert wird.[1][2]
Prinzip
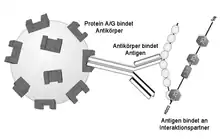
Das aufzureinigende Antigen ist meistens ein Biopolymer, z. B. ein Protein, Peptid, Polysaccharid oder eine Nukleinsäure. Bei Proteinen wird oftmals die Bindung eines Antikörpers an Protein-Tags verwendet, z. B. beim TAP-Tag. Die Aufreinigung von Nukleinsäuren per Immunpräzipitation ist für DNA unter Chromatin-Immunpräzipitation beschrieben, auch unter ChIP-on-Chip und ChIP-Seq, sowie für RNA unter RIP-Seq (iCLIP, PAR-CLIP und CLIP-Seq) und RIP-Chip.
In der Regel ist der Antikörper dabei in vitro an eine feste stationäre Phase (quervernetzte Agarose- oder Dextrankügelchen) gekoppelt und bindet über seine Affinität ein spezifisches Antigen in einer Lösung, beispielsweise in einem durch Filtration oder Zentrifugation geklärten Gewebelysat. Ein bestimmtes Antigen wird dabei mitsamt seinen Interaktionspartnern (Copräzipitate) also aus einem Gemisch präzipitiert. Dadurch eignet sich die Immunpräzipitation auch zum Nachweis von Protein-Protein-Wechselwirkungen, da ganze Proteinkomplexe mit dieser Methode präzipitiert werden können. Um unspezifische Wechselwirkungen von unerwünschten Molekülen zu mindern, werden die Bindungsstellen der stationären Phase mit ausreichenden Mengen des Zielproteins abgesättigt, so dass bindende Moleküle möglichst spezifisch gebunden werden.[3][4]
Ein präzipitiertes Protein und seine Interaktionspartner können im Anschluss mit unterschiedlichen Methoden nachgewiesen werden, beispielsweise mit Immunkonjugaten in einem Western Blot, Peptidmassenfingerprint oder durch vorherige Markierung mit Radioimmunkonjugaten.
Das Proteingemisch kann ein Homogenisat eines Gewebes sein oder aber Zellen aus der Zellkultur. Die Zellkultur ermöglicht es dabei auch, die Partner der vermuteten Interaktion zu überexprimieren, d. h. diese Proteine werden vermehrt gebildet. Der Interaktionspartner sollte in dieser Situation noch gebunden sein. Nachdem Zellen oder Gewebe aufgebrochen wurden, gibt man nun Antikörper hinzu, welche an eines der Proteine spezifisch binden. Über diese Antikörper wird dann das gesuchte Protein samt Interaktionspartner aus der Lösung per Zentrifugation oder MACS sedimentiert. Mehrere Waschschritte dienen der Entfernung unspezifisch gebundener Proteine. Die Proteine werden von den Beads durch Denaturierung gelöst und der Nachweis erfolgt meistens über einen Western Blot.
Hierbei bedient man sich in der Regel der spezifischen Eigenschaften von so genanntem Protein A, das aus der Zellwand des Bakteriums Staphylococcus aureus stammt, und/oder Protein G, welches ein Bestandteil der Zellwand von bestimmten Streptokokken-Stämmen ist. Protein A und G binden mit hoher Spezifität an die Fc-Region der meisten Säugetier-Immunglobuline. Mit diesen Proteinen werden nun Kügelchen beschichtet (engl. beads, z. B. aus Sepharose oder magnetischen Mikropartikeln), um in einer solchen Immunpräzipitation die Antikörper-Protein-Komplexe an sich zu binden.[5]
Quantitative Immunpräzipitation
Ausgefallene oder lösliche Antigen-Antikörper Immunkomplexe trüben eine Lösung. Verwendet man gereinigte Antigenstandards so kann durch Messung der Trübung per Immunnephelometrie auf die Antigenkonzentration in der Probe geschlossen werden.[6]
.svg.png.webp)
Vor- und Nachteile
Eingesetzt wird die IP im Zuge einer Proteinreinigung und Proteincharakterisierung als alternativer Nachweis einer Interaktion, z. B. nach einem Hefe-Zwei-Hybrid-Screen. Sie ermöglicht die Untersuchung von Protein-Protein-Interaktionen in zumindest in vivo-ähnlichen Verhältnissen, d. h. im Milieu einer Zelle und mit in Eukaryoten vorkommenden posttranslationalen Modifikationen wie Glykosylierung (Anhängen von Zuckerketten), Palmitoylierung (Anhängen von Fettsäuren) oder einer Faltung durch Chaperone.
Es ist aber möglich, dass sich Proteine durch das Aufbrechen der Zellen verändern oder auch abgebaut werden. Ein weiteres Problem der Methode ist bei der Immunpräzipitation eine mangelnde Reinheit des bindenden Proteins, die zu falsch positiven Ergebnissen führen kann. Weiterhin sind die Ergebnisse der Immunpräzipitation zum Teil von der spezifischen Bindung des Antikörpers abhängig, heterophile Antikörper können unspezifisch unerwünschte Proteine präzipitieren. Auf der anderen Seite binden manche Proteine auch direkt an die Beads oder an die Oberfläche der Reaktionsgefäße. Diese gebundenen Moleküle können eine nicht vorhandene Interaktion vorgaukeln (falsch positiv), welche nur über zusätzliche Kontrollversuche behoben werden kann. Des Weiteren ist es möglich, dass zwei Proteine zwar im IP-Versuch interagieren, aber im Zellzyklus, im Zellorganell oder im Zelltyp nicht gleichzeitig auftreten und deshalb nicht tatsächliche Interaktionspartner sein können. Da unter suboptimalen Umgebungsbedingungen auch falsch-negative Ergebnisse produziert werden können, dienen wiederholte Versuchsserien mit veränderten Pufferbedingungen einer Minderung falsch-negativer Ergebnisse.
Aus den genannten Gründen muss die Interpretation von IP-Ergebnissen mit Vorsicht erfolgen. Positive Interaktionen müssen immer mit weiteren Techniken verifiziert werden, wie beispielsweise Hefe-Zwei-Hybrid-System oder FRET. Eine Immunpräzipitation gibt zwar Auskunft über die mögliche Interaktion von zwei Proteinen, jedoch keine Informationen darüber, wie diese Interaktion stattfindet. Dazu sind detailliertere Untersuchungen der Struktur der beteiligten Proteine nötig.
Einzelnachweise
- B. Kaboord, M. Perr: Isolation of proteins and protein complexes by immunoprecipitation. In: Methods Mol Biol. (2008), Band 424, S. 349–364. doi:10.1007/978-1-60327-064-9_27. PMID 18369874.
- C. Dickson: Protein techniques: immunoprecipitation, in vitro kinase assays, and Western blotting. In: Methods Mol Biol. (2008), Band 461, S. 735–744. doi:10.1007/978-1-60327-483-8_53. PMID 19030838.
- Bonifacino, J. S., Dell'Angelica, E. C. and Springer, T. A. 2001. Immunoprecipitation. Current Protocols in Molecular Biology. 10.16.1–10.16.29.
- Ian Rosenberg: Protein analysis and purification: benchtop techniques. Springer, 2005, ISBN 978-0-8176-4340-9, S. 520.
- S. C. Masters: Co-immunoprecipitation from transfected cells. In: Methods Mol Biol. (2004), Band 261, S. 337–350. PMID 15064468.
- Christine Schütt, Barbara Bröker: Grundwissen Immunologie, 2. Ausgabe, Springer, 2009, ISBN 9783827420275