Hefe-Zwei-Hybrid-System
Beim Hefe-Zwei-Hybrid-System (englisch Yeast Two-Hybrid System, abgekürzt Y2H) handelt es sich um eine biochemische Technik zur Aufklärung von Protein-Protein-Interaktionen.[1][2]
Prinzip
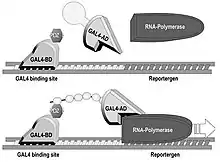
Das Hefe-Zwei-Hybrid-System ist eine In-vitro-Methode zum Nachweis von Protein-Protein-Wechselwirkungen in Hefe, in der Regel der Bäckerhefe Saccharomyces cerevisiae.[3] Grundlage hierfür ist ein zur Genregulation benötigtes Protein, ein so genannter Transkriptionsfaktor. In Saccharomyces cerevisiae bedient man sich normalerweise des Transkriptionsfaktors GAL4. Dieser besitzt zwei verschiedene Domänen, eine zum Binden an der DNA (Bindedomäne, GAL4-BD) und eine, welche die Transkription aktiviert (Aktivierungsdomäne, GAL4-AD). Obwohl die beiden Domänen sich normalerweise auf derselben Polypeptidkette befinden, sind diese auch dann wirksam, wenn sie von zwei unterschiedlichen Proteinen über nicht-kovalente Protein-Protein-Interaktionen zusammengebracht werden. Dazu bedient man sich zweier in Hefe kompatibler Expressionsvektoren.
Jedes der beiden Plasmide trägt jeweils ein für das entsprechende Experiment konstruiertes Fusionsgen. Dieses codiert im ersten Fall für ein Hybridprotein, das aus der GAL4-BD besteht und an der sich die Aminosäuresequenz anschließt, für die ein potentieller Bindungspartner gefunden werden soll (Bait-Protein, Köderprotein). Das zweite Plasmid codiert ein Hybridprotein, das sich aus der GAL4-AD und im Anschluss aus einem möglichen Bindepartner für das Bait-Protein zusammensetzt (Prey-Protein, Beuteprotein). Beide Hefe-Zwei-Hybrid-Plasmide replizieren sich sowohl in Hefe als auch in E. coli autonom (Shuttle-Vektoren).
Ein Hefestamm, der kein funktionierendes GAL4-Gen hat und ein oder mehrere Reportergene trägt, wird mit beiden Plasmiden transformiert. Als Reporter dieser Interaktion fungieren Gene, denen eine oder mehrere Bindestellen für den GAL4-Transkriptionsfaktor vorgeschaltet sind und die für Proteine codieren, die entweder bestimmte Aminosäuren bzw. Basen herstellen können (z. B. Histidin, Uracil oder Adenin) oder eine optische Erkennung ermöglichen (z. B. ein Farbumschlag katalysiert durch das lacZ-Gen). Wenn es zwischen Bait und Prey zu einer Interaktion kommt, resultiert daraus in der Regel eine funktionelle Rekonstitution des GAL4-Transkriptionsfaktors, was eine Expression der Reportergene zur Folge hat. Letzteres kann durch Wachstum auf entsprechenden Selektionsmedien nachgewiesen werden, z. B. auf einem Mangelmedium für Histidin wachsen in der Folge nur Hefen, bei welchen Bait und Prey interagieren und so das Enzym zur Histidin-Synthese exprimieren.
In einem Screening-Verfahren können in einem eher empirischen Ansatz mit einer cDNA-Bank als „Prey“ mögliche Interaktionspartner identifiziert werden, oder aber es kann bei dem so genannten „Single mating“ mit diesem System gezielt die Interaktion für bestimmte Proteine überprüft werden. Die DNA-Sequenzen können aus einer DNA-Reinigung oder einer Synthese stammen.[2] Es gibt Varianten des Y2H in E. coli und Säugetier-Zellen.[2][4]
Vor- und Nachteile
Das Hefe-Zwei-Hybrid-System ermöglicht die Untersuchung von Protein-Protein-Interaktionen in zumindest in vivo-ähnlichen Verhältnissen, d. h. im Milieu einer Zelle und mit in Eukaryoten vorkommenden posttranslationalen Modifikationen wie Glykosylierung (Anhängen von Zuckerketten), Palmitoylierung (Anhängen von Fettsäuren) oder Faltung durch Chaperone. Da Hefe zudem relativ billig und robust zu handhaben ist, können viele Interaktionspartner, z. B. bei Screening-Ansätzen, überprüft werden.
Problematisch beim klassischen Hefe-Zwei-Hybrid-System ist aber, dass die Interaktion der zu untersuchenden Proteine im Zellkern der Hefe stattfinden muss, da die Transkription nur dort ablaufen kann. Es ist aber möglich, dass sich Proteine in diesem Milieu anders falten als in dem Bereich der Zelle, in dem sie üblicherweise auftreten. Auch die Modifikationen der Hefe sind teilweise verschieden zu denen anderer eukaryotischer Organismen. Diese veränderte Faltung beeinflusst Struktur und Oberflächeneigenschaften der Proteine, wodurch es zu falschen Versuchsergebnissen kommen kann:
- Proteine, die normalerweise nicht miteinander interagieren würden, tun dies durch die veränderte Oberfläche (falsch positiv)
- Proteine, die tatsächlich miteinander interagieren, können dies durch die veränderte Faltung im Zellkern nicht (falsch negativ)
Des Weiteren ist es möglich, dass zwei Proteine zwar im Y2H-Versuch interagieren, aber im Zellzyklus, im Zellorganell oder im Zelltyp nicht gleichzeitig auftreten und deshalb nicht tatsächliche Interaktionspartner sein können.
Aus den genannten Gründen muss die Interpretation von Y2H-Ergebnissen mit großer Vorsicht erfolgen. Positive Interaktionen müssen immer mit weiteren Techniken aus der Molekularbiologie verifiziert werden, wie beispielsweise Immunpräzipitation oder FRET. Das Y2H-Experiment gibt weiterhin zwar Auskunft über die mögliche Interaktion von zwei Proteinen, jedoch keine Informationen darüber, wie diese Interaktion stattfindet. Dazu sind detailliertere Untersuchungen der Struktur der beteiligten Proteine nötig.
Das klassische, hier beschriebene Hefe-Zwei-Hybrid-System besitzt noch weitere Einschränkungen. So lassen sich bestimmte Proteine nicht in den Zellkern transportieren, wie beispielsweise Membranproteine. Auch Proteinkomplexe, an denen mehr als zwei Proteine beteiligt sind, lassen sich mit klassischen Y2H-Experimenten nicht direkt untersuchen. Um diesen Problemen zu begegnen, wurden inzwischen modifizierte Hefe-Zwei-Hybrid-Systeme entwickelt, bei denen erst weitere Schritte die Rekonstitution des Transkriptionsfaktors ermöglichen. Auch gibt es inzwischen Systeme, die auf anderen Prinzipien beruhen, wie z. B. das Split-Ubiquitin System, das besonders für Interaktionsstudien von Membranproteinen interessant ist.
Einzelnachweise
- S. Fields, O. Song: A novel genetic system to detect protein-protein interactions. (abstract) In: Nature. 340, Nr. 6230, 1989, S. 245–6. bibcode:1989Natur.340..245F. doi:10.1038/340245a0. PMID 2547163.
- Hurt J, Thibodeau S, Hirsh A, Pabo C, Joung J: Highly specific zinc finger proteins obtained by directed domain shuffling and cell-based selection. In: Proc. Natl. Acad. Sci. U.S.A.. 100, Nr. 21, 2003, S. 12271–6. bibcode:2003PNAS..10012271H. doi:10.1073/pnas.2135381100. PMID 14527993. PMC 218748 (freier Volltext).
- V. Ratushny, E. Golemis: Resolving the network of cell signaling pathways using the evolving yeast two-hybrid system. In: BioTechniques. Band 44, Nummer 5, April 2008, ISSN 0736-6205, S. 655–662, doi:10.2144/000112797, PMID 18474041, PMC 2526548 (freier Volltext).
- B. Stynen, H. Tournu, J. Tavernier, P. Van Dijck: Diversity in genetic in vivo methods for protein-protein interaction studies: from the yeast two-hybrid system to the mammalian split-luciferase system. In: Microbiology and molecular biology reviews : MMBR. Band 76, Nummer 2, Juni 2012, ISSN 1098-5557, S. 331–382, doi:10.1128/MMBR.05021-11, PMID 22688816, PMC 3372256 (freier Volltext).