Cystathionin-β-Lyase
Die Cystathionin-β-Lyase (CBL) ist ein Enzym aus der Gruppe der Lyasen, dass an der Übertragung von schwefelhaltigen Molekülen (Transsulfurierung) im Aminosäurestoffwechsel beteiligt ist.[1]
| Cystathionin-β-Lyase (Escherichia coli) | ||
|---|---|---|
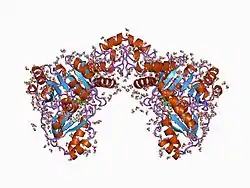 | ||
| Tertiärstruktur der Cystathionin-β-Lyase von Escherichia coli nach PDB 1cl1 | ||
| Andere Namen |
| |
| Masse/Länge Primärstruktur | 395 Aminosäuren, 43212 Da | |
| Sekundär- bis Quartärstruktur | Homotetramer | |
| Kofaktor | Pyridoxalphosphat | |
| Bezeichner | ||
| Gen-Name(n) | metC (E. coli), STR3 (S. cerevisiae) | |
| Externe IDs |
| |
| Enzymklassifikation | ||
| EC, Kategorie | 4.4.1.8, Lyasen | |
| Reaktionsart | Transsulfurierung | |
| Substrat | Cystathionin, Wasser | |
| Produkte | Homocystein, Pyruvat, Ammoniak | |
| Vorkommen | ||
| Übergeordnetes Taxon | Bakterien, Hefen und Pflanzen | |
Eigenschaften
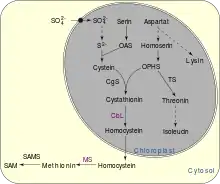
Die Cystathionin-β-Lyase kommt in Bakterien, Hefen und Pflanzen vor.[2] Sie katalysiert die Hydrolyse von Cystathionin, wodurch unter Abgang eines Amins Homoserin und Pyruvat entsteht.[3] Sie ist unter anderem am Methionin-, Cystein-, Selenocystein-, Stickstoff- und am Schwefelstoffwechsel (Transsulfurierung) beteiligt. Als Cofaktor wird Pyridoxalphosphat (PLP) verwendet.[2] Die Cystathionin-β-Lyase ist strukturell mit der Cystathionin-γ-Synthase verwandt.[4] Die CBL kommt nicht in Tieren vor, weshalb sie ein Target bei der Entwicklung von Antibiotika ist.[2] Die pflanzliche CBL ist etwas größer und bindet neben Cystathionin auch die nichtproteinogene Aminosäure Djenkolsäure.[5]
Einzelnachweise
- S. Holt, A. G. Cordente, C. Curtin: Saccharomyces cerevisiae STR3 and yeast cystathionine β-lyase enzymes: The potential for engineering increased flavor release. In: Bioengineered bugs. Band 3, Nummer 3, 2012 May-Jun, S. 178–180, doi:10.4161/bbug.19566, PMID 22572787, PMC 3370937 (freier Volltext).
- D. Q. Nguyen, H. P. Ngo, Y. J. Ahn, S. H. Lee, L. W. Kang: Expression, crystallization and preliminary X-ray crystallographic analysis of cystathionine β-lyase from Acinetobacter baumannii OXA-23. In: Acta crystallographica. Section F, Structural biology communications. Band 70, Pt 10 Oktober 2014, S. 1368–1371, doi:10.1107/S2053230X14017981, PMID 25286941.
- A. F. Jaworski, S. M. Aitken: Exploration of the six tryptophan residues of Escherichia coli cystathionine β-lyase as probes of enzyme conformational change. In: Archives of biochemistry and biophysics. Band 538, Nummer 2, Oktober 2013, S. 138–144, doi:10.1016/j.abb.2013.07.006, PMID 23969077.
- A. L. Manders, A. F. Jaworski, M. Ahmed, S. M. Aitken: Exploration of structure-function relationships in Escherichia coli cystathionine γ-synthase and cystathionine β-lyase via chimeric constructs and site-specific substitutions. In: Biochimica et Biophysica Acta. Band 1834, Nummer 6, Juni 2013, S. 1044–1053, doi:10.1016/j.bbapap.2013.02.036, PMID 23470500.
- U. Breitinger, T. Clausen, S. Ehlert, R. Huber, B. Laber, F. Schmidt, E. Pohl, A. Messerschmidt: The three-dimensional structure of cystathionine beta-lyase from Arabidopsis and its substrate specificity. In: Plant physiology. Band 126, Nummer 2, Juni 2001, S. 631–642, PMID 11402193, PMC 111155 (freier Volltext).