Bodo-saltans-Virus
„Bodo-saltans-Virus“ (BsV, englisch „Bodo saltans virus)“ ist eine vorgeschlagene Spezies von Riesenviren im Phylum Nucleocytoviricota (früher Nucleocytoplasmic large DNA viruses, NCDLV) der Familie Mimiviridae und möglicherweise in der Gattung „Klosneuvirus“,[3] oder wenigstens der Klade der Klosneuviren (vorgeschlagene Unterfamilie „Klosneuvirinae“). Diese wurde in einem Süßwasserteich in British Columbia entdeckt. Der natürliche Wirt von BsV ist die Spezies Bodo saltans[4] [en], ein Einzeller, der zu den Kinetoplastiden zählt. Die meisten bis dato bekannten anderen Mitglieder der Mimiviridae infizieren stattdessen Amöben der Gattung Acanthamoeba.[1][5]
| „Bodo-saltans-Virus“ | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
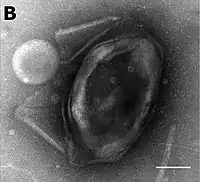
BsV-Partikel mit einem blütenartig | ||||||||||||||||||||
| Systematik | ||||||||||||||||||||
| ||||||||||||||||||||
| Taxonomische Merkmale | ||||||||||||||||||||
| ||||||||||||||||||||
| Wissenschaftlicher Name | ||||||||||||||||||||
| „Bodo saltans virus“ | ||||||||||||||||||||
| Kurzbezeichnung | ||||||||||||||||||||
| BsV | ||||||||||||||||||||
| Links | ||||||||||||||||||||
|
Die Spezies ist (Stand März 2020) noch nicht vom International Committee on Taxonomy of Viruses (ICTV) bestätigt.[2]
Beschreibung
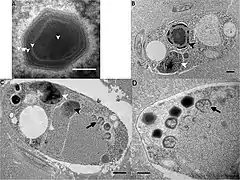
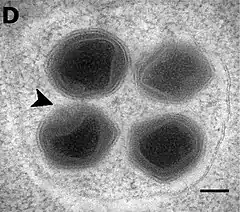
Die Viruspartikel (Virionen) von BsV ähneln denen der Mimiviren und besitzen wie diese auf dem Kapsid eine seesternartige Struktur, „englisch Stargate“ (Sternentor) genannt. Sie haben einen Durchmesser von ca. 300 nm. BsV-Virionen replizieren sich in für Mimiviridae typischer Weise in zytoplasmatischen Virusfabriken.[1]
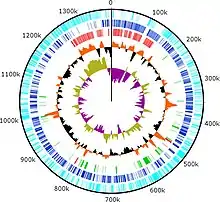
Das Genom von BsV ist linear und besteht aus doppelsträngiger DNA (dsDNA). Es hat eine Länge von 1.385.869 bp (Basenpaaren). Der GC-Gehalt liegt bei 25,3 %. Nach den Untersuchungen sollte das Genom aus 1.227 offenen Leserahmen (englisch openreading frames, ORFs) bestehen und vorhergesagt 1.207 Proteine kodieren. Bei 27 % dieser kodierten Proteine wurde Homologie zu bekannten eukaryotischen Sequenzen gefunden, bei mehr als der Hälfte gibt es keine erkennbaren Übereinstimmungen. Die kodierten Proteine sind beteiligt an der DNA-Replikation und -Reparatur, der RNA-Synthese und -Modifikation. Außerdem gibt es Translationsproteine und Kapsidproteine.[1][5][6]
Anders als bei anderen bekannten Riesenviren gibt es keine Gene für tRNAs. Da BsV keine an der Proteinsynthese beteiligten Enzyme kodiert, muss das Virus bei zu diesem Zweck Wirts-tRNAs verwenden.[1][5]
Gene, die ein System zur Fusion von Membranen kodieren, könnten durch horizontalen Gentransfer vom eukaryotischen Wirt abstammen. Diese könnten es den Virionen ermöglichen, in die Wirtszelle einzudringen.[1][5]
Mobile genetische Elemente sind in großer Zahl vorhanden, einschließlich selbstspleißender Proteine (sogenannte Inteine), sowie als Ribozyme wirksame Sequenzen, die sich aus der RNA herausspleißen können. Solche mobilen genetischen Elemente hatte man zuvor noch nie bei anderen Riesenviren gefunden.[1][5]
Von einem Protein, das an der Bekämpfung der Wirtsabwehr beteiligt sein könnte, sind 148 Kopien vorhanden. Nach Ansicht der Autoren wurde dieses Gen wiederholt dupliziert, da eine größere Menge davon benötigt wurde, damit sich das Virus gut in den Wirtszellen replizieren kann. Eine ähnliche Vervielfachung von Genen, die einen Antagonisten eines Immunproteins des Wirts kodieren, wurde auch im Genom des Vacciniavirus (ebenfalls ein NCLDV aus der Familie der Poxviridae) beobachtet.[1][5][7]
Wirte
Der BsV-Stamm NG1 (BsV-NG1) infiziert den Bodo saltans-Stamm NG (CCCM6296), aber nicht HFCC12,[1]
Systematik
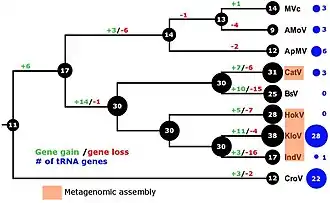
Die nächsten Verwandten von BsV sind unter anderem die in Klosterneuburg gefundenen Klosneuviren „Klosneuvirus“ (KnV bzw. KlosnV), „Hokovirus“ (HokV), „Catovirus“ (CatV) und „Indivirus“ (IndV). Mit ihnen zusammen gehört BsV zur Familie der vom ICTV bestätigten Familie Mimiviridae. Im Einzelnen sind die Verwandtschaftsbeziehungen noch in Diskussion. Meist wird BsV mit den in Klosterneuburg gefundenen vier Gattungen und ggf. weiteren zu einer vorgeschlagenen Unterfamilie „Klosneuvirinae“ zusammengefasst.[8]
Meist zeigt sich eine engere Verwandtschaft der Klosneuviren zu den Mimiviren als zu den Cafeteriaviren.[1] Abweichend davon sehen manche Autoren umgekehrt eine engere Verwandtschaft der Klosneuviren zu den Cafeteriaviren und „Namao-Virus“ und schlagen daher eine gemeinsame Unterfamilie „Aquavirinae“ dieser beiden Kladen vor.[9][10]
Während das NCBI das „Bodo-saltans-Virus“ (BsV) als Spezies in der Gattung „Klosneuvirus“ sieht,[3] schlagen Disa Bäckström et al. (2019), Fig. 3, eine Systematik der Klosneuviren mit dem „Catovirus“ als nächsten Verwandten von BsV unter den vier ursprünglichen Klosneuviren vor.[11] Weitere Kladogramme zur inneren Systematik der Klosneuviren finden sich unter Klosneuvirus §Systematik.
Deeg et al. (2018) verstehen die Klosneuviren als Subtaxon einer erweiterten Unterfamilie „Megavirinae“, die alle herkömmlichen Mimiviridae umfasst, und zu der die ehemals zu den Phycodnaviridae gestellten Kandidaten (mit der „OLPG“, „Organic Lake Phycodnavirus Group“ genannten Gruppe) mit einer Unterfamilie „Mesomimivirinae“ ein Schwestertaxon innerhalb einer umfangreichen Familie Mimiviridae bilden.[1]
Einzelnachweise
- Christoph M. Deeg, Cheryl-Emiliane T. Chow, Curtis A. Suttle: The kinetoplastid-infecting Bodo saltans virus (BsV), a window into the most abundant giant viruses in the sea. In: eLife. 7, 27. März 2018, S. e33014. doi:10.7554/eLife.33014.
- ICTV: ICTV Master Species List 2019.v1, New MSL including all taxa updates since the 2018b release, March 2020 (MSL #35)
- NCBI: Bodo saltans virus (Spezies), Zugriffsdatum: 2. August 2019
- NCBI: Bodo saltans Ehrenberg (species)
- Vincent Racaniello, David Tuller, Gertrud U. Rey: Bodo saltans virus, an abundant giant aquatic Mimivirus, auf: virology blog, 28. Dezember 2017
- David M. Needham, Alexandra Z. Worden et al.: A distinct lineage of giant viruses brings a rhodopsin photosystem to unicellular marine predators, in: PNAS, 23. September 2019, doi:10.1073/pnas.1907517116, ISSN 0027-8424, hier: Supplement 1 (xlsx)
- Vincent Racaniello, Alan Dove, Rich Condit, Kathy Spindler: Pox has got a squeeze-box, seals are gonna sneeze all night, auf: This Week in Virology, 2. September 2008
- Frederik Schulz, Natalya Yutin, Natalia N. Ivanova, Davi R. Ortega, Tae Kwon Lee, Julia Vierheilig, Holger Daims, Matthias Horn, Michael Wagner, Grant J. Jensen, Nikos C. Kyrpides, Eugene V. Koonin, Tanja Woyke: Giant viruses with an expanded complement of translation system components. In: Science. Band 356, Nr. 6333, 7. April 2017, ISSN 0036-8075, S. 82–85, doi:10.1126/science.aal4657, PMID 28386012.
- Centre national de la recherche scientifique: List of the main “giant” viruses known as of today, Université Aix Marseille, 18. April 2018.
- Centre national de la recherche scientifique: List of the main “giant” viruses known as of today (March 2019), Université Aix Marseille, März 2019.
- Disa Bäckström, Natalya Yutin, Steffen L. Jørgensen, Jennah Dharamshi, Felix Homa, Katarzyna Zaremba-Niedwiedzka, Anja Spang, Yuri I. Wolf, Eugene V. Koonin, Thijs J. G. Ettema; Richard P. Novick (Hrsg.): Virus Genomes from Deep Sea Sediments Expand the Ocean Megavirome and Support Independent Origins of Viral Gigantism, in: mBio Vol. 10 Nr. 2 2019, doi:10.1128/mBio.02497-18