SIR-Modell
Als SIR-Modell (susceptible-infected-removed model) bezeichnet man in der mathematischen Epidemiologie, einem Teilgebiet der Theoretischen Biologie, einen klassischen Ansatz zur Beschreibung der Ausbreitung von ansteckenden Krankheiten mit Immunitätsbildung, der eine Erweiterung des SI-Modells darstellt. Benannt ist es nach der Gruppeneinteilung der Population in Suszeptible (S), das heißt Ansteckbare, Infizierte (I) und aus dem Infektionsgeschehen entfernte Personen (R), wie unten erläutert. Die Erweiterung des SIR-Modells unter Einbezug der Exponierten, also Personen, die infiziert, aber noch nicht ansteckend sind, wird mit dem SEIR-Modell beschrieben. Üblicherweise wird ein deterministisches, durch miteinander verknüpfte gewöhnliche Differentialgleichungen formuliertes Modell betrachtet, bei dem die Variablen kontinuierlich sind und großen Gesamtheiten entsprechen. Es werden aber auch andere, insbesondere stochastische Modelle mit SIR bezeichnet, die mit dem deterministischen SIR-Modell die Gruppeneinteilung gemeinsam haben.
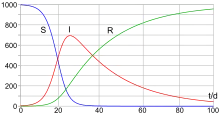
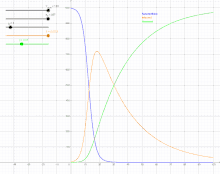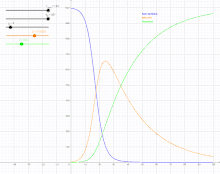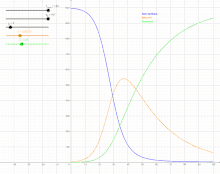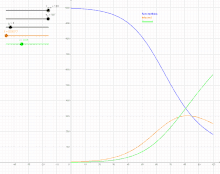
Das Modell stammt von William Ogilvy Kermack und Anderson Gray McKendrick (1927)[1] und wird auch manchmal nach beiden benannt (Kermack-McKendrick-Modell). Die Autoren konnten damit trotz der Einfachheit des Modells gut die Daten einer Pestepidemie in Bombay 1905/06 modellieren.
Differentialgleichungen
Beim SIR-Modell werden drei Gruppen von Individuen unterschieden: Zum Zeitpunkt bezeichnet die Anzahl der gegen die Krankheit nicht immunen Gesunden (susceptible individuals), die Zahl der ansteckenden Infizierten (infectious individuals) sowie die Anzahl der aus dem Krankheitsgeschehen „entfernten“ Personen (removed individuals). Letzteres erfolgt entweder durch Genesen mit erworbener Immunität gegen die Krankheit oder durch Versterben.[2][3] In anderer Lesart sind es die resistenten Personen.[4] Weiterhin sei die Gesamtzahl der Individuen, das heißt:
Der einfacheren Schreibweise wegen wird die Zeitabhängigkeit bei im Folgenden weggelassen. Es gilt für jede Zeit , , , .
Im SIR-Modell werden eine Reihe von Annahmen gemacht:
- Jedes Individuum kann von einem Erreger nur einmal infiziert werden und wird danach entweder immun oder stirbt.
- Infizierte sind sofort ansteckend, eine Annahme die im SEIR-Modell nicht getroffen wird.
- Die jeweiligen Raten sind konstant.
- Die durch die Infektion Verstorbenen werden wie die Immunisierten zu gerechnet.
Dann sind die Ratengleichungen des SIR-Modells:[4]
Mit den Raten:
- Rate, mit der infizierte Personen in der Zeiteinheit genesen oder sterben (da die Toten auch zu gerechnet werden)
- allgemeine Sterberate pro Person einer Population (also „pro Kopf“)
- Geburtsrate pro Person einer Population (also „pro Kopf“)
- , die Rate, die die Anzahl neuer Infektionen angibt, die ein erster infektiöser Fall pro Zeiteinheit verursacht. wird auch als Transmissionsrate oder Transmissionskoeffizient bezeichnet.[5]
kann weiter aufgeschlüsselt werden: , mit der Kontaktrate und der Wahrscheinlichkeit einer Infektionsübertragung bei Kontakt.
Die Infektionsrate (englisch force of infection) , also die "pro Kopf" Rate mit der suszeptible Personen infiziert werden, ist
wobei den Anteil infizierter Personen an der Gesamtbevölkerung und damit die Wahrscheinlichkeit des Kontakts mit einer infizierten Person darstellt. Es werden Personen pro Zeiteinheit infiziert.
Vernachlässigt man die Geburts- und Sterberaten (N ist dann konstant) ergeben sich die Gleichungen:
Die Gleichungen sind ähnlich den Lotka-Volterra-Gleichungen in Räuber-Beute-Systemen und gekoppelten Bilanzgleichungen auf vielen anderen Gebieten (Replikatorgleichungen).
In der Literatur wird zuweilen eine Variante der Gleichungen benutzt, in die der Transmissionskoeffizient eingeht und dabei oft ebenfalls mit bezeichnet wird, obwohl er einen anderen Wert hat: Ist etwa und unser Koeffizient oben , so muss in die Variante der Differentialgleichung der Wert eingesetzt werden. Verwenden wir der Klarheit halber einen anderen Bezeichner , so schreibt sich die erste Differentialgleichung in der Variante:[Anm. 1]
Basisreproduktionszahl und Verlauf einer Epidemie
Die Basisreproduktionszahl ist
Hierbei wird die übliche Bezeichnung für die Basisreproduktionszahl benutzt (sie ist nicht mit dem Anfangswert der Anzahl resistenter Personen zum Zeitpunkt zu verwechseln, der zuweilen auch mit bezeichnet wird).
Die Basisreproduktionszahl gibt an, welche Anzahl an weiteren Infektionen eine infizierte Person (während der Gesamtdauer ihrer infektiösen Periode) in der Anfangszeit der Epidemie in einer komplett suszeptiblen Bevölkerung verursacht. Neben tritt hier noch der Faktor auf, der die Dauer der infektiösen Periode angibt. In der Anfangszeit einer Epidemie kann man näherungsweise Geburts- und Sterberaten vernachlässigen, also setzen; dann erhält man die am Schluss des letzten Abschnitts angegebene Form der SIR-Gleichungen. Für den Beginn einer Epidemie muss sein und folglich (da am Anfang gilt) gemäß den SIR-Gleichungen und somit (siehe auch den folgenden Abschnitt über die diskretisierte Form der Gleichungen).
Im weiteren Verlauf wächst nach den SIR-Gleichungen die Zahl der Infizierten , wenn , also und damit
Links steht das Produkt aus Basisreproduktionszahl und Anteil der Infizierbaren (Suszeptiblen) an der Population. Letzterer ist gleichzeitig die Wahrscheinlichkeit, bei einem Kontakt auf einen Infizierbaren zu treffen. Die Ungleichung ist gleichbedeutend mit[4]
Das Wachstum der Infizierten nimmt ab (Abflauen bzw. Ende der Epidemie), falls den Wert unterschreitet. Bei einem Wert der Basisreproduktionszahl wäre das die Hälfte der Bevölkerung und bei ein Drittel, so dass im ersten Fall die Hälfte und in letzterem Fall zwei Drittel der Population infiziert oder resistent sind, d. h. nicht mehr empfänglich für eine Infektion sind; man spricht dann von „Herdenimmunität“.
Bei Influenza liegen beispielsweise die Basisreproduktionszahlen üblicherweise zwischen 2 und 3.
Diskretisierte Form der Differentialgleichungen
Die diskretisierte Form der Differentialgleichungen mit Zeitschritt lautet:[6]
entspricht der Zahl neu Infizierter Personen im Beobachtungszeitraum , also dem was auch in den offiziellen Statistiken als Zahl Neuinfizierter auftaucht,[6] wobei in der Praxis Korrekturen für Meldeverzug und anderes angebracht werden. Häufig wird ein Tag als Zeiteinheit und als Beobachtungszeitraum für die Meldung gewählt und gesetzt.
Für die Ableitung der Basisreproduktionszahl betrachte man die diskretisierte Form (Schritt ) der Differentialgleichung für :
Mit der Dauer der infektiösen Periode eingesetzt für und nach Definition der Basisreproduktionszahl bzw. Nettoreproduktionszahl :
wobei am Anfang der Epidemie als bezeichnet wird, ergibt sich
Damit ist
und für , da am Beginn der Epidemie , ergibt sich:
Mathematische Behandlung
Mit Hilfe des SIR-Modells können wir für gegebene Anfangswerte bestimmen, ob der Krankheitsverlauf in einer Epidemie münden wird. Diese Frage ist äquivalent zu der Frage, ob die Zahl der Infizierten zum Zeitpunkt steigt. Betrachte die Ableitung:
.
Hierbei nennen wir den Schwellenwert einer Epidemie, da aus für alle Zeiten die Ungleichung für alle folgt und für die Epidemie abflaut:
.
für alle .
Eine Epidemie tritt im SIR-Modell also genau dann auf, wenn ist. Dies ist eine wesentliche Aussage des Modells, auch als Schwellwert-Theorem bekannt.[7] Um eine Epidemie zu starten, muss eine Mindestdichte von Infizierbaren vorhanden sein. Wird die Zahl der Infizierbaren im Lauf der Epidemie umgekehrt unter diese Schwelle gedrückt, erlischt die Epidemie.
Maximale Zahl der Infizierten
Aus den obigen Differentialgleichungen für und folgt:
.
Integration durch Trennung der Variablen liefert:
mit der Logarithmusfunktion. Die Funktion ist ein erstes Integral des Systems und konstant auf den Trajektorien des Systems im durch und gegebenen Phasenraum. Die maximale Zahl der Infizierten ergibt sich offensichtlich für und bei . Mit der obigen Gleichung ergibt sich unter Annahme von :
Setzt man und sowie erhält man:
Aus den ersten Integralen ergibt sich auch die Gleichung für („final size equation“):
aus den Werten für (mit ) und (mit ). Die Gleichung kann zur Bestimmung von benutzt werden. Insbesondere ergibt sich für die Lösung , das heißt, es gibt keinen Ausbruch.[8]
Zahl der „Überlebenden“
Es stellt sich auch die Frage, ob die Epidemie überhaupt „überlebt“ wird, das heißt, ob am Ende noch Suszeptible übrigbleiben. Dazu berechnen wir , also mit der Zeit gegen Unendlich (). Analog ergibt sich aus den obigen Differentialgleichungen
, deren Lösung ist, mit der Exponentialfunktion .
Damit folgt offensichtlich , es wird also nicht die gesamte Population infiziert. Aus folgt damit außerdem . Es zeigt sich, dass es am Ende einer Epidemie weniger an Suszeptiblen als eher an Infizierten mangelt!
Näherungen: Reduziere Zahl der Parameter
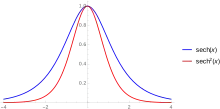
Wenn wir die Anfangswerte kennen, können wir mit den obigen Differentialgleichungen schnell die Dynamik einer Krankheit bestimmen. Oft lassen sich aber gerade diese Konstanten nur schwer bestimmen, weshalb wir im Folgenden die obigen Gleichungen nähern wollen.
Aus den besprochenen Differentialgleichungen folgt sofort[9]
Die Gleichung vereinfacht sich zu einer riccatischen Differentialgleichung, wenn durch die ersten 3 Summanden der Taylorreihe um angenähert wird:
also
wobei eingeführt wurden:
Die Funktion ist der Sekans hyperbolicus und der Tangens hyperbolicus, dessen Umkehrfunktion.
Damit lässt sich die Differentialgleichung für mit nur drei Parametern ausdrücken:
Diese drei Parameter sind also (bei dem anfänglich exponentiellen Wachstum entspricht mit der Verdopplungszeit), die Phase und . Je nach Datenlage kann hierbei die Differentialgleichung oder die implizite Gleichung für verwendet werden.
Setzt man und erhält man und damit:
Mit erhält man einen Näherungswert für das „Ausmaß der Epidemie“ :
Damit erhält man den zweiten Teil des Schwellwerttheorems. Sei am Anfang mit , dann ist das „Ausmaß der Epidemie“:
und . Die Anzahl suszeptibler Personen ist am Ende um gegenüber dem Stand vor der Epidemie reduziert.
Für die Zahl der Infizierten ergibt sich gemäß der letzten Differentialgleichung im SIR-Modell:
Der Verlauf von hat die Form einer Glockenkurve mit anfangs exponentiellem Anstieg. Kermack und McKendrick fanden zum Beispiel für die Pestepidemie in Bombay 1905/06 (mit fast immer tödlichem Ausgang, so dass , als Zeiteinheit für die Raten wurde eine Woche genommen) gute Übereinstimmung mit:[10]
Die gute Übereinstimmung machte dies zu einem häufig zitierten Beispiel in der mathematischen Epidemiologie, ist aber auch kritisiert worden.[11]
David George Kendall fand 1956 exakte Lösungen für und das SIR-Modell,[12] doch werden die Differentialgleichungen meist numerisch gelöst.
Erweiterung des Modells
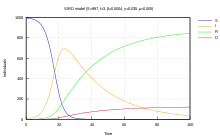
Will man die Toten separat betrachten (statt zur Gruppe R hinzuzurechnen), so kann man es zum SIRD-Modell (Susceptible-Infected-Recovered-Deceased-Model) erweitern. Hierbei gehören zur Gruppe R nur die Individuen, welche die Krankheit überlebt haben und immun geworden sind, und die Gestorbenen bilden eine eigene Gruppe D.[13]
Hierbei ist folgendes System von Differentialgleichungen zu lösen:[Anm. 2]
Zum Zeitpunkt gibt die Zahl der Genesenen und die Anzahl der an der Krankheit Verstorbenen an. Weiter bedeutet die Rate, mit der Infizierte gesunden, und die Mortalitätsrate, mit der Infizierte versterben. , sowie Transmissionsrate haben dieselbe Bedeutung wie beim SIR-Modell.
Eine weitere Modifikation berücksichtigt die Impfung von Neugeborenen mit einem Anteil :
Es gibt auch Varianten, in denen zwei (oder mehr) Bevölkerungsgruppen betrachtet werden, zum Beispiel die Wechselwirkung einer Kerngruppe, die besonders aktiv eine Infektion befördert, mit der Restpopulation.[4]
Stochastische SIR-Modelle[4] dienen der Untersuchung kleinerer Populationen, die mit deterministischen Modellen nicht gut behandelt werden können. Dabei werden nur ganzzahlige Werte der Populationsanteile betrachtet und statistische Verteilungen für die Übergangsraten wie . Der Verlauf von Epidemien ist hier nicht deterministisch vorbestimmt, d. h. eine Epidemie kann auch bei stoppen, wenn zufällig in der Infektionsperiode (infektiöse Periode, d. h. der Zeitspanne, in der ein Infizierter die Infektion übertragen kann) keine Kontakte stattfinden. Meist werden Simulationen (üblicherweise mit Monte-Carlo-Verfahren) mit den gleichen Parametern mehrfach durchgeführt und die Ergebnisse dann statistisch ausgewertet.
Eine Variante, die Quarantänemaßnahmen und Isolierungsmaßnahmen wie Soziale Distanzierung berücksichtigt, wurde für die Erklärung subexponentiellen Wachstums, das heißt Wachstum der Infizierten gemäß einem Potenzgesetz in der Zeit, bei Covid-19 in China ab Ende Januar 2020 herangezogen (SIR-X-Modell).[14] Die Differentialgleichungen lauten in diesem Fall nach Dirk Brockmann und Benjamin Maier (mit Anpassung an die hier gebrauchte Form der SIR-Gleichungen):
Dabei ist eine neu eingeführte Gruppe von symptomatischen infizierten Personen in Quarantäne und Isolation. Sie soll auch dem empirischen Vergleich mit den offiziell gemeldeten und bestätigten Fällen dienen. R sind die aus dem weiteren Infektionsgeschehen im Modell Entfernten (removed), entweder weil verstorben, genesen oder durch die allgemeinen Isolationsmaßnahmen, so weit sie nicht unter X fallen. Die allgemeinen Maßnahmen zur Kontaktreduzierung werden mit beschrieben (soziale Distanzierung u. a.) und betreffen Infizierte und nicht Infizierte gleichermaßen, die speziellen Quarantänemaßnahmen für Infizierte mit dem Koeffizienten . Entfallen die jeweiligen Maßnahmen ist . Es ergibt sich ein neues effektives mit einer effektiven Infektionsperiode . Dieses neue effektive ist kleiner als . Eine andere Methode den Einfluss von isolierenden Maßnahmen zu simulieren besteht darin, für zeitlich variable Ansätze zu machen.[15]
Eine weitere Erweiterung besteht darin, nicht die Gesamtzahl der Infizierten, sondern deren Dichte (Zahl der Infizierten pro Flächeneinheit) zu berechnen, sodass auch die Verteilung der Infizierten im Raum betrachtet werden kann. Hierzu wird das gewöhnliche SIR-Modell um Diffusionsterme erweitert:
wobei , und Diffusionskonstanten sind. Auf diese Weise erhält man eine Reaktions-Diffusions-Gleichung. (Damit die Einheiten korrekt sind, muss der Parameter modifiziert werden.) SIR-Modelle mit Diffusion wurden beispielsweise zur Beschreibung der Ausbreitung der Pest in Europa verwendet.[16] Erweiterte raumzeitliche SIR-Modelle ermöglichen die Beschreibung von kontaktreduzierenden Maßnahmen (social distancing).[17]
Ein von Matthias Kreck und Erhard Scholz entwickeltes an Covid-19 adaptiertes Modell berücksichtigt Effekte von Impfungen, Massentests und Mutanten. Das Modell wurde speziell auf die Entwicklung in Deutschland angewendet. Ein vergleichsweise milder Eingriff, der die Zeit bis zur Quarantäne um einen Tag reduziert kann zu einer drastischen Verbesserung führen, ebenso bestimmte Massentestungen. Das von Kreck und Scholz angepasste SIR-Modell weist im Unterschied zu dem Standard-SIR-Modell erhebliche Unterschiede auf, wenn die Kontaktraten nicht konstant sind. Die Modell-Reproduktionsrate weicht von der des RKI ab.[18]
Siehe auch
- Mathematische Modellierung der Epidemiologie (einführender Artikel)
- SI-Modell (Ansteckung ohne Gesundung)
- SIS-Modell (Ausbreitung von ansteckenden Krankheiten ohne Immunitätsbildung)
- SEIR-Modell (Ausbreitung von ansteckenden Krankheiten mit Immunitätsbildung, bei denen Infizierte nicht sofort infektiös sind)
- Dynamisches System (mathematischer Oberbegriff)
Literatur
- N. F. Britton: Essential Mathematical Biology. 1. Auflage. Springer, Berlin 2003, ISBN 1-85233-536-X.
- Michael Li: An introduction to mathematical modeling of infectious diseases, Springer, 2018
Weblinks
- Eric W. Weisstein: Kermack-McKendrick Model. In: MathWorld (englisch).
- Numberphile: The Coronavirus Curve auf YouTube, 25. März 2020, abgerufen am 16. Mai 2020.
Einzelnachweise
- Kermack, McKendrick: A contribution to the mathematical theory of epidemics. Proc. Roy. Soc. A, Band 115, 1927, S. 700–721.
- Sebastian Möhler: Ausbreitung von Infektionskrankheiten. (tu-freiberg.de [PDF; abgerufen am 12. März 2020]).
- Regina Dolgoarshinnykh: Introduction to Epidemic Modelling. (columbia.edu [PDF; abgerufen am 12. März 2020]).
- Eichner, Kretzschmar: Mathematische Modelle in der Infektionsepidemiologie. In: A. Krämer, R. Reintjes (Hrsg.): Infektionsepidemiologie. Springer, 2003, S. 81–94.
- Michael Li: An introduction to mathematical modeling of infectious diseases. Springer, 2018, Abschnitt 2.1, Kermick-McKendrick-Model.
- Viola Priesemann u.a.: Inferring change points in the spread of COVID-19 reveals the effectiveness of interventions. Science, 15. Mai 2020.
- Norman Bailey: The mathematical theory of infectious diseases. Griffin and Company, 1975, S. 11, treshold theorem.
- Odo Diekmann, Hans Heesterbeek, Tom Britton: Mathematical tools for understanding infectious disease dynamics. Princeton UP 2013, S. 15.
- Die nachfolgende Ableitung mit den zugehörigen Formeln findet sich z. B. in Michael Li: An introduction to mathematical modeling of infectious diseases. Springer, 2018, S. 45.
- Kermack, McKendrick: A contribution to the mathematical theory of epidemics. Proc. Royal Soc. A, Band 115, 1927, S. 714.
- Nicholas Bacaer: The model of Kermack and McKendrick for the plague epidemic in Bombay and the type reproduction number with seasonality. Journal of Mathematical Biology, Band 64, 2012, S. 403–422. Danach sind die erhaltenen konstanten Parameter unrealistisch und die Pestepidemie trat 1897 bis mindestens 1911 saisonal in Bombay auf, gekoppelt an die Rattenpopulation, so dass ein komplexeres Modell nötig ist.
- D. G. Kendall: Deterministic and stochastic epidemics in closed populations. Proc. Third Berkeley Symposium Math. Stat. & Prob., Band 4, 1956, University of California Press, S. 149–165 (Project Euclid).
- Amenaghawon Osemwinyen, Aboubakary Diakhaby: Mathematical Modelling of the Transmission Dynamics of Ebola Virus. Juli 2015 (researchgate.net [abgerufen am 12. März 2020]).
- Benjamin Maier, Dirk Brockmann: Effective containment explains subexponential growth in recent confirmed COVID-19 cases in China. Science, 8. April 2020 (Online).
- Zum Beispiel Q. Lin u.a.: A conceptual model for the coronavirus disease 2019 (COVID-19) outbreak in Wuhan, China with individual reaction and governmental action. Int. J. Infectious Diseases, Band 93, 2020, S. 211–216.
- J.V. Noble: Geographic and temporal development of plagues. In: Nature. 250, 1974, S. 726–729. doi:10.1038/250726a0.
- Michael te Vrugt, Jens Bickmann, Raphael Wittkowski: Effects of social distancing and isolation on epidemic spreading modeled via dynamical density functional theory. In: Nature Communications. 11, 2020, S. 5576. doi:10.1038/s41467-020-19024-0.
- Matthias Kreck, Erhard Scholz: Back to the roots: A discrete Kermack-McKendrick model adapted to Covid-19. 1. April 2021 (cornell.edu [abgerufen am 10. Juli 2021]).
Anmerkungen
- Diese Form (in der nicht vorkommt) benutzen Kermack und McKendrick für ihr SIR-Modell. Auf S. 713 benutzen sie die Funktionen für sowie für Infektionsrate bzw. die Rate für die Gruppe R und schreiben
- Die in der Quelle angegebenen Differentialgleichungen sind hierbei der Vergleichbarkeit halber in dieselbe Form gebracht worden wie beim SIR-Modell oben. Es gibt in der Literatur keine einheitliche Verwendung der Parameter; neben der obigen DGL ist z. B. auch (Infektionsrate der Klarheit halber hier benannt) üblich. Es gilt dann die Identität . Bei den oben verwendeten Abbildungen ist , und die Beschreibungen auf Wikimedia Commons beziehen sich auf die letztgenannte Variante der DGL, sodass die dort genannten Infektionsraten nur der Werte laut Nomenklatur im Artikel betragen (die Bildunterschriften wurden für den Artikel angepasst).