Methionin-Salvage
Der Methionin-Salvage-Stoffwechselweg (selten: Yang-Zyklus) ist eine Abfolge von sechs chemischen Reaktionen, die das Ausgangsprodukt 5′-Methylthioadenosin (MTA) in das Endprodukt L-Methionin umwandeln. Die Enzyme, die die einzelnen Reaktionen katalysieren, können (bis auf wenige Bakterienarten) in allen Lebewesen gefunden werden. Der Prozess findet vollständig im Zytosol statt. Er ist zumindest in den auf Methionin angewiesenen Organismen lebenswichtig zur Rückgewinnung des Schwefelatoms, dessen Assimilation energieaufwändig ist. Der Stoffwechselweg wurde zunächst in Klebsiella pneumoniae und Saccharomyces cerevisiae ausgiebig untersucht. Auch in Pflanzen wurde er im Zusammenhang mit der Ethen-Biosynthese untersucht. Im Menschen sind noch nicht alle Einzelschritte völlig geklärt.[1][2][3]
| Übergeordnet |
| Methioninsynthese |
| Gene Ontology |
|---|
| QuickGO |
MTA entsteht bei der Synthese der Polyamine aus Adenosylmethionin (SAM) beziehungsweise Decarboxy-SAM durch Übertragung einer Aminopropylgruppe, Teil des Methionins, von dem noch die Methylthio-Gruppe übrig bleibt. Im Folgenden wird nach Abspaltung des Adenin schrittweise diese Aminopropylgruppe wiederhergestellt, auf Kosten des Riboserings.
5′-Methylthioribose-1-phosphat
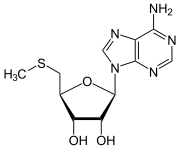 + Pi + H+
+ Pi + H+ 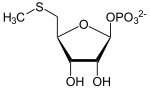 +
+ 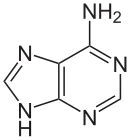
In der ersten Reaktion katalysiert das Enzym 5′-Methylthioadenosin-Phosphorylase (EC 2.4.2.28) die Abspaltung von Adenin und gleichzeitige Phosphorylierung von MTA zum 5′-Methylthioribose-1-phosphat. In manchen Bakterien und Pflanzen ist die Enzymaktivität auf zwei Enzyme, eine Methylthioadenosin-Nukleosidase (EC 3.2.2.16) und eine Methylthioribose-Kinase (EC 2.7.1.100) verteilt, wobei Letztere jedoch ein Molekül ATP verbraucht.[4]
5′-Methylthioribulose-1-phosphat

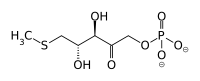
Zwischen 5′-Methylthioribose-1-phosphat und 5′-Methylthioribulose-1-phosphat herrscht ein von der 5′-Methylthioribose-1-phosphat-Isomerase (EC 5.3.1.23) katalysiertes Gleichgewicht.[5]
2,3-Diketo-5′-methylthiopentan-1-phosphat

-2%252C3-dioxopentyl_phosphate.svg.png.webp) + H2O
+ H2O
Die Dehydratation von 5′-Methylthioribulose-1-phosphat mithilfe der 5′-Methylthioribulose-1-phosphat-Dehydratase (EC 4.2.1.109) ist schwer umkehrbar. Beim Menschen wird diese Reaktion wahrscheinlich vom Produkt des APIP-Gens katalysiert, das ortholog zum gut untersuchten Mde1p-Gen der Bäckerhefe ist.[2][6]
Acireducton
-2%252C3-dioxopentyl_phosphate.svg.png.webp) + H2O
+ H2O 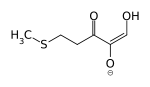 + 2H+ + Pi
+ 2H+ + Pi
Das Enzym Enolase-Phosphatase E1 (auch: Acireducton-Synthase, EC 3.1.3.77) katalysiert die Dephosphorylierung und anschließende Umwandlung zum Enol mit dem Ergebnis 1,2-Dihydroxy-3-keto-5′-methylthiopenten (Acireducton).[7]
4-Methylthio-2-ketobutanoat (MOB)
 + O2
+ O2 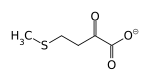 + HCOOH
+ HCOOH
 + O2
+ O2  + CO + HCOOH
+ CO + HCOOH
In einem weiteren Schritt wird Acireducton mittels der Acireducton-Dioxygenase und Disauerstoff oxidiert, wobei zwei Reaktionswege möglich sind, je nachdem ob das Enzym Eisen(II) oder Nickel(II) als Kofaktor trägt. Mit Nickel entsteht 3-Methylthiopropionat, Kohlenmonoxid und Formiat (EC 1.13.11.53) -- diese Reaktion ist bei Klebsiella nachgewiesen. Mit Eisen entsteht 4-Methylthio-2-ketobutanoat (MOB, KMTB) und Formiat (EC 1.13.11.54). Ob Nickel beim Menschen als Cofaktor eine Rolle spielt, ist ungeklärt.[8][9]
Methionin
 + R-CH(NH3+)-COO−
+ R-CH(NH3+)-COO− ![]() + R-CO-COO−
+ R-CO-COO−
Zuletzt wird MOB zu Methionin transaminiert. Transaminasen können ein breites Spektrum an Substraten haben: laut Untersuchungen in der Hefe fungieren Aro8p, Aro9p, Bat1p und Bat2p als MOB-Transaminase; im Parasiten Crithidia fasciculata ist dies die Aspartat-Transaminase, die ortholog zur menschlichen gamma-Glutamyltransferase ist. In einer Studie an der Ratte zeigte sich, dass mehrere Transaminasen im Leberzytosol für die Methioninsynthese verantwortlich sind, während für die umgekehrte Richtung, die Verarbeitung eines Überschusses Methionin, mitochondrielle Transaminasen zuständig sind. Leider gibt es keine Untersuchungen über die MOB-Transaminaseaktivität menschlicher Transaminasen.[2][10][11]
Regulation
Die Aktivität des Stoffwechselwegs ist prinzipiell abhängig vom Ausgangssubstrat MTA. Bei Methioninüberschuss kann jedoch das Gleichgewicht der Transaminierung umkippen und zu einem MOB-Überschuss führen, der in den Mitochondrien wahrscheinlich decarboxyliert und weiter abgebaut wird.[11][12]
Gewebsspezifität des Methionin-Salvage-Stoffwechselweges
Ursprünglich hatte man angenommen, dass der Methionin-Salvage-Stoffwechselweg in allen Zellen von Pflanzen abläuft. Molekularbiologische Untersuchungen in der Ackerschmalwand (Arabidopsis thaliana) und dem Großen Wegerich (Plantago major) zeigten jedoch, dass die Gene für die Enzyme, die an diesem Stoffwechselweg beteiligt sind, nur im Phloem exprimiert werden.[13]
Einzelnachweise
- Wray JW, Abeles RH: The methionine salvage pathway in Klebsiella pneumoniae and rat liver. Identification and characterization of two novel dioxygenases. In: J. Biol. Chem.. 270, Nr. 7, Februar 1995, S. 3147–53. PMID 7852397.
- Pirkov I, Norbeck J, Gustafsson L, Albers E: A complete inventory of all enzymes in the eukaryotic methionine salvage pathway. In: FEBS J.. 275, Nr. 16, August 2008, S. 4111–20. doi:10.1111/j.1742-4658.2008.06552.x. PMID 18625006.
- Miyazaki JH, Yang SF: The methionine salvage pathway in relation to ethylene and polyamine biosynthesis. In: Phys. Plant.. 69, Nr. 2, Februar 1987, S. 366–370. doi:10.1111/j.1399-3054.1987.tb04302.x.
- Kamatani N, Nelson-Rees WA, Carson DA: Selective killing of human malignant cell lines deficient in methylthioadenosine phosphorylase, a purine metabolic enzyme. In: Proc. Natl. Acad. Sci. USA. 78, Nr. 2, Februar 1981, S. 1219–23. PMID 6785752. PMC 319979 (freier Volltext).
- Kabuyama Y, Litman ES, Templeton PD, et al.: A mediator of Rho-dependent invasion moonlights as a methionine salvage enzyme. In: Mol. Cell Proteomics. 8, Nr. 10, Oktober 2009, S. 2308–20. doi:10.1074/mcp.M900178-MCP200. PMID 19620624. PMC 2758758 (freier Volltext).
- UniProt Q96GX9
- Wang H, Pang H, Bartlam M, Rao Z: Crystal structure of human E1 enzyme and its complex with a substrate analog reveals the mechanism of its phosphatase/enolase activity. In: J. Mol. Biol.. 348, Nr. 4, Mai 2005, S. 917–26. doi:10.1016/j.jmb.2005.01.072. PMID 15843022.
- Ju T, Goldsmith RB, Chai SC, Maroney MJ, Pochapsky SS, Pochapsky TC: One protein, two enzymes revisited: a structural entropy switch interconverts the two isoforms of acireductone dioxygenase. In: J. Mol. Biol.. 363, Nr. 4, November 2006, S. 823–34. doi:10.1016/j.jmb.2006.08.060. PMID 16989860. PMC 1808343 (freier Volltext).
- Oram SW, Ai J, Pagani GM, et al.: Expression and function of the human androgen-responsive gene ADI1 in prostate cancer. In: Neoplasia. 9, Nr. 8, August 2007, S. 643–51. PMID 17786183. PMC 1950434 (freier Volltext).
- Berger LC, Wilson J, Wood P, Berger BJ: Methionine regeneration and aspartate aminotransferase in parasitic protozoa. In: J. Bacteriol.. 183, Nr. 15, August 2001, S. 4421–34. doi:10.1128/JB.183.15.4421-4434.2001. PMID 11443076. PMC 95336 (freier Volltext).
- Scislowski PW, Pickard K: Methionine transamination--metabolic function and subcellular compartmentation. In: Mol. Cell. Biochem.. 129, Nr. 1, Dezember 1993, S. 39–45. PMID 8177225.
- Jones SM, Yeaman SJ: Oxidative decarboxylation of 4-methylthio-2-oxobutyrate by branched-chain 2-oxo acid dehydrogenase complex. In: Biochem. J.. 237, Nr. 2, Juli 1986, S. 621–3. PMID 3800905. PMC 1147032 (freier Volltext).
- Pommerrenig B, Feussner K, Zierer W, Rabinovych V, Klebl F, Feussner I, Sauer N: Phloem-specific expression of Yang cycle genes and identification of novel Yang cycle enzymes in Plantago and Arabidopsis. In: The Plant Cell. 23, Nr. 5, Mai 2011, S. 1904–19. PMID 21540433.